Virus de ARN
Un virus de ARN es un virus que tiene ácido ribonucleico (ARN) como material genético. El ácido nucleico suele ser ARN monocatenario (ssRNA), pero puede ser de doble cadena (dsRNA). Las enfermedades humanas notables causadas por virus de ARN incluyen el resfriado común, la influenza, el SARS, el MERS, el Covid-19, el virus del dengue, la hepatitis C, la hepatitis E, la fiebre del Nilo Occidental, la enfermedad por el virus del Ébola, la rabia, la poliomielitis, las paperas y el sarampión.
El Comité Internacional de Taxonomía de Virus (ICTV) clasifica los virus de ARN como aquellos que pertenecen al Grupo III, Grupo IV o Grupo V del sistema de clasificación de Baltimore. Esta categoría excluye el Grupo VI, virus con material genético de ARN pero que utilizan intermediarios de ADN en su ciclo de vida: estos se denominan retrovirus, incluidos el VIH-1 y el VIH-2 que causan el SIDA.
A partir de mayo de 2020, se cree que todos los virus de ARN conocidos que codifican una ARN polimerasa dirigida por ARN forman un grupo monofilético, conocido como el reino Riboviria. La mayoría de estos virus de ARN pertenecen al reino Orthornavirae y el resto tiene un posicionamiento aún no definido. El reino no contiene todos los virus de ARN: Deltavirus, Asunviroidae y Pospiviroidae son taxones de virus de ARN que se incluyeron por error en 2019, pero se corrigieron en 2020.
Características
Virus de ARN monocatenario y sentido de ARN
Los virus de ARN se pueden clasificar según el sentido o la polaridad de su ARN en virus de ARN de sentido negativo y sentido positivo o ambisentido. El ARN viral de sentido positivo es similar al ARNm y, por lo tanto, la célula huésped puede traducirlo inmediatamente. El ARN viral de sentido negativo es complementario del ARNm y, por lo tanto, debe convertirse en ARN de sentido positivo mediante una ARN polimerasa dependiente de ARN antes de la traducción. El ARN purificado de un virus de sentido positivo puede causar infección directamente, aunque puede ser menos infeccioso que la partícula de virus completa. Por el contrario, el ARN purificado de un virus de sentido negativo no es infeccioso por sí mismo, ya que debe transcribirse en ARN de sentido positivo; cada virión se puede transcribir en varios ARN de sentido positivo. Los virus de ARN ambisense se parecen a los virus de ARN de sentido negativo,
Virus de ARN de doble cadena
Los virus de ARN bicatenario (ds) representan un grupo diverso de virus que varían ampliamente en el rango de huéspedes (humanos, animales, plantas, hongos,y bacterias), el número de segmento del genoma (uno a doce) y la organización del virión (número de triangulación, capas de la cápside, picos, torretas, etc.). Los miembros de este grupo incluyen los rotavirus, que son la causa más común de gastroenteritis en niños pequeños, y los picobirnavirus, que son los virus más comunes en muestras fecales de humanos y animales con o sin signos de diarrea. El virus de la lengua azul es un patógeno económicamente importante que infecta al ganado vacuno y ovino. En los últimos años, se ha avanzado en la determinación de las estructuras de resolución atómica y subnanométrica de varias proteínas virales clave y cápsides de viriones de varios virus dsRNA, destacando los paralelos significativos en la estructura y los procesos replicativos de muchos de estos virus.
Tasas de mutación
Los virus de ARN generalmente tienen tasas de mutación muy altas en comparación con los virus de ADN, porque las polimerasas de ARN virales carecen de la capacidad de corrección de pruebas de las polimerasas de ADN. La diversidad genética de los virus de ARN es una de las razones por las que es difícil fabricar vacunas eficaces contra ellos. Los retrovirus también tienen una alta tasa de mutación a pesar de que su ADN intermedio se integra en el genoma del huésped (y, por lo tanto, está sujeto a la revisión del ADN del huésped una vez integrado), porque los errores durante la transcripción inversa se incrustan en ambas cadenas de ADN antes de la integración. Algunos genes del virus de ARN son importantes para los ciclos de replicación viral y no se toleran las mutaciones. Por ejemplo, la región del genoma del virus de la hepatitis C que codifica la proteína central está muy conservada,porque contiene una estructura de ARN involucrada en un sitio interno de entrada al ribosoma.
Replicación
Los virus de ARN animal están clasificados por el ICTV. Hay tres grupos distintos de virus de ARN según su genoma y modo de replicación:
- Los virus de ARN de doble cadena (Grupo III) contienen de una a una docena de moléculas de ARN diferentes, cada una de las cuales codifica una o más proteínas virales.
- Los virus ssRNA de sentido positivo (Grupo IV) tienen su genoma utilizado directamente como ARNm, y los ribosomas del huésped lo traducen en una sola proteína que es modificada por las proteínas virales y del huésped para formar las diversas proteínas necesarias para la replicación. Uno de estos incluye la ARN polimerasa dependiente de ARN (ARN replicasa), que copia el ARN viral para formar una forma replicativa de doble cadena. A su vez, este dsRNA dirige la formación de nuevo RNA viral.
- Los virus ssRNA de sentido negativo (Grupo V) deben tener su genoma copiado por una replicasa de ARN para formar ARN de sentido positivo. Esto significa que el virus debe traer consigo la enzima ARN replicasa. La molécula de ARN de sentido positivo luego actúa como ARNm viral, que los ribosomas del huésped traducen en proteínas.
Los retrovirus (Grupo VI) tienen un genoma de ARN monocatenario pero, en general, no se consideran virus de ARN porque utilizan intermediarios de ADN para replicarse. La transcriptasa inversa, una enzima viral que proviene del propio virus después de que se quita la cubierta, convierte el ARN viral en una cadena complementaria de ADN, que se copia para producir una molécula de ADN viral de doble cadena. Después de que este ADN se integre en el genoma del huésped utilizando la enzima viral integrasa, la expresión de los genes codificados puede conducir a la formación de nuevos viriones.
Recombinación
Numerosos virus de ARN son capaces de recombinación genética cuando al menos dos genomas virales están presentes en la misma célula huésped. Muy raramente, el ARN viral puede recombinarse con el ARN del huésped. La recombinación de ARN parece ser una fuerza impulsora importante para determinar la arquitectura del genoma y el curso de la evolución viral entre Picornaviridae ((+)ssRNA), por ejemplo, poliovirus. En los Retroviridae ((+)ssRNA), por ejemplo, el VIH, el daño en el genoma del ARN parece evitarse durante la transcripción inversa mediante el cambio de cadena, una forma de recombinación. La recombinación también ocurre en Reoviridae (dsRNA), por ejemplo, reovirus; Orthomyxoviridae ((-)ssRNA), por ejemplo, virus de la gripe; y Coronaviridae((+)ARNss), por ejemplo, SARS. La recombinación en los virus de ARN parece ser una adaptación para hacer frente al daño del genoma. La recombinación puede ocurrir con poca frecuencia entre virus animales de la misma especie pero de linajes divergentes. Los virus recombinantes resultantes a veces pueden causar un brote de infección en humanos.
Clasificación
La clasificación de los virus de ARN es difícil. Esto se debe en parte a las altas tasas de mutación que experimentan estos genomas. La clasificación se basa principalmente en el tipo de genoma (doble cadena, monocatenario negativo o positivo) y el número y organización del gen. Actualmente, hay 5 órdenes y 47 familias de virus de ARN reconocidos. También hay muchas especies y géneros no asignados.
Relacionados pero distintos de los virus de ARN están los viroides y los virus satélite de ARN. Actualmente, estos no están clasificados como virus de ARN y se describen en sus propias páginas.
Un estudio de varios miles de virus de ARN ha mostrado la presencia de al menos cinco taxones principales: un grupo de levivirus y parientes; un supergrupo de picornavirus; un supergrupo de alfavirus más un supergrupo de flavivirus; los virus dsRNA; y los virus de cadena -ve. El grupo de lentivirus parece ser basal para todos los virus de ARN restantes. La siguiente gran división se encuentra entre el picornasupragrupo y los virus restantes. Los virus dsRNA parecen haber evolucionado a partir de un ancestro de RNA +ve y los virus de RNA -ve de dentro de los virus dsRNA. El pariente más cercano a los virus de ARN de cadena -ve es el Reoviridae.
Virus de ARN de cadena positiva
Este es el grupo más grande de virus de ARN con 30 familias. Se han hecho intentos de agrupar a estas familias en órdenes superiores. Estas propuestas se basaron en un análisis de las polimerasas de ARN y todavía están bajo consideración. Hasta la fecha, las sugerencias propuestas no han sido ampliamente aceptadas debido a las dudas sobre la idoneidad de un solo gen para determinar la taxonomía del clado.
La clasificación propuesta de los virus de ARN de cadena positiva se basa en la ARN polimerasa dependiente de ARN. Se han reconocido tres grupos:
- Bymovirus, comovirus, nepovirus, nodavirus, picornavirus, potyvirus, sobemovirus y un subconjunto de luteovirus (beet western yellows virus y potato leafroll virus)—el grupo tipo picorna (Picornavirata).
- Carmovirus, dianthovirus, flavivirus, pestivirus, estatovirus, tombusvirus, bacteriófagos de ARN monocatenario, virus de la hepatitis C y un subconjunto de luteovirus (virus del enanismo amarillo de la cebada), el grupo similar a los flavi (Flavivirata).
- Alphavirus, carlavirus, furovirus, hordeivirus, potexvirus, rubivirus, tobravirus, tricornavirus, tymovirus, virus de la mancha foliar clorótica de la manzana, virus del amarillo de la remolacha y virus de la hepatitis E: el grupo alfa (Rubivirata).
Se ha propuesto una división del supergrupo similar a alfa (similar a Sindbis) sobre la base de un nuevo dominio ubicado cerca de los extremos N de las proteínas involucradas en la replicación viral. Los dos grupos propuestos son: el grupo de los 'altovirus' (alfavirus, furovirus, virus de la hepatitis E, hordeivirus, tobamovirus, tobravirus, tricornavirus y probablemente rubivirus); y el grupo 'typovirus' (virus de la mancha foliar clorótica de la manzana, carlavirus, potexvirus y tymovirus).
El supergrupo tipo alfa se puede dividir en tres clados: los virus tipo rubi, tipo tobamo y tipo tymo.
El trabajo adicional ha identificado cinco grupos de virus de ARN de cadena positiva que contienen cuatro, tres, tres, tres y un orden(es), respectivamente.Estos catorce órdenes contienen 31 familias de virus (incluidas 17 familias de virus de plantas) y 48 géneros (incluidos 30 géneros de virus de plantas). Este análisis sugiere que los alfavirus y los flavivirus se pueden separar en dos familias, Togaviridae y Flaviridae, respectivamente, pero sugiere que otras asignaciones taxonómicas, como los pestivirus, el virus de la hepatitis C, los rubivirus, el virus de la hepatitis E y los arterivirus, pueden ser incorrectas. Los coronavirus y los torovirus parecen ser familias distintas en órdenes distintos y no géneros distintos de la misma familia como se clasifica actualmente. Los luteovirus parecen ser dos familias en lugar de una, y el virus de la mancha foliar clorótica de la manzana parece no ser un closterovirus sino un nuevo género de Potexviridae.
Evolución
La evolución de los picornavirus basada en un análisis de sus ARN polimerasas y helicasas parece datar de la divergencia de los eucariotas. Sus ancestros putativos incluyen los retroelementos bacterianos del grupo II, la familia de proteasas HtrA y bacteriófagos de ADN.
Los partitivirus están relacionados y pueden haber evolucionado a partir de un ancestro totivirus.
Los hipovirus y los barnavirus parecen compartir un linaje con los linajes de potyvirus y sobemovirus, respectivamente.
Virus de ARN de doble cadena
Este análisis también sugiere que los virus dsRNA no están estrechamente relacionados entre sí, sino que pertenecen a cuatro clases adicionales (Birnaviridae, Cystoviridae, Partitiviridae y Reoviridae) y un orden adicional (Totiviridae) de una de las clases de virus ssRNA positivos en el mismo subfilo que los virus de ARN de cadena positiva.
Un estudio ha sugerido que hay dos grandes clados: uno incluye las familias Caliciviridae, Flaviviridae y Picornaviridae y un segundo que incluye las familias Alphatetraviridae, Birnaviridae, Cystoviridae, Nodaviridae y Permutotretraviridae .
Virus de ARN de cadena negativa
Estos virus tienen múltiples tipos de genoma que van desde una sola molécula de ARN hasta ocho segmentos. A pesar de su diversidad, parece que pueden haberse originado en los artrópodos y haberse diversificado a partir de ahí.
Virus satelitales
También se conocen varios virus satélite, virus que requieren la ayuda de otro virus para completar su ciclo de vida. Su taxonomía aún no se ha establecido. Se han propuesto los cuatro géneros siguientes para virus satélite de ARN monocatenario de sentido positivo que infectan plantas: Albetovirus, Aumaivirus, Papanivirus y Virtovirus. Se ha propuesto una familia, Sarthroviridae, que incluye el género Macronovirus, para los virus satélite de ARN monocatenario de sentido positivo que infectan a los artrópodos.
Grupo III: virus dsRNA
Hay doce familias y varios géneros y especies no asignados reconocidos en este grupo.
- Familia Amalgaviridae
- Familia Birnaviridae
- Familia Chrysoviridae
- Familia Cystoviridae
- Familia Endornaviridae
- Familia Hypoviridae
- Familia Megabirnaviridae
- Familia Partitiviridae
- Familia Picobirnaviridae
- Familia Reoviridae - incluye Rotavirus
- Familia Totiviridae
- Familia Quadriviridae
- Género Botybirnavirus
- Especies no asignadas
- Botrytis porri ARN virus 1
- Circulifer tenellus virus 1
- Colletotrichum camelliae virus filamentoso 1
- Virus asociado al amarillo de las cucurbitáceas
- Virus asociado al debilitamiento de Sclerotinia sclerotiorum
- Spissistilus festinus virus 1
Grupo IV: virus ssRNA de sentido positivo
Hay tres órdenes y 34 familias reconocidas en este grupo. Además, hay una serie de especies y géneros sin clasificar.
- Orden Nidovirales
- Familia Arteriviridae
- Familia Coronaviridae: incluye coronavirus humano (virus del resfriado común HCoV-229E, HCoV-HKU1, HCoV-NL63 y HCoV-OC43), MERS-CoV, SARS-CoV-1 y SARS-CoV-2
- Familia Mesoniviridae
- Familia Roniviridae
- Orden Picornavirales
- Familia Dicistroviridae
- Familia Iflaviridae
- Familia Marnaviridae
- Familia Picornaviridae: incluye poliovirus, rinovirus (un virus del resfriado común), virus de la hepatitis A
- La familia Secoviridae incluye la subfamilia Comovirinae
- Género Bacillariornavirus
- Especies Virus de la mosca Kelp
- Orden Tymovirales
- Familia Alphaflexiviridae
- Familia Betaflexiviridae
- Familia Gammaflexiviridae
- Familia Tymoviridae
- Sin asignar
- Familia Alphatetraviridae
- Familia Alvernaviridae
- Familia Astroviridae
- Familia Barnaviridae
- Familia Benyviridae
- Familia Botourmiaviridae
- Familia Bromoviridae
- Familia Caliciviridae - incluye el virus de Norwalk
- Familia Carmotetraviridae
- Familia Closteroviridae
- Familia Flaviviridae: incluye el virus de la fiebre amarilla, el virus del Nilo Occidental, el virus de la hepatitis C, el virus de la fiebre del dengue y el virus Zika.
- Familia Fusariviridae
- Familia Hepeviridae
- Familia Hypoviridae
- Familia Leviviridae
- Familia Luteoviridae: incluye el virus del enanismo amarillo de la cebada
- Familia Polycipiviridae
- Familia Narnaviridae
- Familia Nodaviridae
- Familia Permutotetraviridae
- Familia Potyviridae
- Familia Sarthroviridae
- Estatovirus familiar
- Familia Togaviridae: incluye el virus de la rubéola, el virus del río Ross, el virus Sindbis y el virus Chikungunya.
- Familia Tombusviridae
- Familia Virgaviridae
- géneros no asignados
- Género Blunervirus
- Género Cilevirus
- Género Higrevirus
- Género Idaeovirus
- Género Negevirus
- Género Ourmiavirus
- Género Polemovirus
- Género Sinaivirus
- Género Sobemovirus
- Especies no asignadas
- Virus Acyrthosiphon pisum
- bastrovirus
- Virus Blackford
- Virus de la mancha anular necrótica del arándano
- Cadicistrovirus
- virus chara australis
- Virus extrapequeño
- Virus de la clorosis de las bayas de Goji
- Virus de la armonía axyridis 1
- Hepelivirus
- Virus de la garrapata Jingmen
- virus le blanc
- nedicistrovirus
- Nesidiocoris tenuis virus 1
- Niflavirus
- Nylanderia fulva virus 1
- virus orsay
- Osedax japonicus ARN virus 1
- Picalivirus
- Nidovirus de células secretoras de planarias
- Plasmopara halstedii virus
- Rosellinia necatrix fusarivirus 1
- virus santeil
- Secalivirus
- Solenopsis invicta virus 3
- Virus del gusano redondo del cerdo grande de Wuhan
Virus satelitales
- Familia Sarthroviridae
- Género Albetovirus
- género Aumaivirus
- Género Papanivirus
- Género Virtovirus
- Virus de la parálisis crónica de las abejas
También se ha descrito un virus tipo astrovirus/hepevirus no clasificado.
Grupo V: virus ssRNA de sentido negativo
Con la excepción del virus de la hepatitis D, este grupo de virus se ha colocado en un solo filo: Negarnaviricota. Este filo se ha dividido en dos subfilos: Haploviricotina y Polyploviricotina. Dentro del subfilo Haploviricotina se reconocen actualmente cuatro clases: Chunqiuviricetes, Milneviricetes, Monjiviricetes y Yunchangviricetes. En el subfilo Polyploviricotina se reconocen dos clases: Ellioviricetes e Insthoviricetes.
Actualmente se reconocen en este grupo seis clases, siete órdenes y veinticuatro familias. Aún no se han clasificado varias especies y géneros no asignados.
- Filo Negarnaviricota
- Subfilo Haploviricotina
- Clase Chunqiuviricetes
- Orden Muvirales
- Familia Qinviridae
- Orden Muvirales
- Clase Milneviricetes
- Orden Serpentovirales
- Familia Aspiviridae
- Orden Serpentovirales
- Clase Monjiviricetes
- Orden Jingchuvirales
- Familia Chuviridae
- Orden Mononegavirales
- Familia Bornaviridae - Virus de la enfermedad de Borna
- Familia Filoviridae: incluye el virus del Ébola y el virus de Marburg
- Familia Mymonaviridae
- Familia Nyamiviridae
- Familia Paramyxoviridae: incluye el virus del sarampión, el virus de las paperas, el virus Nipah, el virus Hendra y el VEN
- Familia Pneumoviridae: incluye RSV y Metapneumovirus
- Familia Rhabdoviridae: incluye el virus de la rabia
- Familia Sunviridae
- Género Anphevirus
- género Arlivirus
- Género Chengtivirus
- Género Crustavirus
- Género Wastrivirus
- Orden Jingchuvirales
- Clase Yunchangviricetes
- Orden Goujianvirales
- Familia Yueviridae
- Orden Goujianvirales
- Clase Chunqiuviricetes
- Subfilo Polyploviricotina
- Clase Ellioviricetes
- Orden Bunyavirales
- Familia Arenaviridae - incluye el virus Lassa
- Familia Cruliviridae
- Familia Feraviridae
- Familia Fimoviridae
- Familia Hantaviridae
- Familia Jonviridae
- Familia Nairoviridae
- Familia Peribunyaviridae
- Familia Phasmaviridae
- Familia Phenuiviridae
- Familia Tospoviridae
- Género Tilapineviridae
- Orden Bunyavirales
- Clase Insthoviricetes
- Orden Articulavirales
- Familia Amnoonviridae - incluye el virus Taastrup
- Familia Orthomyxoviridae: incluye los virus de la influenza
- Orden Articulavirales
- Clase Ellioviricetes
- Subfilo Haploviricotina
- Géneros no asignados:
- Género Deltavirus: incluye el virus de la hepatitis D (no es un virus verdadero, sino un agente subviral)
Galería
Virus Lassa (Arenaviridae)
 Virus de la coriomeningitis linfocítica (Arenaviridae)
Virus de la coriomeningitis linfocítica (Arenaviridae) Hantavirus (bunyaviridae)
Hantavirus (bunyaviridae) Virus de Marburgo (Filoviridae)
Virus de Marburgo (Filoviridae) Virus del Ébola (Filoviridae)
Virus del Ébola (Filoviridae)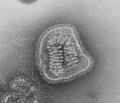 Influenza (Orthomyxoviridae)
Influenza (Orthomyxoviridae)Sarampión (Paramyxoviridae)
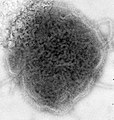 Virus de las paperas (Paramyxoviridae)
Virus de las paperas (Paramyxoviridae)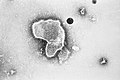 Virus respiratorio sincitial humano (Paramyxoviridae)
Virus respiratorio sincitial humano (Paramyxoviridae)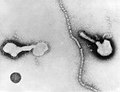 Parainfluenza (Paramyxoviridae)
Parainfluenza (Paramyxoviridae)Rabia (Rhabdoviridae)
 Virus de la estomatitis vesicular (Rhabdoviridae)
Virus de la estomatitis vesicular (Rhabdoviridae)
Contenido relacionado
Síndrome de radiación aguda
Trastornos de lectura
Toxina