Ciprínidos
(leer más)
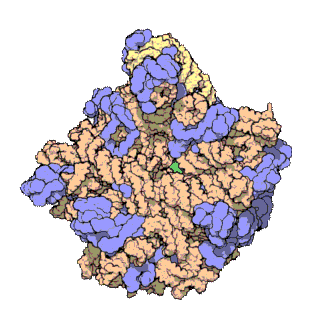
Los ribosomas, también llamados gránulos de Palade (en honor al descubridor George Palade y debido a su estructura granular), son máquinas macromoleculares, que se encuentran dentro de todas las células, que realizan proteínas biológicas síntesis (traducción de ARNm). Los ribosomas unen los aminoácidos en el orden especificado por los codones de las moléculas de ARN mensajero (ARNm) para formar cadenas polipeptídicas. Los ribosomas constan de dos componentes principales: las subunidades ribosómicas pequeña y grande. Cada subunidad consta de una o más moléculas de ARN ribosómico (ARNr) y muchas proteínas ribosómicas (RP o proteínas r).Los ribosomas y las moléculas asociadas también se conocen como el aparato de traducción.
La secuencia de ADN que codifica la secuencia de aminoácidos de una proteína se transcribe en una cadena de ARN mensajero. Los ribosomas se unen a los ARN mensajeros y usan sus secuencias para determinar la secuencia correcta de aminoácidos para generar una proteína determinada. Los aminoácidos se seleccionan y transportan al ribosoma mediante moléculas de ARN de transferencia (ARNt), que ingresan al ribosoma y se unen a la cadena de ARN mensajero a través de un bucle de tallo anticodón. Para cada triplete de codificación (codón) en el ARN mensajero, hay un ARN de transferencia único que debe tener la coincidencia exacta de anticodón y lleva el aminoácido correcto para incorporarlo a una cadena polipeptídica en crecimiento. Una vez que se produce la proteína, puede plegarse para producir una estructura tridimensional funcional.
Un ribosoma está hecho de complejos de ARN y proteínas y, por lo tanto, es un complejo de ribonucleoproteínas. Cada ribosoma se compone de componentes pequeños (30S) y grandes (50S), llamados subunidades, que están unidos entre sí:
La síntesis de proteínas a partir de sus componentes básicos tiene lugar en cuatro fases: iniciación, elongación, terminación y reciclaje. El codón de inicio en todas las moléculas de ARNm tiene la secuencia AUG. El codón de parada es uno de UAA, UAG o UGA; dado que no hay moléculas de ARNt que reconozcan estos codones, el ribosoma reconoce que la traducción está completa. Cuando un ribosoma termina de leer una molécula de ARNm, las dos subunidades se separan y generalmente se rompen, pero se pueden reutilizar. Los ribosomas son ribozimas, porque la actividad catalítica de la peptidil transferasa que une los aminoácidos la realiza el ARN ribosómico.
Los ribosomas a menudo se asocian con las membranas intracelulares que forman el retículo endoplásmico rugoso.
Los ribosomas de bacterias, arqueas y eucariotas en el sistema de tres dominios se parecen entre sí en un grado notable, evidencia de un origen común. Difieren en su tamaño, secuencia, estructura y proporción de proteína a ARN. Las diferencias en la estructura permiten que algunos antibióticos eliminen las bacterias al inhibir sus ribosomas, mientras que los ribosomas humanos no se ven afectados. En todas las especies, más de un ribosoma puede moverse a lo largo de una sola cadena de ARNm al mismo tiempo (como un polisoma), cada uno "leyendo" una secuencia específica y produciendo una molécula de proteína correspondiente.
Los ribosomas mitocondriales de las células eucariotas se asemejan funcionalmente a muchas características de los de las bacterias, lo que refleja el probable origen evolutivo de las mitocondrias.
Los ribosomas fueron observados por primera vez a mediados de la década de 1950 por el biólogo celular rumano-estadounidense George Emil Palade, utilizando un microscopio electrónico, como partículas densas o gránulos. El término "ribosoma" fue propuesto por el científico Haguenau a fines de 1958:
Durante el transcurso del simposio se hizo evidente una dificultad semántica. Para algunos de los participantes, "microsomas" significa las partículas de ribonucleoproteína de la fracción de microsomas contaminadas por otras proteínas y material lipídico; para otros, los microsomas consisten en proteínas y lípidos contaminados por partículas. La frase "partículas microsómicas" no parece adecuada, y "partículas de ribonucleoproteínas de la fracción microsómica" es demasiado incómoda. Durante la reunión se sugirió la palabra "ribosoma", que tiene un nombre muy satisfactorio y un sonido agradable. La confusión actual se eliminaría si se adoptara "ribosoma" para designar partículas de ribonucleoproteína en tamaños que oscilan entre 35 y 100S.— Albert, Partículas microsomales y síntesis de proteínas
Albert Claude, Christian de Duve y George Emil Palade recibieron conjuntamente el Premio Nobel de Fisiología o Medicina en 1974 por el descubrimiento del ribosoma. El Premio Nobel de Química 2009 fue otorgado a Venkatraman Ramakrishnan, Thomas A. Steitz y Ada E. Yonath por determinar la estructura detallada y el mecanismo del ribosoma.
El ribosoma es una máquina celular compleja. Se compone en gran parte de ARN especializado conocido como ARN ribosómico (ARNr), así como de docenas de proteínas distintas (el número exacto varía ligeramente entre especies). Las proteínas ribosómicas y los ARNr se organizan en dos piezas ribosómicas distintas de diferentes tamaños, conocidas generalmente como la subunidad grande y pequeña del ribosoma. Los ribosomas constan de dos subunidades que encajan (Figura 2) y funcionan como una sola para traducir el ARNm en una cadena polipeptídica durante la síntesis de proteínas (Figura 1). Debido a que están formados por dos subunidades de diferente tamaño, son ligeramente más largas en el eje que en el diámetro.
Los ribosomas procarióticos miden alrededor de 20 nm (200 Å) de diámetro y están compuestos por un 65 % de ARNr y un 35 % de proteínas ribosómicas. Los ribosomas eucarióticos tienen entre 25 y 30 nm (250–300 Å) de diámetro con una proporción de ARNr a proteína cercana a 1. El trabajo cristalográfico ha demostrado que no hay proteínas ribosómicas cerca del sitio de reacción para la síntesis de polipéptidos. Esto sugiere que los componentes proteicos de los ribosomas no participan directamente en la catálisis de formación de enlaces peptídicos, sino que estas proteínas actúan como un andamio que puede mejorar la capacidad del ARNr para sintetizar proteínas (ver: ribozima).
Las subunidades ribosómicas de procariotas y eucariotas son bastante similares.
La unidad de medida utilizada para describir las subunidades ribosómicas y los fragmentos de ARNr es la unidad de Svedberg, una medida de la velocidad de sedimentación en la centrifugación en lugar del tamaño. Esto explica por qué los nombres de los fragmentos no cuadran: por ejemplo, los ribosomas bacterianos 70S están formados por subunidades 50S y 30S.
Los procariotas tienen ribosomas 70S, cada uno de los cuales consta de una subunidad pequeña (30S) y una grande (50S). E. coli, por ejemplo, tiene una subunidad de ARN 16S (que consta de 1540 nucleótidos) que está unida a 21 proteínas. La subunidad grande está compuesta por una subunidad de ARN 5S (120 nucleótidos), una subunidad de ARN 23S (2900 nucleótidos) y 31 proteínas.
| ribosoma | subunidad | ARNr | proteínas r |
|---|---|---|---|
| 70S | 50S | 23S (2904 nt) | 31 |
| 5S (120 nt) | |||
| 30S | 16S (1542 nt) | 21 |
La etiqueta de afinidad para los sitios de unión de ARNt en el ribosoma de E. coli permitió la identificación de las proteínas de los sitios A y P muy probablemente asociadas con la actividad de la peptidiltransferasa; las proteínas marcadas son L27, L14, L15, L16, L2; al menos L27 está ubicado en el sitio donante, como lo muestran E. Collatz y AP Czernilofsky. Investigaciones adicionales han demostrado que las proteínas S1 y S21, en asociación con el extremo 3′ del ARN ribosómico 16S, están involucradas en el inicio de la traducción.
Los ribosomas de las arqueas comparten las mismas dimensiones generales de los de las bacterias, siendo un ribosoma 70S formado por una subunidad grande 50S, una subunidad pequeña 30S y que contiene tres cadenas de ARNr. Sin embargo, a nivel de secuencia, están mucho más cerca de las eucariotas que de las bacterianas. Cada proteína ribosómica adicional que las arqueas tienen en comparación con las bacterias tiene una contraparte eucariota, mientras que tal relación no se aplica entre las arqueas y las bacterias.
Los eucariotas tienen ribosomas 80S ubicados en su citosol, cada uno de los cuales consta de una subunidad pequeña (40S) y una grande (60S). Su subunidad 40S tiene un ARN 18S (1900 nucleótidos) y 33 proteínas. La subunidad grande está compuesta por subunidades de ARN 5S (120 nucleótidos), ARN 28S (4700 nucleótidos), ARN 5.8S (160 nucleótidos) y 46 proteínas.
| ribosoma | subunidad | ARNr | proteínas r |
|---|---|---|---|
| años 80 | 60S | 28S (4718 nt) | 49 |
| 5.8S (160 nt) | |||
| 5S (120 nt) | |||
| 40S | 18S (1874 nt) | 33 |
Durante 1977, Czernilofsky publicó una investigación que utilizaba el marcado por afinidad para identificar sitios de unión a ARNt en ribosomas de hígado de rata. Varias proteínas, incluidas L32/33, L36, L21, L23, L28/29 y L13, estaban implicadas en el centro de la peptidil transferasa o cerca del mismo.
En los eucariotas, los ribosomas están presentes en las mitocondrias (a veces llamados mitoribosomas) y en plástidos como los cloroplastos (también llamados plastoribosomas). También consisten en subunidades grandes y pequeñas unidas con proteínas en una partícula 70S. Estos ribosomas son similares a los de las bacterias y se cree que estos orgánulos se originaron como bacterias simbióticas. De los dos, los ribosomas cloroplásticos están más cerca de los bacterianos que de los mitocondriales. Muchas piezas de ARN ribosomal en las mitocondrias se acortan y, en el caso del ARNr 5S, se reemplazan por otras estructuras en animales y hongos. En particular, Leishmania tarentolae tiene un conjunto mínimo de ARNr mitocondrial.Por el contrario, los mitoribosomas de las plantas tienen ARNr extendido y proteínas adicionales en comparación con las bacterias, en particular, muchas proteínas repetidas de pentatricopéptidos.
Las algas cryptomonad y chlorarachniophyte pueden contener un nucleomorfo que se asemeja a un núcleo eucariótico vestigial. Los ribosomas eucariotas 80S pueden estar presentes en el compartimento que contiene el nucleomorfo.
Los químicos farmacéuticos explotan las diferencias entre los ribosomas bacterianos y eucarióticos para crear antibióticos que pueden destruir una infección bacteriana sin dañar las células de la persona infectada. Debido a las diferencias en sus estructuras, los ribosomas bacterianos 70S son vulnerables a estos antibióticos, mientras que los ribosomas eucariotas 80S no lo son. Aunque las mitocondrias poseen ribosomas similares a los bacterianos, estos antibióticos no afectan a las mitocondrias porque están rodeadas por una doble membrana que no admite fácilmente estos antibióticos en el orgánulo. Sin embargo, un contraejemplo digno de mención incluye el antibiótico antineoplásico cloranfenicol, que inhibe con éxito los ribosomas 50S bacterianos y mitocondriales 50S eucariotas.No se puede decir lo mismo de las mitocondrias de los cloroplastos, donde la resistencia a los antibióticos en las proteínas ribosómicas es un rasgo a introducir como marcador en la ingeniería genética.
Los diversos ribosomas comparten una estructura central, que es bastante similar a pesar de las grandes diferencias de tamaño. Gran parte del ARN está altamente organizado en varios motivos estructurales terciarios, por ejemplo, pseudonudos que exhiben apilamiento coaxial. El ARN adicional en los ribosomas más grandes se encuentra en varias inserciones largas y continuas, de modo que forman bucles fuera de la estructura central sin alterarla ni cambiarla. Toda la actividad catalítica del ribosoma la lleva a cabo el ARN; las proteínas residen en la superficie y parecen estabilizar la estructura.
La estructura molecular general del ribosoma se conoce desde principios de la década de 1970. A principios de la década de 2000, la estructura se logró a altas resoluciones, del orden de unos pocos ångströms.
Los primeros artículos que dan la estructura del ribosoma en resolución atómica se publicaron casi simultáneamente a finales de 2000. La subunidad 50S (procariota grande) se determinó a partir del archaeon Haloarcula marismortui y la bacteria Deinococcus radiodurans, y la estructura de la subunidad 30S se determinó a partir de Termus thermophilus. Estos estudios estructurales fueron galardonados con el Premio Nobel de Química en 2009. En mayo de 2001, estas coordenadas se utilizaron para reconstruir toda la partícula T. thermophilus 70S con una resolución de 5,5 Å.
En noviembre de 2005 se publicaron dos artículos con estructuras del ribosoma 70S de Escherichia coli. Las estructuras de un ribosoma vacante se determinaron a una resolución de 3,5 Å utilizando cristalografía de rayos X. Luego, dos semanas más tarde, se publicó una estructura basada en microscopía crioelectrónica, que representa el ribosoma con una resolución de 11–15 Å en el acto de pasar una hebra de proteína recién sintetizada al canal conductor de proteínas.
Las primeras estructuras atómicas del ribosoma complejadas con moléculas de ARNt y ARNm se resolvieron utilizando cristalografía de rayos X por dos grupos de forma independiente, a 2,8 Å ya 3,7 Å. Estas estructuras permiten ver los detalles de las interacciones del ribosoma de Thermus thermophilus con el ARNm y con los ARNt unidos a sitios ribosómicos clásicos. Las interacciones del ribosoma con ARNm largos que contenían secuencias de Shine-Dalgarno se visualizaron poco después con una resolución de 4,5–5,5 Å.
En 2011, se obtuvo mediante cristalografía la primera estructura atómica completa del ribosoma eucariota 80S de la levadura Saccharomyces cerevisiae. El modelo revela la arquitectura de elementos específicos de eucariotas y su interacción con el núcleo universalmente conservado. Al mismo tiempo, se publicó el modelo completo de una estructura ribosómica 40S eucariótica en Tetrahymena thermophila y se describió la estructura de la subunidad 40S, así como mucho sobre la interacción de la subunidad 40S con eIF1 durante el inicio de la traducción. De manera similar, la estructura de la subunidad eucariota 60S también se determinó a partir de Tetrahymena thermophila en complejo con eIF6.
Los ribosomas son partículas diminutas que consisten en ARN y proteínas asociadas que funcionan para sintetizar proteínas. Las proteínas son necesarias para muchas funciones celulares, como reparar daños o dirigir procesos químicos. Los ribosomas se pueden encontrar flotando dentro del citoplasma o unidos al retículo endoplásmico. Su función principal es convertir el código genético en una secuencia de aminoácidos y construir polímeros de proteínas a partir de monómeros de aminoácidos.
Los ribosomas actúan como catalizadores en dos procesos biológicos extremadamente importantes llamados transferencia de péptidos e hidrólisis de péptidos. El "centro PT es responsable de producir enlaces de proteínas durante el alargamiento de proteínas".
En resumen, los ribosomas tienen dos funciones principales: decodificar el mensaje y formar enlaces peptídicos. Estas dos funciones residen en las subunidades ribosómicas. Cada subunidad está formada por uno o más ARNr y muchas proteínas r. La subunidad pequeña (30S en bacterias y arqueas, 40S en eucariotas) tiene la función de decodificación, mientras que la subunidad grande (50S en bacterias y arqueas, 60S en eucariotas) cataliza la formación de enlaces peptídicos, lo que se conoce como actividad peptidil-transferasa. La subunidad pequeña bacteriana (y archaeal) contiene el rRNA 16S y 21 r-proteínas (Escherichia coli), mientras que la subunidad pequeña eucariótica contiene el rRNA 18S y 32 r-proteínas (Saccharomyces cerevisiae; aunque los números varían entre especies). La subunidad grande bacteriana contiene los ARNr 5S y 23S y 34 proteínas r (E. coli),
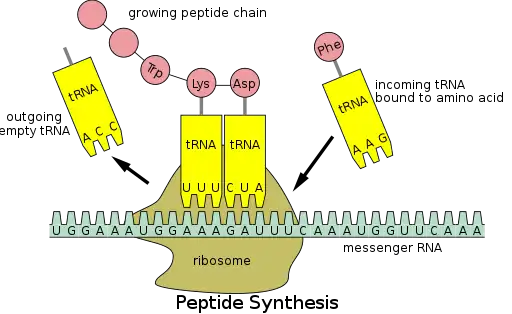
Los ribosomas son los lugares de trabajo de la biosíntesis de proteínas, el proceso de traducir el ARNm en proteína. El ARNm comprende una serie de codones que son decodificados por el ribosoma para producir la proteína. Usando el mRNA como plantilla, el ribosoma atraviesa cada codón (3 nucleótidos) del mRNA, emparejándolo con el aminoácido apropiado provisto por un aminoacil-tRNA. El aminoacil-tRNA contiene un anticodón complementario en un extremo y el aminoácido apropiado en el otro. Para un reconocimiento rápido y preciso del ARNt apropiado, el ribosoma utiliza grandes cambios conformacionales (corrección de pruebas conformacionales).La subunidad ribosómica pequeña, típicamente unida a un aminoacil-tRNA que contiene el primer aminoácido metionina, se une a un codón AUG en el mRNA y recluta la subunidad ribosómica grande. El ribosoma contiene tres sitios de unión de ARN, denominados A, P y E. El sitio A se une a un aminoacil-tRNA o factores de liberación de terminación; el sitio P se une a un peptidil-tRNA (un tRNA unido a la cadena polipeptídica); y el sitio E (salida) se une a un tRNA libre. La síntesis de proteínas comienza en un codón de inicio AUG cerca del extremo 5' del ARNm. El ARNm se une primero al sitio P del ribosoma. El ribosoma reconoce el codón de inicio utilizando la secuencia Shine-Dalgarno del ARNm en procariotas y la caja de Kozak en eucariotas.
Aunque la catálisis del enlace peptídico involucra el hidroxilo C2 de la adenosina del sitio P del ARN en un mecanismo de transporte de protones, otros pasos en la síntesis de proteínas (como la translocación) son causados por cambios en las conformaciones de las proteínas. Dado que su núcleo catalítico está hecho de ARN, los ribosomas se clasifican como "ribozimas" y se cree que podrían ser restos del mundo del ARN.

Figura 5: Traducción de ARNm (1) por un ribosoma (2) (mostrado como subunidades
pequeñas y
grandes ) en una
cadena polipeptídica (3). El ribosoma comienza en el codón de inicio del ARN (
AUG ) y termina en el codón de terminación (
UAG ).
En la Figura 5, ambas subunidades ribosómicas ( pequeña y grande ) se ensamblan en el codón de inicio (hacia el extremo 5' del ARNm). El ribosoma usa ARNt que coincide con el codón actual (triplete) en el ARNm para agregar un aminoácido a la cadena polipeptídica. Esto se hace para cada triplete en el ARNm, mientras que el ribosoma se mueve hacia el extremo 3' del ARNm. Por lo general, en las células bacterianas, varios ribosomas trabajan en paralelo en un solo ARNm, formando lo que se denomina un polirribosoma o polisoma.
Se sabe que el ribosoma participa activamente en el plegamiento de proteínas. Las estructuras obtenidas de esta manera suelen ser idénticas a las obtenidas durante el replegamiento químico de proteínas; sin embargo, las vías que conducen al producto final pueden ser diferentes. En algunos casos, el ribosoma es crucial para obtener la forma de proteína funcional. Por ejemplo, uno de los posibles mecanismos de plegamiento de las proteínas profundamente anudadas se basa en que el ribosoma empuja la cadena a través del bucle adjunto.
La presencia de una proteína de control de calidad del ribosoma Rqc2 está asociada con el alargamiento de la proteína independiente del ARNm. Esta elongación es el resultado de la adición ribosómica (a través de ARNt traídos por Rqc2) de colas CAT : los ribosomas extienden el extremo C de una proteína estancada con secuencias aleatorias independientes de la traducción de a laninas y treoninas.
Los ribosomas se clasifican como "libres" o "unidos a la membrana".
Los ribosomas libres y unidos a la membrana difieren solo en su distribución espacial; son idénticos en estructura. El hecho de que el ribosoma exista en un estado libre o unido a la membrana depende de la presencia de una secuencia de señal dirigida al ER en la proteína que se sintetiza, por lo que un ribosoma individual podría estar unido a la membrana cuando está produciendo una proteína, pero libre en el citosol. cuando produce otra proteína.
Los ribosomas a veces se denominan orgánulos, pero el uso del término orgánulo a menudo se restringe a la descripción de componentes subcelulares que incluyen una membrana de fosfolípidos, que los ribosomas, al ser completamente particulados, no tienen. Por esta razón, los ribosomas a veces pueden describirse como "orgánulos no membranosos".
Los ribosomas libres pueden moverse por cualquier parte del citosol, pero están excluidos del núcleo celular y otros orgánulos. Las proteínas que se forman a partir de los ribosomas libres se liberan en el citosol y se utilizan dentro de la célula. Dado que el citosol contiene altas concentraciones de glutatión y, por lo tanto, es un entorno reductor, las proteínas que contienen enlaces disulfuro, que se forman a partir de residuos de cisteína oxidados, no se pueden producir en él.
Cuando un ribosoma comienza a sintetizar proteínas que se necesitan en algunos orgánulos, el ribosoma que produce esta proteína puede "unirse a la membrana". En las células eucariotas, esto sucede en una región del retículo endoplásmico (ER) llamada "ER rugosa". Las cadenas polipeptídicas recién producidas se insertan directamente en el RE mediante la síntesis vectorial del ribosoma y luego se transportan a sus destinos a través de la vía secretora. Los ribosomas unidos normalmente producen proteínas que se utilizan dentro de la membrana plasmática o se expulsan de la célula por exocitosis.
En las células bacterianas, los ribosomas se sintetizan en el citoplasma a través de la transcripción de múltiples operones de genes de ribosomas. En eucariotas, el proceso tiene lugar tanto en el citoplasma celular como en el nucléolo, que es una región dentro del núcleo celular. El proceso de ensamblaje implica la función coordinada de más de 200 proteínas en la síntesis y procesamiento de los cuatro rRNA, así como el ensamblaje de esos rRNA con las proteínas ribosómicas.
El ribosoma puede haberse originado por primera vez en un mundo de ARN, apareciendo como un complejo autorreplicante que solo más tarde desarrolló la capacidad de sintetizar proteínas cuando comenzaron a aparecer los aminoácidos. Los estudios sugieren que los ribosomas antiguos construidos únicamente con ARNr podrían haber desarrollado la capacidad de sintetizar enlaces peptídicos. Además, la evidencia apunta fuertemente a los ribosomas antiguos como complejos autorreplicantes, donde el rRNA en los ribosomas tenía propósitos informativos, estructurales y catalíticos porque podría haber codificado los tRNA y las proteínas necesarias para la autorreplicación ribosómica. Los organismos celulares hipotéticos con ARN autorreplicante pero sin ADN se denominan ribocitos (o ribocélulas).
A medida que los aminoácidos aparecieron gradualmente en el mundo del ARN en condiciones prebióticas, sus interacciones con el ARN catalítico aumentarían tanto el rango como la eficiencia de la función de las moléculas de ARN catalítico. Por lo tanto, la fuerza impulsora para la evolución del ribosoma de una antigua máquina autorreplicante a su forma actual como máquina de traducción puede haber sido la presión selectiva para incorporar proteínas en los mecanismos autorreplicantes del ribosoma, a fin de aumentar su capacidad para autorreplicación.
Los ribosomas tienen una composición heterogénea entre especies e incluso dentro de la misma célula, como lo demuestra la existencia de ribosomas citoplásmicos y mitocondriales dentro de las mismas células eucariotas. Ciertos investigadores han sugerido que la heterogeneidad en la composición de las proteínas ribosómicas en los mamíferos es importante para la regulación génica, es decir, la hipótesis del ribosoma especializado. Sin embargo, esta hipótesis es controvertida y el tema de la investigación en curso.
Vince Mauro y Gerald Edelman propusieron por primera vez que la heterogeneidad en la composición de los ribosomas estaba involucrada en el control de la traducción de la síntesis de proteínas. Propusieron la hipótesis del filtro de ribosomas para explicar las funciones reguladoras de los ribosomas. La evidencia ha sugerido que los ribosomas especializados específicos para diferentes poblaciones de células pueden afectar la forma en que se traducen los genes. Algunas proteínas ribosómicas se intercambian del complejo ensamblado con copias citosólicas, lo que sugiere que la estructura del ribosoma in vivo puede modificarse sin sintetizar un ribosoma completamente nuevo.
Ciertas proteínas ribosómicas son absolutamente críticas para la vida celular, mientras que otras no lo son. En la levadura en ciernes, 14/78 proteínas ribosómicas no son esenciales para el crecimiento, mientras que en humanos esto depende de la célula de estudio. Otras formas de heterogeneidad incluyen modificaciones postraduccionales de las proteínas ribosómicas, como la acetilación, la metilación y la fosforilación. Arabidopsis, los sitios de entrada de ribosomas internos virales (IRES) pueden mediar las traducciones por ribosomas de composición distinta. Por ejemplo, las unidades ribosómicas 40S sin eS25 en levaduras y células de mamíferos no pueden reclutar CrPV IGR IRES.
La heterogeneidad de las modificaciones del ARN ribosómico juega un papel importante en el mantenimiento y/o función estructural y la mayoría de las modificaciones del ARNm se encuentran en regiones altamente conservadas. Las modificaciones más comunes del ARNr son la seudouridilación y la metilación 2'-O de la ribosa.
(leer más)
(leer más)
(leer más)