Peptidoglucano
El peptidoglicano o mureína es un polímero que consta de azúcares y aminoácidos que forma una capa de peptidoglicano similar a una malla fuera de la membrana plasmática de la mayoría de las bacterias, formando la pared celular. El componente de azúcar consta de residuos alternantes de N -acetilglucosamina (NAG) y ácido N -acetilmurámico (NAM) unidos por β-(1,4). Unido al ácido N -acetilmurámico hay una cadena peptídica de tres a cinco aminoácidos. La cadena peptídica se puede entrecruzar con la cadena peptídica de otra cadena formando la capa similar a una malla 3D.El peptidoglucano cumple una función estructural en la pared celular bacteriana, brindando resistencia estructural y contrarrestando la presión osmótica del citoplasma. El peptidoglicano también está involucrado en la fisión binaria durante la reproducción de células bacterianas.
Esta unión repetitiva da como resultado una densa capa de peptidoglicano. Esta capa de peptidoglicano es crítica para mantener la forma celular y soportar altas presiones osmóticas, y es reemplazada regularmente por la producción de peptidoglicano. La hidrólisis y la síntesis de peptidoglicano son dos procesos que deben ocurrir para que las células crezcan y se multipliquen. Esta técnica se lleva a cabo en tres etapas: recorte del material actual, inserción de material nuevo y recruzamiento del material existente con material nuevo.
La capa de peptidoglicano es sustancialmente más gruesa en las bacterias Gram-positivas (20 a 80 nanómetros) que en las bacterias Gram-negativas (7 a 8 nanómetros). Dependiendo de las condiciones de crecimiento del pH, el peptidoglicano forma alrededor del 40 al 90% del peso seco de la pared celular de las bacterias Gram-positivas, pero solo alrededor del 10% de las cepas Gram-negativas. Por lo tanto, la presencia de altos niveles de peptidoglicano es el principal determinante de la caracterización de bacterias como Gram-positivas. En las cepas Gram-positivas, es importante en funciones de unión y con fines de serotipificación. Tanto para las bacterias Gram-positivas como para las Gram-negativas, las partículas de aproximadamente 2 nm pueden atravesar el peptidoglucano.
Es difícil saber si un organismo es grampositivo o gramnegativo usando un microscopio; Se requiere la tinción de Gram, creada por Hans Christian Gram en 1884. Las bacterias se tiñen con varios tintes, como cristal violeta, alcohol de yodo y safranina, utilizando el procedimiento de tinción de Gram. Las células Gram positivas son de color púrpura después de la tinción, mientras que las células Gram negativas son rojas.
Schleifer y Kandler han descrito los tipos de peptidoglicanos de las paredes celulares bacterianas y sus implicaciones taxonómicas. Archaea no contiene peptidoglicano (mureína). Algunas Archea contienen pseudopeptidoglicano (pseudoureína).
Estructura
La capa de peptidoglicano en la pared celular bacteriana es una estructura de red cristalina formada por cadenas lineales de dos aminoazúcares alternos, a saber, N -acetilglucosamina (GlcNAc o NAGA) y ácido N -acetilmurámico (MurNAc o NAMA). Los azúcares alternos están conectados por un enlace β-(1,4)-glucosídico. Cada MurNAc está unido a una cadena de aminoácidos corta (de 4 a 5 residuos), que contiene L -alanina, ácido D -glutámico, ácido meso -diaminopimélico y D -alanina en el caso de Escherichia coli (una bacteria Gram-negativa) o L -alanina, D -glutamina, L -lisina y D-alanina con un puente de 5-glicina entre tetrapéptidos en el caso de Staphylococcus aureus (una bacteria Gram-positiva). El peptidoglicano es una de las fuentes más importantes de D-aminoácidos en la naturaleza.
Al encerrar la membrana interna, la capa de peptididoglicano protege a la célula de la lisis causada por la presión de turgencia de la célula. Cuando la pared celular crece, conserva su forma a lo largo de su vida, por lo que una forma de barra seguirá siendo una forma de barra y una forma esférica seguirá siendo una forma esférica de por vida. Esto sucede porque el material septal de síntesis recién agregado se transforma en una llamada hemisférica para las células descendientes.
El entrecruzamiento entre aminoácidos en diferentes cadenas lineales de aminoazúcar ocurre con la ayuda de la enzima DD-transpeptidasa y da como resultado una estructura tridimensional que es fuerte y rígida. La secuencia específica de aminoácidos y la estructura molecular varían según la especie bacteriana.
 La estructura del peptidoglicano. NAG = N -acetilglucosamina (también llamado GlcNAc o NAGA), NAM = ácido N -acetilmurámico (también llamado MurNAc o NAMA).
La estructura del peptidoglicano. NAG = N -acetilglucosamina (también llamado GlcNAc o NAGA), NAM = ácido N -acetilmurámico (también llamado MurNAc o NAMA). Pared celular Gram-positiva
Pared celular Gram-positiva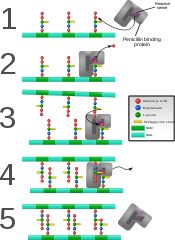 Proteína de unión a penicilina que forma enlaces cruzados en la pared celular bacteriana recién formada.
Proteína de unión a penicilina que forma enlaces cruzados en la pared celular bacteriana recién formada.
Biosíntesis
Los monómeros de peptidoglicano se sintetizan en el citosol y luego se unen a un bactoprenol transportador de membrana. Bactoprenol transporta monómeros de peptidoglicano a través de la membrana celular donde se insertan en el peptidoglicano existente.
En el primer paso de la síntesis de peptidoglicanos, la glutamina, que es un aminoácido, dona un grupo amino a un azúcar, la fructosa 6-fosfato. Esto convierte la fructosa 6-fosfato en glucosamina-6-fosfato. En el paso dos, se transfiere un grupo acetilo de acetil CoA al grupo amino en la glucosamina-6-fosfato creando N -acetil-glucosamina-6-fosfato. En el paso tres del proceso de síntesis, se isomeriza el N -acetil-glucosamina-6-fosfato, que cambiará el N -acetil-glucosamina-6-fosfato a N -acetil-glucosamina-1-fosfato.
En el paso 4, el N -acetil-glucosamina-1-fosfato, que ahora es un monofosfato, ataca la UTP. El trifosfato de uridina, que es un nucleótido de pirimidina, tiene la capacidad de actuar como fuente de energía. En esta reacción particular, después de que el monofosfato ha atacado a la UTP, se desprende un pirofosfato inorgánico que es reemplazado por el monofosfato, creando UDP-N-acetilglucosamina (2,4). (Cuando se usa UDP como fuente de energía, emite un fosfato inorgánico). Esta etapa inicial se usa para crear el precursor de la NAG en el peptidoglicano.
En el paso 5, parte de la UDP-N-acetilglucosamina (UDP-GlcNAc) se convierte en UDP-MurNAc (ácido UDP-N-acetilmurámico) mediante la adición de un grupo lactilo a la glucosamina. También en esta reacción, el grupo hidroxilo C3 eliminará un fosfato del carbono alfa del fosfoenolpiruvato. Esto crea lo que se llama un derivado de enol que se reducirá a un "resto lactilo" por NADPH en el paso seis.
En el paso 7, el UDP-MurNAc se convierte en pentapéptido UDP-MurNAc mediante la adición de cinco aminoácidos, que generalmente incluyen el dipéptido D -alanil- D - alanina. Cada una de estas reacciones requiere la fuente de energía ATP. Todo esto se conoce como Etapa uno.
La segunda etapa ocurre en la membrana citoplasmática. Es en la membrana donde un transportador de lípidos llamado bactoprenol transporta los precursores de peptidoglicanos a través de la membrana celular. Bactoprenol atacará el UDP-MurNAc penta, creando un PP-MurNac penta, que ahora es un lípido. Luego, UDP-GlcNAc se transporta a MurNAc, creando Lipid-PP-MurNAc penta-GlcNAc, un disacárido, también precursor del peptidoglicano. Aún no se comprende cómo se transporta esta molécula a través de la membrana. Sin embargo, una vez que está allí, se agrega a la creciente cadena de glicanos. La siguiente reacción se conoce como transglicosilación. En la reacción, el grupo hidroxilo de GlcNAc se unirá a MurNAc en el glucano, lo que desplazará al lípido-PP de la cadena de glucano. La enzima responsable de esto es la transglicosilasa.
Inhibición
Algunos medicamentos antibacterianos, como la penicilina, interfieren con la producción de peptidoglicano al unirse a enzimas bacterianas conocidas como proteínas de unión a penicilina o DD-transpeptidasas. Las proteínas de unión a penicilina forman los enlaces entre los enlaces cruzados de oligopéptidos en el peptidoglicano. Para que una célula bacteriana se reproduzca a través de la fisión binaria, se deben unir más de un millón de subunidades de peptidoglicano (NAM-NAG+oligopéptido) a las subunidades existentes. Las mutaciones en genes que codifican transpeptidasas que conducen a interacciones reducidas con un antibiótico son una fuente importante de resistencia antibiótica emergente. Las muraymicinas son una subclase de antibióticos nucleósidos que actúan como inhibidores competitivos del sustrato natural UDP-MurNAc-pentapéptido (UM5A) de la fosfo-N-acetilmuramoil-pentapéptido translocasa (MraY).
La lisozima, que se encuentra en las lágrimas y constituye parte del sistema inmunológico innato del cuerpo, ejerce su efecto antibacteriano al romper los enlaces glucosídicos β-(1,4) en el peptidoglucano (ver arriba).
Pseudopeptidoglicano (pseudoureína)
En algunas arqueas, es decir, miembros de Methanobacteriales y del género Methanopyrus, se ha encontrado pseudopeptidoglicano (pseudoureína). En el pseudopeptidoglucano, los residuos de azúcar son N -acetilglucosamina y ácido N -acetiltalosaminurónico unidos por β-(1,3). Esto hace que las paredes celulares de dichas arqueas sean insensibles a la lisozima. Se ha descrito la biosíntesis de pseudopeptidoglicano.
Contenido relacionado
Arginina
Dímeros de pirimidina
Lámina nuclear