Nucleosoma
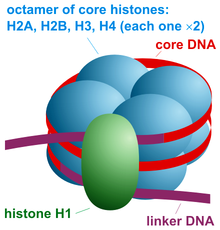
Un nucleosoma es la unidad estructural básica del empaquetamiento del ADN en eucariotas. La estructura de un nucleosoma consta de un segmento de ADN enrollado alrededor de ocho proteínas histonas y se asemeja a un hilo enrollado alrededor de un carrete. El nucleosoma es la subunidad fundamental de la cromatina. Cada nucleosoma está compuesto por un poco menos de dos vueltas de ADN envuelto alrededor de un conjunto de ocho proteínas llamadas histonas, que se conocen como octámero de histonas. Cada octámero de histona se compone de dos copias de cada una de las proteínas de histona H2A, H2B, H3 y H4.
El ADN debe compactarse en nucleosomas para encajar dentro del núcleo celular. Además del envoltorio del nucleosoma, la cromatina eucariótica se compacta aún más al plegarse en una serie de estructuras más complejas, formando eventualmente un cromosoma. Cada célula humana contiene alrededor de 30 millones de nucleosomas.
Se cree que los nucleosomas transportan información heredada epigenéticamente en forma de modificaciones covalentes de sus histonas centrales. Las posiciones de los nucleosomas en el genoma no son aleatorias y es importante saber dónde se encuentra cada nucleosoma porque esto determina la accesibilidad del ADN a las proteínas reguladoras.
Los nucleosomas fueron observados por primera vez como partículas en el microscopio electrónico por Don y Ada Olins en 1974, y Roger Kornberg propuso su existencia y estructura (como octámeros de histonas rodeados por aproximadamente 200 pares de bases de ADN). El papel del nucleosoma como regulador de la transcripción fue demostrado por Lorch et al. in vitro en 1987 y por Han y Grunstein y Clark-Adams et al. en vivo en 1988.
La partícula central del nucleosoma consta de aproximadamente 146 pares de bases (pb) de ADN envuelto en 1,67 vueltas superhelicoidales zurdas alrededor de un octámero de histonas, que consta de 2 copias de cada una de las histonas centrales H2A, H2B, H3 y H4. Las partículas del núcleo están conectadas por tramos de ADN enlazador, que pueden tener una longitud de hasta 80 pb. Técnicamente, un nucleosoma se define como la partícula central más una de estas regiones conectoras; sin embargo, la palabra es a menudo sinónimo de partícula central. Los mapas de posicionamiento de nucleosomas de todo el genoma ahora están disponibles para muchos organismos modelo, incluidos el hígado y el cerebro de ratón.
Las histonas enlazadoras, como H1 y sus isoformas, participan en la compactación de la cromatina y se ubican en la base del nucleosoma cerca de la entrada y salida del ADN y se unen a la región enlazadora del ADN. Los nucleosomas no condensados sin la histona enlazadora se asemejan a "cuentas en una cadena de ADN" bajo un microscopio electrónico.
A diferencia de la mayoría de las células eucariotas, los espermatozoides maduros utilizan en gran medida protaminas para empaquetar su ADN genómico, lo que es más probable que logre una proporción de empaquetamiento aún mayor. También se han encontrado equivalentes de histonas y una estructura de cromatina simplificada en Archaea, lo que sugiere que los eucariotas no son los únicos organismos que utilizan nucleosomas.
Estructura
Estructura de la partícula central
Resumen
Estudios estructurales pioneros realizados en la década de 1980 por el grupo de Aaron Klug proporcionaron la primera evidencia de que un octámero de proteínas histonas envuelve el ADN alrededor de sí mismo en aproximadamente 1,7 vueltas de una superhélice levógira. En 1997, el grupo de Richmond resolvió la primera estructura cristalina de resolución casi atómica del nucleosoma, mostrando los detalles más importantes de la partícula. El ADN palindrómico del satélite alfa humano fundamental para lograr la estructura cristalina del nucleosoma de 1997 fue desarrollado por el grupo Bunick en el Laboratorio Nacional Oak Ridge en Tennessee. Hasta la fecha, se han resuelto las estructuras de más de 20 partículas de núcleo de nucleosoma diferentes, incluidas aquellas que contienen variantes de histonas e histonas de diferentes especies. La estructura de la partícula del núcleo del nucleosoma está notablemente conservada, e incluso un cambio de más de 100 residuos entre histonas de rana y levadura da como resultado mapas de densidad de electrones con una desviación cuadrática media general de solo 1,6 Å.
La partícula central del nucleosoma (NCP)
La partícula central del nucleosoma (que se muestra en la figura) consta de aproximadamente 146 pares de bases de ADN envueltos en 1,67 vueltas superhelicoidales zurdas alrededor del octámero de histonas, que consta de 2 copias de cada una de las histonas centrales H2A, H2B, H3 y H4. Los nucleosomas adyacentes están unidos por un tramo de ADN libre denominado ADN enlazador (que varía de 10 a 80 pb de longitud según la especie y el tipo de tejido). Toda la estructura genera un cilindro de 11 nm de diámetro y 5,5 nm de altura.
Se observan partículas del núcleo del nucleosoma cuando se trata la cromatina en interfase para hacer que la cromatina se despliegue parcialmente. La imagen resultante, a través de un microscopio electrónico, es "cuentas en una cuerda". La cadena es el ADN, mientras que cada cuenta en el nucleosoma es una partícula central. La partícula central del nucleosoma está compuesta de ADN y proteínas histonas.
La digestión parcial con ADNasa de la cromatina revela su estructura de nucleosoma. Debido a que las porciones de ADN de las partículas del núcleo del nucleosoma son menos accesibles para la ADNasa que las secciones de unión, el ADN se digiere en fragmentos de longitudes iguales a la multiplicidad de distancia entre los nucleosomas (180, 360, 540 pares de bases, etc.). Por lo tanto, un patrón muy característico similar a una escalera es visible durante la electroforesis en gel de ese ADN. Dicha digestión también puede ocurrir en condiciones naturales durante la apoptosis ('suicidio celular' o muerte celular programada), porque su función suele ser la autodestrucción del ADN.
Interacciones de proteínas dentro del nucleosoma
Las proteínas histonas centrales contienen un motivo estructural característico denominado "pliegue de histonas", que consta de tres hélices alfa (α1-3) separadas por dos bucles (L1-2). En solución, las histonas forman heterodímeros H2A-H2B y heterotetrámeros H3-H4. Las histonas se dimerizan alrededor de sus largas hélices α2 en una orientación antiparalela y, en el caso de H3 y H4, dos de estos dímeros forman un haz de 4 hélices estabilizado por H3-H3' interacción. El dímero H2A/H2B se une al tetrámero H3/H4 debido a las interacciones entre H4 y H2B, que incluyen la formación de un grupo hidrofóbico. El octámero de histonas está formado por un tetrámero central H3/H4 intercalado entre dos dímeros H2A/H2B. Debido a la carga altamente básica de las cuatro histonas centrales, el octámero de histonas es estable solo en presencia de ADN o concentraciones muy altas de sal.
Histona - interacciones ADN
El nucleosoma contiene más de 120 interacciones proteína-ADN directas y varios cientos de interacciones mediadas por agua. Las interacciones directas entre proteína y ADN no se distribuyen uniformemente en la superficie del octámero, sino que se ubican en sitios discretos. Estos se deben a la formación de dos tipos de sitios de unión al ADN dentro del octámero; el sitio α1α1, que utiliza la hélice α1 de dos histonas adyacentes, y el sitio L1L2 formado por los bucles L1 y L2. Los enlaces de sal y los enlaces de hidrógeno entre los grupos básicos e hidroxilo de la cadena lateral y las amidas de la cadena principal con los fosfatos del esqueleto del ADN forman la mayor parte de las interacciones con el ADN. Esto es importante, dado que la distribución ubicua de nucleosomas a lo largo de los genomas requiere que sea un factor de unión al ADN no específico de secuencia. Aunque los nucleosomas tienden a preferir algunas secuencias de ADN sobre otras, son capaces de unirse prácticamente a cualquier secuencia, lo que se cree que se debe a la flexibilidad en la formación de estas interacciones mediadas por agua. Además, se producen interacciones no polares entre las cadenas laterales de las proteínas y los grupos desoxirribosa, y una cadena lateral de arginina se intercala en el surco menor del ADN en los 14 sitios donde mira hacia la superficie del octámero. La distribución y la fuerza de los sitios de unión al ADN sobre la superficie del octámero distorsionan el ADN dentro del núcleo del nucleosoma. El ADN está doblado de manera no uniforme y también contiene defectos de torsión. La torsión del ADN en forma B libre en solución es de 10,5 pb por vuelta. Sin embargo, el giro total del ADN nucleosomal es de solo 10,2 pb por turno, variando desde un valor de 9,4 a 10,9 pb por turno.
Dominios de cola de histonas
Las extensiones de la cola de las histonas constituyen hasta el 30 % en masa de las histonas, pero no son visibles en las estructuras cristalinas de los nucleosomas debido a su alta flexibilidad intrínseca, y se cree que en gran medida no están estructuradas. Las colas N-terminales de las histonas H3 y H2B pasan a través de un canal formado por los surcos menores de las dos hebras de ADN, sobresaliendo del ADN cada 20 pb. La cola N-terminal de la histona H4, por otro lado, tiene una región de aminoácidos altamente básicos (16-25), que, en la estructura cristalina, forma una interacción con la región de superficie altamente ácida de un dímero H2A-H2B. de otro nucleosoma, siendo potencialmente relevante para la estructura de orden superior de los nucleosomas. Se cree que esta interacción también ocurre en condiciones fisiológicas y sugiere que la acetilación de la cola H4 distorsiona la estructura de cromatina de orden superior.
Estructura de orden superior
La organización del ADN que logra el nucleosoma no puede explicar completamente el empaquetamiento del ADN observado en el núcleo celular. Es necesaria una mayor compactación de la cromatina en el núcleo celular, pero aún no se comprende bien. El conocimiento actual es que la repetición de nucleosomas con la intervención de un 'enlazador' El ADN forma una fibra de 10 nm, descrita como "cuentas en un hilo", y tiene una proporción de empaquetamiento de aproximadamente cinco a diez. Una cadena de nucleosomas se puede organizar en una fibra de 30 nm, una estructura compactada con una relación de empaquetamiento de ~50 y cuya formación depende de la presencia de la histona H1.
Se ha presentado y utilizado una estructura cristalina de un tetranucleosoma para construir una estructura propuesta de la fibra de 30 nm como una hélice de dos puntas. Todavía hay cierta controversia con respecto a este modelo, ya que es incompatible con los datos recientes de microscopía electrónica. Más allá de esto, la estructura de la cromatina es poco conocida, pero se sugiere clásicamente que la fibra de 30 nm se organiza en bucles a lo largo de un andamio de proteína central para formar eucromatina transcripcionalmente activa. La compactación adicional conduce a heterocromatina transcripcionalmente inactiva.
Dinámica
Aunque el nucleosoma es un complejo proteína-ADN muy estable, no es estático y se ha demostrado que sufre una serie de reordenamientos estructurales diferentes, incluido el deslizamiento del nucleosoma y la exposición del sitio del ADN. Según el contexto, los nucleosomas pueden inhibir o facilitar la unión del factor de transcripción. Las posiciones del nucleosoma están controladas por tres contribuciones principales: primero, la afinidad de unión intrínseca del octámero de histonas depende de la secuencia de ADN. En segundo lugar, el nucleosoma puede ser desplazado o reclutado por la unión competitiva o cooperativa de otros factores proteicos. Tercero, el nucleosoma puede ser translocado activamente por complejos de remodelación dependientes de ATP.
Deslizamiento de nucleosomas
El trabajo realizado en el laboratorio de Bradbury mostró que los nucleosomas reconstituidos en la secuencia de posicionamiento de ADN 5S podían reposicionarse de forma traduccional en secuencias adyacentes cuando se incubaban térmicamente. El trabajo posterior mostró que este reposicionamiento no requería la interrupción del octámero de histonas, pero era consistente con que los nucleosomas pudieran 'deslizarse'. a lo largo del ADN en cis. En 2008, se reveló además que los sitios de unión de CTCF actúan como anclajes de posicionamiento de nucleosomas de modo que, cuando se usan para alinear varias señales genómicas, se pueden identificar fácilmente múltiples nucleosomas flanqueantes. Aunque los nucleosomas son intrínsecamente móviles, los eucariotas han desarrollado una gran familia de enzimas remodeladoras de cromatina dependientes de ATP para alterar la estructura de la cromatina, muchas de las cuales lo hacen a través del deslizamiento de nucleosomas. En 2012, el laboratorio de Beena Pillai demostró que el deslizamiento de nucleosomas es uno de los posibles mecanismos para la expresión de genes específicos de tejidos a gran escala. El trabajo muestra que el sitio de inicio de la transcripción para los genes expresados en un tejido en particular se agota en el nucleosoma, mientras que el mismo conjunto de genes en otro tejido donde no se expresan se une al nucleosoma.
Exposición del sitio de ADN
El trabajo del laboratorio Widom ha demostrado que el ADN nucleosomal está en equilibrio entre un estado envuelto y desenvuelto. Las mediciones de estas tasas utilizando FRET resuelto en el tiempo revelaron que el ADN dentro del nucleosoma permanece completamente envuelto durante solo 250 ms antes de que se desenvuelva durante 10-50 ms y luego se vuelva a envolver rápidamente. Esto implica que el ADN no necesita estar disociado activamente del nucleosoma, sino que hay una fracción de tiempo significativa durante la cual es completamente accesible. De hecho, esto se puede extender a la observación de que la introducción de una secuencia de unión a ADN dentro del nucleosoma aumenta la accesibilidad de las regiones adyacentes de ADN cuando se unen. Esta propensión del ADN dentro del nucleosoma a "respirar" tiene importantes consecuencias funcionales para todas las proteínas de unión al ADN que operan en un entorno de cromatina. En particular, la respiración dinámica de los nucleosomas juega un papel importante en la restricción del avance de la ARN polimerasa II durante el alargamiento de la transcripción.
Región libre de nucleosomas
Los promotores de genes activos tienen regiones libres de nucleosomas (NFR). Esto permite la accesibilidad del ADN promotor a varias proteínas, como los factores de transcripción. La región libre de nucleosomas normalmente abarca 200 nucleótidos en S. cerevisiae Los nucleosomas bien posicionados forman los límites de NFR. Estos nucleosomas se denominan +1-nucleosoma y -1-nucleosoma y están ubicados a distancias canónicas aguas abajo y aguas arriba, respectivamente, desde el sitio de inicio de la transcripción. El nucleosoma +1 y varios nucleosomas aguas abajo también tienden a incorporar la variante de histona H2A.Z.
Estructura moduladora del nucleosoma
Los genomas eucarióticos se asocian de manera ubicua con la cromatina; sin embargo, las células deben regular espacial y temporalmente loci específicos independientemente de la cromatina en masa. Para lograr el alto nivel de control requerido para coordinar procesos nucleares como la replicación, reparación y transcripción del ADN, las células han desarrollado una variedad de medios para modular local y específicamente la estructura y función de la cromatina. Esto puede implicar la modificación covalente de histonas, la incorporación de variantes de histonas y la remodelación no covalente por enzimas de remodelación dependientes de ATP.
Modificaciones postraduccionales de histonas
Desde que se descubrieron a mediados de la década de 1960, se ha predicho que las modificaciones de las histonas afectan la transcripción. El hecho de que la mayoría de las primeras modificaciones postraduccionales encontradas se concentraran en las extensiones de la cola que sobresalen del núcleo del nucleosoma conduce a dos teorías principales sobre el mecanismo de modificación de las histonas. La primera de las teorías sugirió que pueden afectar las interacciones electrostáticas entre las colas de las histonas y el ADN para 'aflojarse'. estructura de la cromatina. Posteriormente se propuso que las combinaciones de estas modificaciones pueden crear epítopos de unión con los que reclutar otras proteínas. Recientemente, dado que se han encontrado más modificaciones en las regiones estructuradas de las histonas, se ha propuesto que estas modificaciones pueden afectar las interacciones histona-ADN e histona-histona dentro del núcleo del nucleosoma. Se predice que las modificaciones (como la acetilación o la fosforilación) que reducen la carga del núcleo de histonas globulares se 'aflojarán'. asociación núcleo-ADN; la fuerza del efecto depende de la ubicación de la modificación dentro del núcleo. Se ha demostrado que algunas modificaciones están correlacionadas con el silenciamiento de genes; otros parecen estar correlacionados con la activación de genes. Las modificaciones comunes incluyen acetilación, metilación o ubiquitinación de lisina; metilación de arginina; y fosforilación de serina. La información almacenada de esta manera se considera epigenética, ya que no está codificada en el ADN sino que aún se hereda a las células hijas. El mantenimiento de un estado reprimido o activado de un gen a menudo es necesario para la diferenciación celular.
Variantes de histonas
Aunque las histonas se conservan notablemente a lo largo de la evolución, se han identificado varias formas variantes. Esta diversificación de la función de las histonas está restringida a H2A y H3, siendo H2B y H4 mayoritariamente invariantes. H2A puede ser reemplazado por H2AZ (que conduce a una menor estabilidad del nucleosoma) o H2AX (que está asociado con la reparación del ADN y la diferenciación de células T), mientras que los cromosomas X inactivos en los mamíferos están enriquecidos en macroH2A. H3 puede ser reemplazado por H3.3 (que se correlaciona con genes activos y elementos reguladores) y en centrómeros H3 es reemplazado por CENPA.
Remodelación de nucleosomas dependiente de ATP
Varias reacciones distintas están asociadas con el término remodelación de la cromatina dependiente de ATP. Se ha demostrado que las enzimas remodeladoras deslizan los nucleosomas a lo largo del ADN, interrumpen los contactos histona-ADN hasta el punto de desestabilizar el dímero H2A/H2B y generan una torsión superhelicoidal negativa en el ADN y la cromatina. Recientemente, se ha demostrado que la enzima de remodelación Swr1 introduce la variante de histona H2A.Z en los nucleosomas. En la actualidad, no está claro si todos estos representan reacciones distintas o simplemente resultados alternativos de un mecanismo común. Lo que comparten todos, y de hecho el sello distintivo de la remodelación de la cromatina dependiente de ATP, es que todos dan como resultado una accesibilidad alterada del ADN.
Los estudios que analizan la activación génica in vivo y, lo que es más sorprendente, la remodelación in vitro han revelado que los eventos de remodelación de la cromatina y la unión del factor de transcripción son de naturaleza cíclica y periódica. Si bien se desconocen las consecuencias de esto para el mecanismo de reacción de la remodelación de la cromatina, la naturaleza dinámica del sistema puede permitirle responder más rápido a los estímulos externos. Un estudio reciente indica que las posiciones de los nucleosomas cambian significativamente durante el desarrollo de células madre embrionarias de ratón, y estos cambios están relacionados con la unión de factores de transcripción del desarrollo.
Remodelación dinámica de nucleosomas en el genoma de la levadura
Los estudios realizados en 2007 catalogaron las posiciones de los nucleosomas en la levadura y demostraron que los nucleosomas están agotados en las regiones promotoras y los orígenes de la replicación. Alrededor del 80% del genoma de la levadura parece estar cubierto por nucleosomas y el patrón de posicionamiento de los nucleosomas se relaciona claramente con las regiones de ADN que regulan la transcripción, las regiones que se transcriben y las regiones que inician la replicación del ADN. Más recientemente, un nuevo estudio examinó los cambios dinámicos en el reposicionamiento de nucleosomas durante un evento de reprogramación transcripcional global para dilucidar los efectos sobre el desplazamiento de nucleosomas durante los cambios transcripcionales en todo el genoma de la levadura (Saccharomyces cerevisiae). Los resultados sugirieron que los nucleosomas que estaban localizados en las regiones promotoras se desplazan en respuesta al estrés (como un golpe de calor). Además, la eliminación de los nucleosomas generalmente correspondía a la activación transcripcional y el reemplazo de los nucleosomas generalmente correspondía a la represión transcripcional, presumiblemente porque los sitios de unión del factor de transcripción se volvieron más o menos accesibles, respectivamente. En general, solo se reposicionaron uno o dos nucleosomas en el promotor para efectuar estos cambios transcripcionales. Sin embargo, incluso en regiones cromosómicas que no estaban asociadas con cambios transcripcionales, se observó un reposicionamiento de nucleosomas, lo que sugiere que la cobertura y el descubrimiento del ADN transcripcional no necesariamente produce un evento transcripcional. Después de la transcripción, la región del ADNr debe protegerse de cualquier daño, lo que sugiere que las proteínas HMGB desempeñan un papel importante en la protección de la región libre de nucleosomas.
Defectos de torsión del ADN
Los defectos de torsión del ADN se producen cuando la adición de uno o unos pocos pares de bases de un segmento de ADN se transfiere al segmento siguiente, lo que da como resultado un cambio en la torsión del ADN. Esto no solo cambiará la torsión del ADN, sino que también cambiará la longitud. Este defecto de giro finalmente se mueve alrededor del nucleosoma a través de la transferencia del par de bases, lo que significa que los giros del ADN pueden causar el deslizamiento del nucleosoma. Las estructuras cristalinas del nucleosoma han demostrado que las ubicaciones 2 y 5 de la superhélice en el nucleosoma se encuentran comúnmente donde ocurren los defectos de torsión del ADN, ya que estos son sitios comunes de unión del remodelador. Hay una variedad de remodeladores de la cromatina, pero todos comparten la existencia de un motor ATPasa que facilita el deslizamiento de la cromatina en el ADN a través de la unión e hidrólisis de ATP. ATPasa tiene un estado abierto y cerrado. Cuando el motor de la ATPasa cambia de estado abierto y cerrado, el dúplex de ADN cambia de geometría y muestra una inclinación del par de bases. El inicio de los defectos de torsión a través del motor ATPasa hace que se acumule tensión alrededor del sitio del remodelador. La tensión se libera cuando el deslizamiento del ADN se ha completado por todo el nucleosoma a través de la propagación de dos defectos de torsión (uno en cada hebra) en direcciones opuestas.
Ensamblaje de nucleosomas in vitro
Los nucleosomas se pueden ensamblar in vitro utilizando histonas nativas o recombinantes purificadas. Una técnica estándar para cargar el ADN alrededor de las histonas implica el uso de diálisis con sal. Una reacción que consta de los octámeros de histonas y una plantilla de ADN desnudo se puede incubar juntos a una concentración de sal de 2 M. Al disminuir constantemente la concentración de sal, el ADN se equilibrará en una posición en la que se envuelve alrededor de los octámeros de histonas, formando nucleosomas. En condiciones apropiadas, este proceso de reconstitución permite mapear experimentalmente la afinidad de posicionamiento del nucleosoma de una secuencia dada.
Partículas de núcleo de nucleosoma entrecruzadas con disulfuro
Un avance reciente en la producción de partículas de núcleo de nucleosoma con estabilidad mejorada involucra enlaces cruzados de disulfuro específicos del sitio. Se pueden introducir dos enlaces cruzados diferentes en la partícula del núcleo del nucleosoma. Una primera entrecruza las dos copias de H2A a través de una cisteína introducida (N38C) que da como resultado un octámero de histona que es estable frente a la pérdida de dímero H2A/H2B durante la reconstitución del nucleosoma. Se puede introducir un segundo entrecruzamiento entre la cola de la histona N-terminal de H3 y los extremos del ADN del nucleosoma a través de un nucleótido convertible incorporado. El entrecruzamiento del octámero de histonas de ADN estabiliza la partícula del núcleo del nucleosoma contra la disociación del ADN a concentraciones de partículas muy bajas y concentraciones de sal elevadas.
Ensamblaje de nucleosomas in vivo
Los nucleosomas son la unidad de empaquetamiento básica del ADN construida a partir de proteínas histonas alrededor de las cuales se enrolla el ADN. Sirven como un andamio para la formación de una estructura de cromatina de orden superior, así como para una capa de control regulador de la expresión génica. Los nucleosomas se ensamblan rápidamente en el ADN recién sintetizado detrás de la horquilla de replicación.
H3 y H4
Las histonas H3 y H4 de los nucleosomas antiguos desmontados se mantienen en las inmediaciones y se distribuyen aleatoriamente en el ADN recién sintetizado. Son ensamblados por el complejo del factor de ensamblaje de cromatina-1 (CAF-1), que consta de tres subunidades (p150, p60 y p48). El H3 y H4 recién sintetizados se ensamblan mediante el factor de ensamblaje de acoplamiento de replicación (RCAF). RCAF contiene la subunidad Asf1, que se une a las proteínas H3 y H4 recién sintetizadas. Las antiguas proteínas H3 y H4 conservan sus modificaciones químicas, lo que contribuye a la transmisión de la firma epigenética. Las proteínas H3 y H4 recién sintetizadas se acetilan gradualmente en diferentes residuos de lisina como parte del proceso de maduración de la cromatina. También se cree que las viejas proteínas H3 y H4 en los nuevos nucleosomas reclutan enzimas modificadoras de histonas que marcan las nuevas histonas, lo que contribuye a la memoria epigenética.
H2A y H2B
A diferencia de las H3 y H4 antiguas, las histonas H2A y H2B antiguas se liberan y degradan; por lo tanto, las proteínas H2A y H2B recién ensambladas se incorporan a nuevos nucleosomas. H2A y H2B se ensamblan en dímeros que luego se cargan en los nucleosomas mediante la proteína de ensamblaje de nucleosomas-1 (NAP-1), que también ayuda con el deslizamiento de los nucleosomas. Los nucleosomas también están espaciados por complejos de remodelación de nucleosomas dependientes de ATP que contienen enzimas como Isw1 Ino80 y Chd1, y posteriormente se ensamblan en una estructura de orden superior.
Galería
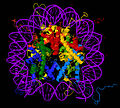
La estructura cristalina de la partícula del núcleo del nucleosoma (PDB: 1EQZ): diferentes vistas que muestran detalles del plegamiento y la organización de las histonas. Histonas H2A, H2B, H3, H4 y DNA están coloreados.
Contenido relacionado
Zooplancton
Dibujo de embriones
Nemertea