Nucleoide
El nucleoide (que significa parecido a un núcleo) es una región de forma irregular dentro de la célula procariótica que contiene todo o la mayor parte del material genético. El cromosoma de un procariota típico es circular y su longitud es muy grande en comparación con las dimensiones de la célula, por lo que es necesario compactarlo para que quepa. A diferencia del núcleo de una célula eucariota, no está rodeado por una membrana nuclear. En cambio, el nucleoide se forma por condensación y disposición funcional con la ayuda de proteínas arquitectónicas cromosómicas y moléculas de ARN, así como del superenrollamiento del ADN. La longitud de un genoma varía ampliamente (generalmente al menos unos pocos millones de pares de bases) y una célula puede contener múltiples copias del mismo.
Aún no se conoce una estructura de alta resolución de un nucleoide bacteriano; sin embargo, se han investigado características clave en Escherichia coli como organismo modelo. En E. coli, el ADN cromosómico está, en promedio, superenrollado negativamente y plegado en bucles plectonémicos, que están confinados a diferentes regiones físicas y rara vez se difunden entre sí. Estos bucles se organizan espacialmente en regiones del tamaño de megabases llamadas macrodominios, dentro de las cuales los sitios de ADN interactúan con frecuencia, pero entre las cuales las interacciones son raras. El ADN condensado y organizado espacialmente forma un elipsoide helicoidal que está confinado radialmente en la célula. La estructura tridimensional del ADN en el nucleoide parece variar según las condiciones y está vinculada a la expresión genética, de modo que la arquitectura del nucleoide y la transcripción genética son estrechamente interdependientes y se influyen mutuamente de forma recíproca.

Antecedentes
En muchas bacterias, el cromosoma es una única molécula de ADN bicatenario covalentemente cerrada (circular) que codifica la información genética en forma haploide. El tamaño del ADN varía de 500.000 a varios millones de pares de bases (pb) y codifica de 500 a varios miles de genes, según el organismo. El ADN cromosómico está presente en las células en una forma muy compacta y organizada llamada nucleoide (que significa parecido a un núcleo), que no está recubierto por una membrana nuclear como en las células eucariotas. El nucleoide aislado contiene 80% de ADN, 10% de proteína y 10% de ARN en peso.
La bacteria gramnegativa Escherichia coli es un sistema modelo para la investigación de nucleoides sobre cómo el ADN cromosómico se convierte en nucleoide, los factores implicados en él, lo que se sabe sobre su estructura y cómo parte del ADN Los aspectos estructurales influyen en la expresión genética.
Hay dos aspectos esenciales de la formación de nucleoides; Condensación de un ADN grande en un espacio celular pequeño y organización funcional del ADN en forma tridimensional. El cromosoma circular haploide en E. coli consta de ~ 4,6 x 106 pb. Si el ADN se relaja en la forma B, tendría una circunferencia de ~1,5 milímetros (0,332 nm x 4,6 x 106). Sin embargo, una molécula de ADN grande como la E. coli el ADN cromosómico no permanece como una molécula recta y rígida en suspensión. El movimiento browniano generará curvaturas y dobleces en el ADN. La longitud máxima hasta la cual un ADN de doble hélice permanece recto resistiendo la flexión impuesta por el movimiento browniano es ~50 nm o 150 pb, lo que se denomina longitud de persistencia. Así, el ADN puro se condensa sustancialmente sin ningún factor adicional; en equilibrio térmico, asume una forma de bobina aleatoria. La bobina aleatoria de E. coli el ADN cromosómico ocuparía un volumen (4/3 π r3) de ~ 523 μm3, calculado a partir del radio de giro (R g = (√N a)/√6) donde a es la longitud de Kuhn (2 x longitud de persistencia), y N es el número de segmentos de longitud de Kuhn en el ADN (longitud total del ADN dividida por a). Aunque el ADN ya está condensado en forma de espiral aleatoria, todavía no puede asumir el volumen del nucleoide, que es inferior a una micra. Por lo tanto, la propiedad inherente del ADN no es suficiente: factores adicionales deben ayudar a condensar aún más el ADN del orden de ~103 (volumen de la bobina aleatoria dividido por el volumen del nucleoide). El segundo aspecto esencial de la formación de nucleoides es la disposición funcional del ADN. El ADN cromosómico no sólo está condensado sino también funcionalmente organizado de manera compatible con los procesos de transacción del ADN, como la replicación, la recombinación, la segregación y la transcripción. Casi cinco décadas de investigaciones iniciadas en 1971, han demostrado que la forma final del nucleoide surge de una organización jerárquica del ADN. En la escala más pequeña (1 kb o menos), las proteínas arquitectónicas del ADN asociadas a nucleoides condensan y organizan el ADN doblando, formando bucles, formando puentes o envolviéndolo. A mayor escala (10 kb o más), el ADN forma bucles plectonémicos, una forma trenzada de ADN inducida por superenrollamiento. En la escala de megabase, los bucles plectonémicos se fusionan en seis dominios organizados espacialmente (macrodominios), que se definen por interacciones físicas más frecuentes entre sitios de ADN dentro del mismo macrodominio que entre diferentes macrodominios. Las conexiones ADN-ADN de largo y corto alcance formadas dentro y entre los macrodominios contribuyen a la condensación y la organización funcional. Finalmente, el nucleoide es un elipsoide helicoidal con regiones de ADN muy condensado en el eje longitudinal.
Condensación y organización

Proteínas asociadas a la nucleoide (NAPs)
En los eucariotas, el ADN genómico se condensa en forma de una serie repetida de partículas de proteínas de ADN llamadas nucleosomas.
Un nucleosoma consta de ~146 pb de ADN envuelto alrededor de un complejo octamérico de proteínas histonas. Aunque las bacterias no tienen histonas, poseen un grupo de proteínas de unión al ADN denominadas proteínas asociadas a nucleoides (NAP) que son funcionalmente análogas a las histonas en un sentido amplio. Las NAP son muy abundantes y constituyen una proporción significativa del componente proteico del nucleoide.
Una característica distintiva de los NAP es su capacidad para unirse al ADN tanto de manera específica (ya sea de secuencia o de estructura específica) como de manera no específica de secuencia. Como resultado, las NAP son proteínas de doble función. La unión específica de NAP está implicada principalmente en la transcripción, replicación, recombinación y reparación de genes específicos. En el pico de su abundancia, el número de moléculas de muchas NAP es varios órdenes de magnitud mayor que el número de sitios de unión específicos en el genoma. Por lo tanto, se razona que las NAP se unen al ADN cromosómico principalmente en un modo no específico de secuencia y es este modo el que es crucial para la compactación cromosómica. Es de destacar que la denominada unión no específica de secuencia de una NAP puede no ser completamente aleatoria. Podría haber una especificidad de secuencia baja o una especificidad estructural debido a la conformación del ADN dependiente de la secuencia o la conformación del ADN creada por otros NAP.
Aunque los mecanismos moleculares de cómo las NAP condensan el ADN in vivo no se comprenden bien, según los extensos estudios in vitro parece que las NAP participan en la compactación cromosómica a través de lo siguiente Mecanismos: los NAP inducen y estabilizan curvaturas en el ADN, lo que ayuda a la condensación del ADN al reducir la duración de la persistencia. Los NAP condensan el ADN mediante puentes, envolturas y agrupaciones que podrían ocurrir entre segmentos de ADN cercanos o segmentos de ADN distantes del cromosoma. Otro mecanismo por el cual los NAP participan en la compactación cromosómica es restringiendo los superenrollamientos negativos en el ADN, contribuyendo así a la organización topológica del cromosoma.
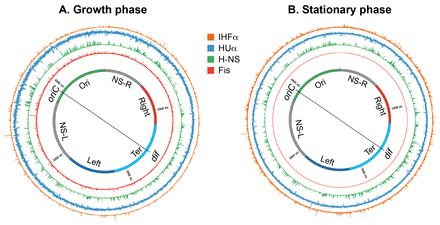
Hay al menos 12 NAP identificados en E. coli, de los cuales los más estudiados son HU, IHF, H-NS y Fis. Su abundancia y propiedades de unión al ADN y su efecto sobre la condensación y organización del ADN se resumen en las tablas siguientes.
| Proteína | Masa molecular (kDa) | Unidad funcional nativa | Abundancia1 en la fase de crecimiento | Abundancia1 en fase estacionaria |
|---|---|---|---|---|
| HUα y HUβ | ~ 9 | Homo y hetero-dimer | 55.000 (23) | 30.000 (12.5) |
| IHFα e IHFβ | ~ 11 | Heterodimer | 12,000 (5) | 55.000 (23) |
| H-NS | ~ 15 | Homodimer | 20.000 (8) | 15.000 (6) |
| Fis | ~ 11 | Homodimer | 60.000 (25) | Indetectable |
| Dps | ~ 19 | Dodecamer | 6.000 (0,4) | 180.000 (12.5) |
1 Los datos de abundancia (moléculas/células) se tomaron de; El número entre paréntesis es la concentración micromolar calculada mediante la siguiente fórmula: (número de unidades funcionales nativas/número de Avogadro) x (1/volumen de celda en litro) x 103. El volumen celular en litros (2 x 10−15) se determinó asumiendo el volumen de E. coli es de 2 μm3.
| Proteína | Motivo de unión | Afinidad específica de ADN vinculante1 | Afinidad de ADN vinculante aleatoria1 |
|---|---|---|---|
| HU | Un motivo estructural definido por curvas y broches en ADN | 7.5 x 10−9 | 4.0 x 10−7 |
| SI | WATCAANNNNTTR | 1,5 x 10−9 | 1,7 x 10−6 |
| H-NS | TCGATAAATT | 10-15 x 10−9 | 6 x 10−8 |
| Fis | GNTYAAAWTTTRANC | 0,2-1.0 x 10−9 | 8.0 x 10−6 |
| Dps | ND | ND | 1.65 x 10−7 |
| MatP | GTGACRNYGTCAC | 8.0 x 10−9 | ND |
| MukBEF | ND | ND | ND |
1 La afinidad de unión se refiere a la constante de disociación de equilibrio (Kd) en unidades molares (M). ND = no determinado
HU
Proteína similar a las histonas de E. coli cepa U93 (HU) es una proteína conservada evolutivamente en bacterias. HU existe en E. coli como homo y heterodímeros de dos subunidades HUα y HUβ que comparten un 69% de identidad de aminoácidos. Aunque se la conoce como una proteína similar a las histonas, los parientes funcionales cercanos de HU en eucariotas son proteínas del grupo de alta movilidad (HMG) y no histonas. HU es una proteína de unión al ADN no específica de secuencia. Se une con baja afinidad a cualquier ADN lineal. Sin embargo, se une preferentemente con alta afinidad a un ADN estructuralmente distorsionado. Ejemplos de sustratos de ADN distorsionados incluyen ADN cruciforme, ADN abultado, ADN ds que contiene una rotura monocatenaria como muescas, espacios o bifurcaciones. Además, HU se une y estabiliza específicamente un bucle de ADN mediado por proteínas. En el modo de unión al ADN estructuralmente específico, HU reconoce un motivo estructural común definido por curvaturas o torceduras creadas por distorsión, mientras que se une a un ADN lineal bloqueando la columna vertebral de fosfato. Si bien la unión estructuralmente específica de alta afinidad es necesaria para funciones especializadas de HU, como la recombinación específica de sitio, la reparación del ADN, el inicio de la replicación del ADN y la regulación genética, parece que la unión general de baja afinidad está involucrada en la condensación del ADN. En la inmunoprecipitación de cromatina junto con la secuenciación de ADN (ChIP-Seq), HU no revela ningún evento de unión específico. En cambio, muestra una unión uniforme en todo el genoma, lo que presumiblemente refleja su unión en su mayoría débil y no específica de secuencia, enmascarando así la unión de alta afinidad in vivo.
En las cepas que carecen de HU, el nucleoide está "descondensado", lo que coincide con el papel de la HU en la compactación del ADN. Los siguientes estudios in vitro sugieren posibles mecanismos de cómo HU podría condensar y organizar el ADN in vivo. HU no solo se une de manera estable al ADN distorsionado con curvaturas, sino que también induce curvaturas flexibles incluso en un ADN lineal a una concentración inferior a 100 nM. Por el contrario, HU muestra el efecto arquitectónico opuesto sobre el ADN en concentraciones fisiológicamente relevantes más altas. Forma filamentos rígidos de nucleoproteínas que provocan el estiramiento del ADN y no su flexión. Los filamentos pueden formar además una red de ADN (Agrupación de ADN) expandible tanto lateral como medialmente debido a la multimerización HU-HU provocada por la unión de ADN no específica de secuencia.
¿Cómo son relevantes estos comportamientos de HU dentro de la célula? La formación de filamentos requiere una unión de alta densidad de HU al ADN, un dímero de HU por ADN de 9 a 20 pb. Pero sólo hay un dímero de HU cada ~150 pb del ADN cromosómico según la abundancia estimada de 30.000 dímeros de HU por célula (4.600.000 pb/30.000). Esto indica que es más probable que las curvaturas flexibles ocurran in vivo. La flexión flexible provocaría condensación debido a una reducción en la longitud de persistencia del ADN, como lo demuestran los experimentos con pinzas magnéticas, que permiten estudiar la condensación de una sola molécula de ADN por una proteína de unión al ADN. Sin embargo, debido a la cooperatividad, los filamentos y redes rígidos podrían formarse en algunas regiones del cromosoma. La formación de filamentos por sí sola no induce la condensación, pero la formación de redes o agrupaciones de ADN puede contribuir sustancialmente a la condensación al unir segmentos cromosómicos distantes o cercanos.
IHF
El factor huésped de integración (IHF) es estructuralmente casi idéntico al HU, pero se comporta de manera diferente en muchos aspectos. A diferencia de HU, que se une preferentemente a un motivo estructural independientemente de la secuencia, IHF se une preferentemente a una secuencia de ADN específica aunque la especificidad surge a través de la estructura y deformabilidad del ADN dependiente de la secuencia. La unión específica de IHF en sitios afines dobla bruscamente el ADN en >160 grados. Una aparición del motivo de secuencia afín es aproximadamente 3000 en el E. coli genoma. La abundancia estimada de IHF en la fase de crecimiento es de aproximadamente 6000 dímeros por célula. Suponiendo que un dímero de IHF se une a un solo motivo y que el nucleoide contiene más de un equivalente genómico durante la fase de crecimiento exponencial, la mayoría de las moléculas de IHF ocuparían sitios específicos en el genoma y probablemente solo condensarían el ADN induciendo una flexión brusca.
Además de la unión preferencial a una secuencia de ADN específica, IHF también se une al ADN de una manera no específica de secuencia con afinidades similares a HU. El papel de la unión no específica de IHF en la condensación del ADN parece ser crítico en la fase estacionaria porque la abundancia de IHF aumenta cinco veces en la fase estacionaria y los dímeros de IHF adicionales probablemente se unirían al ADN cromosómico de manera no específica. A diferencia del HU, el IHF no forma filamentos rígidos gruesos en concentraciones más altas. En cambio, su unión no específica también induce la flexión del ADN, aunque el grado de flexión es mucho menor que el de sitios específicos y es similar a la flexión flexible inducida por HU en un ADN lineal en bajas concentraciones. In vitro, la flexión inducida por la unión no específica del IHF puede provocar la condensación del ADN y promueve la formación de complejos de nucleoproteínas de orden superior dependiendo de las concentraciones de cloruro de potasio y cloruro de magnesio. La organización del ADN de orden superior realizada por IHF in vivo aún no está clara.
H-NS
Una característica distinguible de la proteína estructurante de nucleoides (H-NS) similar a histonas o termoestable de otras NAP es la capacidad de cambiar de la forma homodimérica en concentraciones relativamente bajas (<1 x 10-5< /sup>M) a un estado oligomérico en niveles superiores. Debido a las propiedades de oligomerización, H-NS se propaga lateralmente a lo largo del ADN rico en AT en una reacción de nucleación, donde los sitios de alta afinidad funcionan como centros de nucleación. La propagación de H-NS en el ADN produce dos resultados opuestos dependiendo de la concentración de magnesio en la reacción. A bajas concentraciones de magnesio (<2 mM), H-NS forma filamentos de nucleoproteínas rígidos, mientras que forma puentes inter e intramoleculares a concentraciones más altas de magnesio (> 5 mM). La formación de filamentos rígidos da como resultado el enderezamiento del ADN sin condensación, mientras que los puentes provocan un plegamiento sustancial del ADN. El análisis de la unión de H-NS en el genoma mediante ensayos ChIP-Seq proporcionó evidencia indirecta de la propagación de H-NS en el ADN in vivo. H-NS se une selectivamente a 458 regiones del genoma. Aunque se ha demostrado que H-NS prefiere el ADN curvo formado por pistas A repetidas en secuencias de ADN, la base de la unión selectiva es la presencia de un motivo de secuencia conservado que se encuentra en regiones ricas en AT. Más importante aún, la aparición frecuente del motivo de secuencia dentro de una región de unión a H-NS que puede reforzar las interacciones cooperativas proteína-proteína, y la longitud inusualmente larga de la región de unión son consistentes con la propagación de la proteína. Que la formación de filamentos o los puentes de ADN prevalezca in vivo depende de la concentración fisiológica de magnesio dentro de la célula. Si la concentración de magnesio es uniformemente baja (<5 mM), H-NS formaría filamentos rígidos de nucleoproteínas in vivo. Alternativamente, si hay una distribución desigual de magnesio en la célula, podría promover tanto la unión como el endurecimiento del ADN, pero en diferentes regiones del nucleoide.
Además, H-NS es mejor conocido como un silenciador genético global que inhibe preferentemente la transcripción de genes transferidos horizontalmente y es el filamento rígido que conduce al silenciamiento genético. En conjunto, parece que la formación de filamentos rígidos es el resultado más probable de las interacciones H-NS-ADN in vivo que conducen al silenciamiento de genes pero no inducen la condensación del ADN. Consistentemente, la ausencia de H-NS no cambia el volumen del nucleoide. Sin embargo, es posible que E. coli experimenta altas concentraciones de magnesio en algunas condiciones ambientales. En tales condiciones, H-NS puede cambiar de su forma inductora de filamento a la forma inductora de puente que contribuye a la condensación y organización del ADN.
Fis
El factor de estimulación de la inversión (Fis) es una proteína de unión al ADN de secuencia específica que se une a secuencias de ADN específicas que contienen un motivo simétrico de 15 pb. Al igual que IHF, Fis induce la flexión del ADN en sitios afines. La capacidad de doblar el ADN es evidente en la estructura del homodímero de Fis. Un homodímero de Fis posee dos motivos hélice-vuelta-hélice (HTH), uno de cada monómero. Un motivo HTH normalmente reconoce el surco principal del ADN. Sin embargo, la distancia entre las hélices de reconocimiento de ADN de los dos motivos HTH en el homodímero Fis es de 25 Å, es decir, ~ 8 Å más corta que el paso de un ADN B canónico, lo que indica que la proteína debe doblar o torcer el ADN para unirse de manera estable.. Consistentemente, la estructura cristalina de los complejos Fis-ADN muestra que la distancia entre las hélices de reconocimiento permanece sin cambios, mientras que el ADN se curva en el rango de 60 a 75 grados. Hay 1464 regiones de unión de Fis distribuidas en el E. coli y un motivo de unión, identificado computacionalmente, coincide con el motivo conocido de 15 pb. La unión específica de Fis en dichos sitios induciría curvaturas en el ADN y, por lo tanto, contribuiría a la condensación del ADN al reducir la longitud de persistencia del ADN. Además, muchos sitios de unión de Fis se producen en tándem, como los de los promotores de ARN estables, por ejemplo, el promotor P1 del operón de ARNr rrnB. Es probable que la flexión coherente de Fis en los sitios en tándem cree un microbucle de ADN que puede contribuir aún más a la condensación del ADN.
Además de la unión específica de alta afinidad a sitios afines, Fis puede unirse a una secuencia de ADN aleatoria. La unión no específica al ADN es significativa porque Fis es tan abundante como HU en la fase de crecimiento. Por lo tanto, se espera que la mayoría de las moléculas de Fis se unan al ADN de una manera no específica de secuencia. Los experimentos con pinzas magnéticas muestran que esta unión no específica de Fis puede contribuir a la condensación y organización del ADN. Fis provoca una condensación leve de una sola molécula de ADN a <1 mM, pero induce un plegamiento sustancial mediante la formación de bucles de ADN de un tamaño promedio de ~800 pb a >1 mM. Los bucles en los experimentos con pinzas magnéticas son distintos de los microbucles creados por la flexión coherente del ADN en sitios afines, ya que requieren la formación de complejos de ADN-proteína de alta densidad logrados mediante unión independiente de la secuencia. Aunque aún está por demostrar la aparición de dichos bucles in vivo, la unión de alta densidad de Fis puede ocurrir in vivo mediante la acción concertada de una unión tanto específica como no específica. La aparición en tándem de sitios específicos podría iniciar una reacción de nucleación similar a la de H-NS, y luego la unión no específica conduciría a la formación de matrices de Fis localizadas de alta densidad. El puente entre estas regiones localizadas puede crear grandes bucles de ADN. Fis está presente exclusivamente en la fase de crecimiento y no en la fase estacionaria. Por lo tanto, cualquier papel en la condensación cromosómica por parte de Fis debe ser específico de las células en crecimiento.
ARN asociados a nucleoides (naRNA)
Los primeros estudios que examinaron el efecto del tratamiento con ARNasa A en nucleoides aislados indicaron que el ARN participaba en la estabilización del nucleoide en el estado condensado. Además, el tratamiento con ARNasa A rompió las fibras de ADN en fibras más delgadas, como se observó mediante una microscopía de fuerza atómica del nucleoide utilizando el "procedimiento de lisis en sustrato". Estos hallazgos demostraron la participación del ARN en la estructura nucleoide, pero la identidad de las moléculas de ARN permaneció desconocida hasta hace poco. La mayoría de los estudios sobre HU se centraron en su unión al ADN. Sin embargo, HU también se une a híbridos de ARNds y ARN-ADN con una afinidad menor similar a la de un ADNds lineal. Además, HU se une preferentemente a estructuras secundarias que contienen ARN y a un híbrido de ARN-ADN en el que el ARN contiene una mella o saliente. Las afinidades de unión de HU con estos sustratos de ARN son similares a aquellas con las que se une al ADN distorsionado. Una inmunoprecipitación de ARN unido a HU junto con un estudio de microarrays y transcripción inversa (RIP-Chip), así como un análisis de ARN de nucleoides intactos purificados, identificaron moléculas de ARN asociadas a nucleoides que interactúan con HU. Varios de ellos son ARN no codificantes, y uno de esos ARN, denominado naRNA4 (ARN 4 asociado a nucleoides), está codificado en un palíndromo extragénico repetitivo (REP325). En una cepa que carece de REP325, el nucleoide se descondensa como en una cepa que carece de HU. Lo más probable es que naRNA4 participe en la condensación del ADN conectando segmentos de ADN en presencia de HU. Estudios recientes proporcionan información sobre el mecanismo molecular de cómo naRNA4 establece conexiones ADN-ADN. El ARN se dirige a regiones del ADN que contienen estructuras cruciformes y forma un complejo ARN-ADN que es fundamental para establecer conexiones ADN-ADN. Sorprendentemente, aunque HU ayuda en la formación del complejo, no está presente en el complejo final, lo que indica su papel potencial como catalizador (chaperona). La naturaleza del complejo ARN-ADN sigue siendo desconcertante porque la formación del complejo no implica un emparejamiento extenso de bases Watson/Crick pero es sensible a la RNasa H, que escinde el ARN en un híbrido ARN-ADN y el complejo se une a un anticuerpo específico para Híbridos ARN-ADN.
Superenrollamiento
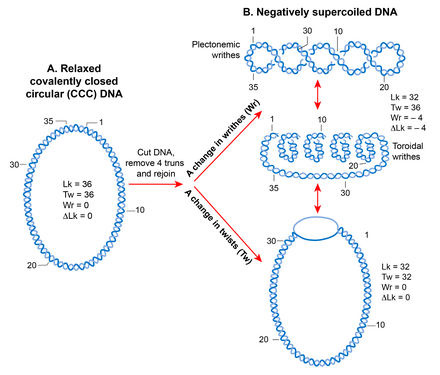
Debido a su estructura helicoidal, una molécula de ADN de doble hebra queda topológicamente restringida en una forma circular covalentemente cerrada que elimina la rotación de los extremos libres. El número de veces que las dos hebras se cruzan en un ADN topológicamente restringido se denomina número de enlace (Lk), que equivale al número de vueltas helicoidales o giros en una molécula circular. La Lk de un ADN topológico permanece invariante, sin importar cómo se deforme la molécula de ADN, siempre que ninguna de las hebras esté rota.
La Lk del ADN en su forma relajada se define como Lk0. Para cualquier ADN, Lk0 se puede calcular dividiendo la longitud (en pb) del ADN por el número de pb por vuelta helicoidal. Esto equivale a 10,4 pb para el ADN en forma B relajada. Cualquier desviación de Lk0 provoca un superenrollamiento en el ADN. Una disminución en el número de enlace (Lk<Lk0) crea un superenrollamiento negativo, mientras que un aumento en el número de enlace (Lk>Lk0) crea un superenrollamiento positivo.
El estado superenrollado (cuando Lk no es igual a Lk0) da como resultado una transición en la estructura del ADN que puede manifestarse como un cambio en el número de vueltas (negativo <10,4 pb/vuelta, positivo >10,4 pb por vuelta) y/o en la formación de retorcimientos, llamados superenrollamientos. Por lo tanto, Lk se define matemáticamente como una suma dependiente del signo de los dos parámetros geométricos, torsión y contorsión. Una medida cuantitativa del superenrollamiento que es independiente del tamaño de las moléculas de ADN es la densidad de superenrollamiento (σ) donde σ =∆Lk/Lk0.
Writhes puede adoptar dos estructuras; plectonema y solenoide o toroide. Una estructura plectonémica surge del entrelazado del eje helicoidal. Las superenrollamientos toroidales se originan cuando el ADN forma varias espirales, alrededor de un eje y que no se cruzan entre sí, como las de un cable telefónico. Las contorsiones en la forma de plectonemas son diestras y zurdas en ADN superenrollado positiva o negativamente, respectivamente. La lateralidad de las superenrollamientos toroidales es opuesta a la de los plectonemas. Tanto los plectonemas como las superenrollamientos toroidales pueden estar en forma libre o restringidos en forma unida con proteínas. El mejor ejemplo de superenrollamiento toroidal ligado en biología es el nucleosoma eucariota en el que el ADN se envuelve alrededor de histonas.
Superenrollamientos plectónicos en E. coli

En la mayoría de las bacterias, el ADN está presente en forma superenrollada. La naturaleza circular del E. coli lo convierte en una molécula topológicamente restringida que está en su mayor parte superenrollada negativamente con una densidad de superenrollamiento promedio estimada (σ) de -0,05. En la cromatina eucariota, el ADN se encuentra principalmente en la forma toroidal que está restringida y definida por histonas mediante la formación de nucleosomas. En cambio, en el E. coli nucleoide, aproximadamente la mitad del ADN cromosómico está organizado en forma de superenrollamientos plectonémicos libres. El ADN restante está restringido en la forma plectonémica o en formas alternativas, incluida, entre otras, la forma toroidal, mediante la interacción con proteínas como las NAP. Por tanto, los superenrollamientos plectonémicos representan un superenrollamiento efectivo de la E. coli genoma que es responsable de su condensación y organización. Tanto el superenrollamiento plectonémico como el toroidal ayudan en la condensación del ADN. Es de destacar que debido a la ramificación de las estructuras plectonémicas, proporciona menos condensación de ADN que la estructura toroidal. Una molécula de ADN del mismo tamaño con iguales densidades de superenrollamiento es más compacta en forma toroidal que en forma plectonémica. Además de condensar el ADN, el superenrollamiento ayuda en la organización del ADN. Promueve el desenredo del ADN al reducir la probabilidad de catenación. El superenrollamiento también ayuda a acercar dos sitios distantes de ADN, promoviendo así una posible interacción funcional entre diferentes segmentos de ADN.
Fuentes de superenrollamiento en E. coli
Tres factores contribuyen a generar y mantener el superenrollamiento del ADN cromosómico en E. coli: (i) actividades de las topoisomerasas, (ii) el acto de transcripción y (iii) NAP.
Topoisomerasas
Las topoisomerasas son una categoría particular de enzimas metabólicas del ADN que crean o eliminan el superenrollamiento rompiendo y luego volviendo a unir las hebras de ADN. E. coli posee cuatro topoisomerasas. La ADN girasa introduce un superenrollamiento negativo en presencia de ATP y elimina un superenrollamiento positivo en ausencia de ATP. En todas las formas de vida, la ADN girasa es la única topoisomerasa que puede crear superenrollamientos negativos y es debido a esta capacidad única que los genomas bacterianos poseen superenrollamientos negativos libres; La ADN girasa se encuentra en todas las bacterias, pero está ausente en los eucariotas superiores. Por el contrario, Topo I se opone a la ADN girasa relajando el ADN superenrollado negativamente. Existe evidencia genética que sugiere que un equilibrio entre las actividades opuestas de la ADN girasa y Topo I es responsable de mantener un nivel estable de superhelicidad negativa promedio en E. coli. Ambas enzimas son esenciales para E. coli supervivencia. Una cepa nula de topA, el gen que codifica Topo I, sobrevive sólo debido a la presencia de mutaciones supresoras en los genes que codifican la ADN girasa. Estas mutaciones dan como resultado una actividad reducida de la girasa, lo que sugiere que el exceso de superenrollamiento negativo debido a la ausencia de Topo I se compensa con una actividad reducida del superenrollamiento negativo de la ADN girasa. Topo III es prescindible en E. coli y no se sabe que tenga ningún papel en el superenrollamiento en E. coli. La función principal de Topo IV es resolver los cromosomas hermanos. Sin embargo, se ha demostrado que también contribuye al nivel de superenrollamiento negativo en estado estacionario al relajar el superenrollamiento negativo junto con Topo I.
| Topoisomerase | Tipo | Función | Escote mono-o doble |
|---|---|---|---|
| Topoisomerasa I | IA | Elimina (-) supercoiling | SS |
| Topoisomerasa III | IA | Elimina (-) supercoiling | SS |
| Topoisomerasa IV | IIA | Elimina (-) supercoiling | DS |
| Síndrome de ADN | IIA | Crea (-) supercoiling y elimina (+) supercoiling | DS |
Transcripción
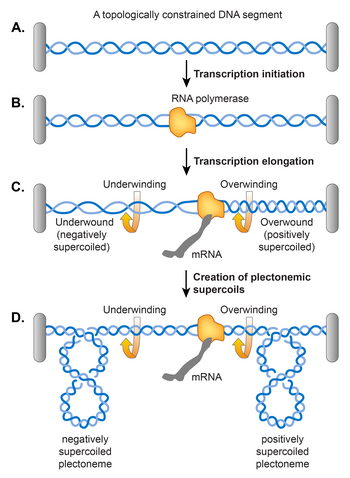
Un modelo de dominio de superenrollamiento gemelo propuesto por Liu y Wang argumentó que el desenrollamiento de la doble hélice del ADN durante la transcripción induce el superenrollamiento en el ADN como se muestra en. Según su modelo, la transcripción de la ARN polimerasa (RNAP) que se desliza a lo largo del ADN obliga al ADN a girar su eje helicoidal. Podría surgir un obstáculo en la libre rotación del ADN debido a una restricción topológica, lo que provocaría que el ADN delante de RNAP se torciera demasiado (superenrollado positivamente) y el ADN detrás de RNAP se torciera insuficientemente (superenrollado negativamente). Se ha descubierto que no se necesita una restricción topológica porque RNAP genera suficiente torque que provoca un superenrollamiento incluso en una plantilla de ADN lineal. Si el ADN ya está superenrollado negativamente, esta acción relaja los superenrollamientos negativos existentes antes de provocar una acumulación de superenrollamientos positivos delante de RNAP e introduce más superenrollamientos negativos detrás de RNAP. En principio, la ADN girasa y la Topo I deberían eliminar el exceso de superenrollamientos positivos y negativos respectivamente, pero si la tasa de elongación de RNAP excede el recambio de las dos enzimas, la transcripción contribuye al nivel de superenrollamiento en estado estacionario.
Control del superenrollamiento mediante NAP
En la cromatina eucariota, el ADN rara vez está presente en la forma superenrollada libre porque los nucleosomas restringen casi todos los superenrollamientos negativos mediante la estrecha unión del ADN a las histonas. De manera similar, en E. coli, los complejos de nucleoproteínas formados por NAP restringen la mitad de la densidad de superenrollamiento del nucleoide. En otras palabras, si una NAP se disocia de un complejo de nucleoproteína, el ADN adoptaría la forma plectonémica libre. Se ha demostrado experimentalmente que la unión al ADN de HU, Fis y H-NS restringe el superenrollamiento negativo en un ADN relajado pero topológicamente restringido. Pueden hacerlo cambiando el paso helicoidal del ADN o generando contorsiones toroidales doblando y envolviendo el ADN. Alternativamente, las NAP pueden unirse preferentemente y estabilizar otras formas de ADN enrollado, como estructuras cruciformes y plectonemas ramificados. Se ha informado que Fis organiza plectonemas ramificados mediante su unión a regiones cruzadas y HU se une preferentemente a estructuras cruciformes.
Los NAP también regulan indirectamente el superenrollamiento del ADN. Fis puede modular el superenrollamiento reprimiendo la transcripción de los genes que codifican la ADN girasa. Existe evidencia genética que sugiere que HU controla los niveles de superenrollamiento al estimular la ADN girasa y reducir la actividad de Topo I. En apoyo de los estudios genéticos, se demostró que la HU estimula la decatenación del ADN catalizada por la ADN girasa in vitro. No está claro desde el punto de vista mecánico cómo HU modula las actividades de la girasa y Topo I. HU podría interactuar físicamente con la ADN girasa y Topo I o las actividades de organización del ADN de HU, como la flexión del ADN, pueden facilitar o inhibir la acción de la ADN girasa y Topo I, respectivamente.
Los superenrollamientos plectónicos se organizan en múltiples dominios topológicos
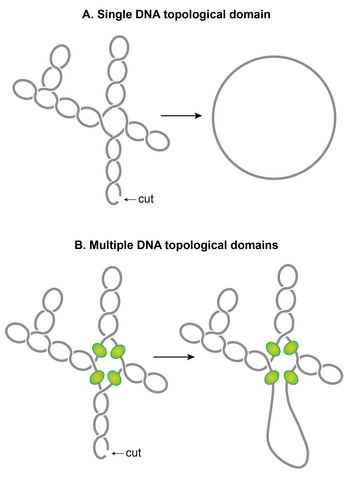
Una de las características sorprendentes del nucleoide es que los superenrollamientos plectonémicos están organizados en múltiples dominios topológicos. En otras palabras, un solo corte en un dominio sólo relajará ese dominio y no los demás. Se forma un dominio topológico debido a una barrera de difusión de superenrollamiento. Estudios independientes que emplean diferentes métodos han informado que los dominios topológicos tienen un tamaño variable que oscila entre 10 y 400 kb. La ubicación aleatoria de las barreras que se observa comúnmente en estos estudios parece explicar la amplia variabilidad en el tamaño de los dominios.
Aunque aún no se han establecido las identidades de las barreras de dominio, los posibles mecanismos responsables de la formación de las barreras incluyen: (i) Se podría formar una barrera de dominio cuando una proteína con la capacidad de restringir superenrollamientos se une simultáneamente a dos sitios distintos en el cromosoma. formando un bucle o dominio de ADN topológicamente aislado. Se ha demostrado experimentalmente que los bucles mediados por proteínas en el ADN superenrollado pueden crear un dominio topológico. Los NAP como H-NS y Fis son candidatos potenciales, según su capacidad de bucle de ADN y la distribución de sus sitios de unión. (ii) Los elementos de mosaico intercalados bacterianos (BIME) también aparecen como candidatos potenciales para barreras de dominio. Los BIME son secuencias de repeticiones palindrómicas que generalmente se encuentran entre genes. Se ha demostrado que un BIME impide la difusión del superenrollamiento en un casete topológico diseñado sintéticamente insertado en el E. coli cromosoma. Hay ~600 BIME distribuidos en todo el genoma, posiblemente dividiendo el cromosoma en 600 dominios topológicos. (iii) Las barreras también podrían resultar de la unión del ADN a la membrana celular a través de una proteína que se une tanto al ADN como a la membrana o mediante la transcripción incipiente y la traducción de proteínas ancladas a la membrana. (iv) La actividad de transcripción puede generar barreras de difusión de superenrollamiento. Se ha demostrado que una RNAP que transcribe activamente bloquea la disipación de superenrollamientos plectonémicos, formando así una barrera de difusión de superenrollamiento.
Dinámica de nucleoides dependiente de la fase de crecimiento
El nucleoide se reorganiza en las células en fase estacionaria, lo que sugiere que la estructura del nucleoide es altamente dinámica, determinada por el estado fisiológico de las células. Una comparación de mapas de contactos de alta resolución del nucleoide reveló que los contactos de largo alcance en el macrodominio Ter aumentaron en la fase estacionaria, en comparación con la fase de crecimiento. Además, los límites del CID en la fase estacionaria fueron diferentes de los encontrados en la fase de crecimiento. Finalmente, la morfología de los nucleoides sufre una transformación masiva durante la fase estacionaria prolongada; el nucleoide exhibe estructuras toroidales ordenadas.
Los cambios específicos de la fase de crecimiento en la estructura de los nucleoides podrían ser provocados por un cambio en los niveles de las proteínas arquitectónicas del ADN asociadas a los nucleoides (las subunidades NAP y Muk), el superenrollamiento y la actividad de transcripción. La abundancia de NAP y las subunidades Muk cambia según el ciclo de crecimiento bacteriano. Fis y la proteína de unión al ADN inducida por el hambre Dps, otra NAP, están presentes casi exclusivamente en la fase de crecimiento y en la fase estacionaria, respectivamente. Los niveles de Fis aumentan al entrar en la fase exponencial y luego disminuyen rápidamente mientras las células todavía están en la fase exponencial, alcanzando niveles que son indetectables en la fase estacionaria. Mientras los niveles de Fis comienzan a disminuir, los niveles de Dps comienzan a aumentar y alcanzan un máximo en la fase estacionaria. Una transición dramática en la estructura nucleoide observada en la fase estacionaria prolongada se ha atribuido principalmente a Dps. Forma conjuntos de ADN/cristalino que actúan para proteger el nucleoide de los agentes que dañan el ADN presentes durante la inanición.
HU, IHF y H-NS están presentes tanto en la fase de crecimiento como en la fase estacionaria. Sin embargo, su abundancia cambia significativamente, de modo que HU y Fis son los NAP más abundantes en la fase de crecimiento, mientras que IHF y Dps se convierten en los NAP más abundantes en la fase estacionaria. HUαα es la forma predominante en la fase exponencial temprana, mientras que la forma heterodimérica predomina en la fase estacionaria, con cantidades menores de homodímeros. Esta transición tiene consecuencias funcionales con respecto a la estructura nucleoide, porque las dos formas parecen organizar y condensar el ADN de manera diferente; Tanto los homodímeros como los heterodímeros forman filamentos, pero solo el homodímero puede unir múltiples segmentos de ADN para formar una red de ADN. El número de copias de MukB se duplica en la fase estacionaria. Un aumento en el número de moléculas de MukB podría influir en la procesividad del complejo MukBEF como factor de extrusión de bucles de ADN, lo que resultaría en un número mayor o mayor de bucles.
El superenrollamiento puede actuar de manera concertada con las proteínas arquitectónicas del ADN para reorganizar el nucleoide. El nivel general de superenrollamiento disminuye en la fase estacionaria y el superenrollamiento muestra un patrón diferente a nivel regional. Los cambios en el superenrollamiento pueden alterar la organización topológica del nucleoide. Además, debido a que una región cromosómica de alta actividad de transcripción forma un límite CID, los cambios en la actividad de transcripción durante diferentes fases de crecimiento podrían alterar la formación de los límites CID y, por tanto, la organización espacial del nucleoide. Es posible que los cambios en los límites de CID observados en la fase estacionaria se deban a la alta expresión de un conjunto diferente de genes en la fase estacionaria en comparación con la fase de crecimiento.
Estructura de nucleoides y expresión genética
NAP y expresión genética
La E. coli la estructura cromosómica y la expresión genética parecen influirse mutuamente de forma recíproca. Por un lado, una correlación de un límite CID con una alta actividad de transcripción indica que la organización cromosómica está impulsada por la transcripción. Por otro lado, la estructura tridimensional del ADN dentro del nucleoide en todas las escalas puede estar relacionada con la expresión genética. En primer lugar, se ha demostrado que la reorganización de la arquitectura 3D del nucleoide en E. coli puede modular dinámicamente el patrón de transcripción celular. Un mutante de HUa hizo que el nucleoide se condensara mucho mediante una mayor superhelicidad positiva del ADN cromosómico. En consecuencia, muchos genes fueron reprimidos y muchos genes inactivos se expresaron. Además, hay muchos casos específicos en los que los cambios arquitectónicos locales mediados por proteínas alteran la transcripción genética. Por ejemplo, la formación de filamentos rígidos de nucleoproteínas por H-NS bloquea el acceso de RNAP al promotor, impidiendo así la transcripción genética. Mediante el silenciamiento de genes, H-NS actúa como un represor global inhibiendo preferentemente la transcripción de genes transferidos horizontalmente. En otro ejemplo, la unión específica de HU en el operón gal facilita la formación de un bucle de ADN que mantiene reprimido el operón gal en ausencia del inductor. El microbucle de ADN topológicamente distinto creado por la flexión coherente del ADN por parte de Fis en promotores de ARN estables activa la transcripción. La flexión del ADN mediante IHF controla diferencialmente la transcripción de los dos promotores en tándem del operón ilvGMEDA en E. coli. Los cambios topológicos específicos de las NAP no solo regulan la transcripción genética, sino que también están involucrados en otros procesos como el inicio de la replicación, la recombinación y la transposición del ADN. A diferencia de la regulación genética específica, queda por determinar cómo la estructura cromosómica de orden superior y su dinámica influyen en la expresión genética globalmente a nivel molecular.
Superenrollamiento del ADN y expresión genética
Existe una interconexión bidireccional entre el superenrollamiento del ADN y la transcripción genética. El superenrollamiento negativo de la región promotora puede estimular la transcripción facilitando la fusión del promotor y aumentando la afinidad de unión al ADN de una proteína reguladora. Los estallidos estocásticos de transcripción parecen ser una característica general de genes altamente expresados, y los niveles de superenrollamiento de la plantilla de ADN contribuyen al estallido transcripcional. Según el modelo de dominio superenrollado gemelo, la transcripción de un gen puede influir en la transcripción de otros genes cercanos a través de un relevo superenrollado. Un ejemplo de ello es la activación del promotor leu-500. El superenrollamiento no sólo media cambios específicos de genes, sino que también media cambios a gran escala en la expresión genética. La organización topológica del nucleoide podría permitir la expresión independiente de genes sensibles al superenrollamiento en diferentes dominios topológicos. Un mapa a escala del genoma de superenrollamiento desenfrenado mostró que las regiones genómicas tienen diferentes densidades de superenrollamiento en estado estacionario, lo que indica que el nivel de superenrollamiento difiere en los dominios topológicos individuales. Como resultado, un cambio en el superenrollamiento puede dar lugar a una expresión genética de dominio específico, dependiendo del nivel de superenrollamiento en cada dominio.
El efecto del superenrollamiento sobre la expresión génica puede estar mediado por NAP que influyen directa o indirectamente en el superenrollamiento. El efecto de HU sobre la expresión genética parece implicar un cambio en el superenrollamiento y quizás una organización del ADN de orden superior. Una correlación positiva entre la unión de la ADN girasa y la regulación positiva de los genes causada por la ausencia de HU sugiere que los cambios en el superenrollamiento son responsables de la expresión diferencial. También se descubrió que HU es responsable de un efecto posicional en la expresión génica al aislar unidades transcripcionales al restringir el superenrollamiento inducido por la transcripción. Las mutaciones puntuales en HUa cambiaron drásticamente el perfil de expresión genética de E. coli, alterando su morfología, fisiología y metabolismo. Como resultado, la cepa mutante era más invasiva de las células de mamíferos. Este efecto dramático fue concomitante con la compactación de nucleoides y un aumento del superenrollamiento positivo. La proteína mutante era un octámero, a diferencia del dímero natural. Envuelve el ADN en su superficie de forma diestra, restringiendo las superenrollamientos positivas a diferencia del HU de tipo salvaje. Estos estudios muestran que las sustituciones de aminoácidos en HU pueden tener un efecto dramático en la estructura de los nucleoides, lo que a su vez resulta en cambios fenotípicos significativos.
Dado que MukB y HU se han convertido en actores críticos en las interacciones de ADN de largo alcance, valdrá la pena comparar el efecto de cada una de estas dos proteínas en la expresión genética global. Aunque HU parece controlar la expresión génica mediante la modulación de la densidad de superenrollamiento, el mecanismo molecular exacto sigue siendo desconocido y el impacto de MukB en la expresión génica aún no se ha analizado.
Organización espacial
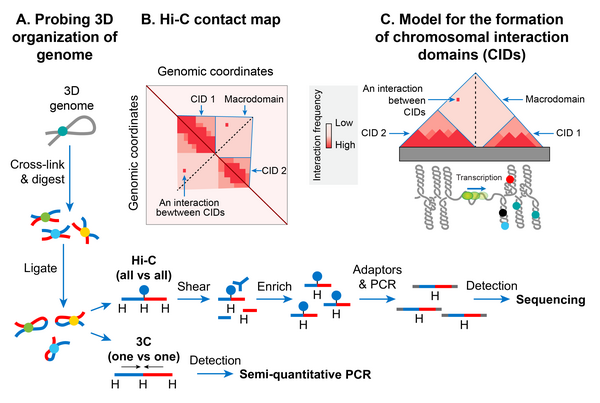
Dominios de interacción cromosómica
En los últimos años, la llegada de un método molecular llamado captura de conformación cromosómica (3C) ha permitido estudiar una organización espacial de alta resolución de los cromosomas tanto en bacterias como en eucariotas. 3C y su versión combinada con secuenciación profunda (Hi-C) determinan la proximidad física, si la hay, entre dos loci genómicos cualesquiera en el espacio 3D. Un mapa de contactos de alta resolución de cromosomas bacterianos, incluido el E. coli ha revelado que un cromosoma bacteriano está segmentado en muchas regiones altamente autointeractivas llamadas dominios de interacción cromosómica (CID). Los CID son equivalentes a los dominios de asociación topológica (TAD) observados en muchos cromosomas eucarióticos, lo que sugiere que la formación de CID es un fenómeno general de la organización del genoma. Dos características definen los CID o TAD. Primero, las regiones genómicas de un CID interactúan físicamente entre sí con más frecuencia que con las regiones genómicas fuera de ese CID o con las de un CID vecino. En segundo lugar, la presencia de un límite entre los CID que impide las interacciones físicas entre regiones genómicas de dos CID vecinos.
La E. coli se encontró que constaba de 31 CID en la fase de crecimiento. El tamaño de los CID osciló entre 40 y ~300 kb. Parece que una barrera de difusión superenrollada responsable de segregar los bucles de ADN plectonémicos en dominios topológicos funciona como un límite CID en E. coli y muchas otras bacterias. En otras palabras, la presencia de una barrera de difusión superenrollada define la formación de CID. Hallazgos del sondeo Hi-C de cromosomas en E. coli, Caulobacter crescentus y Bacillus subtilis convergen en un modelo en el que los CID se forman porque el bucle plectonémico junto con las actividades de organización del ADN de los NAP promueven interacciones físicas entre loci genómicos, y un límite CID consiste en una región libre de plectonema (PFR) que previene estas interacciones. Se crea un PFR debido a la alta actividad de transcripción porque el desenrollado helicoidal del ADN mediante la transcripción activa de RNAP restringe los superenrollamientos plectonémicos. Como resultado, la disipación de los superenrollamientos también se bloquea, creando una barrera de difusión de superenrollamiento. La evidencia indirecta de este modelo proviene de la observación de que las CID de los cromosomas bacterianos, incluido el E. coli muestra genes altamente transcritos en sus límites, lo que indica un papel de la transcripción en la formación de un límite CID. Una evidencia más directa provino del hallazgo de que la colocación de un gen altamente transcrito en una posición donde no había ningún límite creó un nuevo límite CID en el C. cromosoma crescentus. Sin embargo, no todos los límites CID se correlacionaron con genes altamente transcritos en E. coli, lo que sugiere que otros factores desconocidos también son responsables de la formación de límites CID y barreras de difusión superenrolladas.
Macrodomains
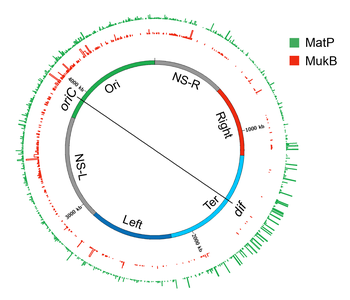
Los bucles de ADN plectonémico organizados como dominios topológicos o CID parecen fusionarse aún más para formar grandes dominios espacialmente distintos llamados macrodominios (MD). En E. coli, las MD se identificaron inicialmente como grandes segmentos del genoma cuyos marcadores de ADN se localizaban juntos (co-localizados) en estudios de hibridación fluorescente in situ (FISH). Una gran región genómica (~1 Mb) que cubre el locus oriC (origen de replicación cromosómica) se co-localizó y se denominó macrodominio Ori. Del mismo modo, una gran región genómica (~1 Mb) que cubre la región terminal de replicación (ter) se co-localizó y se denominó macrodominio Ter. Posteriormente, los MD se identificaron basándose en la frecuencia con la que los pares de sitios lambda att que se insertaban en varias ubicaciones distantes del cromosoma se recombinaban entre sí. En este método basado en recombinación, una MD se definió como una gran región genómica cuyos sitios de ADN pueden recombinarse principalmente entre sí, pero no con aquellos fuera de esa MD. El método basado en recombinación confirmó los MD de Ori y Ter que se identificaron en los estudios FISH e identificó dos MD adicionales.
Los dos MD adicionales estaban formados por las regiones adicionales de ~1 Mb que flanqueaban el Ter y se denominaron Izquierda y Derecha. Estos cuatro MD (Ori, Ter, Izquierda y Derecha) componían la mayor parte del genoma, excepto dos regiones genómicas que flanqueaban a Ori. Estas dos regiones (NS-L y NS-R) eran más flexibles y no estructuradas en comparación con un MD, ya que los sitios de ADN en ellas se recombinaban con sitios de ADN ubicados en MD en ambos lados. La posición genética de oriC parece dictar la formación de los MD, porque el reposicionamiento de oriC mediante manipulación genética da como resultado la reorganización de los MD. Por ejemplo, las regiones genómicas más cercanas al oriC siempre se comportan como NS independientemente de la secuencia de ADN y las regiones más alejadas siempre se comportan como MD.
La técnica Hi-C confirmó además una organización espacial jerárquica de los CID en forma de macrodominios. En otras palabras, los CID de un macrodominio interactuaban físicamente entre sí con más frecuencia que con los CID de un macrodominio vecino o con loci genómicos fuera de ese macrodominio. Los datos de Hi-C mostraron que el E. coli se estaba dividiendo en dos dominios distintos. La región que rodea ter formó un dominio aislado que se superponía con el Ter MD previamente identificado. Los contactos ADN-ADN en este dominio se produjeron sólo en el rango de hasta ~280 kb. El resto del cromosoma formó un dominio único cuyos loci genómicos exhibieron contactos en el rango de >280 kb. Si bien la mayoría de los contactos en este dominio estaban restringidos a una distancia máxima de ~500 kb, hubo dos regiones sueltas cuyos loci genómicos formaron contactos a distancias aún mayores (hasta ~1 Mb). Estas regiones sueltas correspondían a las regiones flexibles y menos estructuradas (NS) previamente identificadas. Los límites del dominio aislado que abarca ter y las dos regiones sueltas identificadas mediante el método Hi-C segmentaron todo el cromosoma en seis regiones que se corresponden con las cuatro regiones MD y dos regiones NS definidas mediante ensayos basados en recombinación..
Proteínas que impulsan la formación de macrodominios
MatP
Una búsqueda de proteínas responsables de la formación de macrodominios condujo a la identificación de la proteína Macrodominio Ter (MatP). MatP se une casi exclusivamente en Ter MD reconociendo un motivo de 13 pb llamado secuencia de macrodominio ter (matS). Hay 23 sitios matS presentes en el dominio Ter, en promedio hay un sitio cada 35 kb. Más evidencia de la unión de MatP en el dominio Ter proviene de imágenes de fluorescencia de MatP. Se observaron focos discretos de MatP que se co-localizaron con marcadores de ADN del dominio Ter. Un fuerte enriquecimiento de la señal ChIP-Seq en Ter MD también corrobora la unión preferencial de MatP a este dominio.
MatP condensa ADN en el dominio Ter porque la falta de MatP aumentó la distancia entre dos marcadores de ADN fluorescentes ubicados a 100 kb de distancia en el dominio Ter. Además, MatP es un actor fundamental a la hora de aislar el dominio Ter del resto del cromosoma. Promueve los contactos ADN-ADN dentro del dominio Ter pero previene los contactos entre los loci de ADN del dominio Ter y los de las regiones flanqueantes. ¿Cómo condensa MatP el ADN y promueve los contactos ADN-ADN? Los resultados experimentales son contradictorios. MatP puede formar un bucle de ADN entre dos sitios matS in vitro y su actividad de bucle de ADN depende de la tetramerización de MatP. La tetramerización se produce mediante interacciones en espiral entre dos moléculas de MatP unidas al ADN. Un modelo obvio basado en resultados in vitro es que MatP promueve los contactos ADN-ADN in vivo al unir sitios matS. Sin embargo, aunque MatP conectó sitios distantes en los estudios Hi-C, no conectó específicamente los sitios matS. Además, un mutante de MatP que no pudo formar tetrámeros se comportó como un tipo salvaje. Estos resultados van en contra del modelo puente matS para la organización Ter, dejando el mecanismo de acción de MatP difícil de alcanzar. Una posibilidad es que MatP se propague a segmentos de ADN cercanos desde su sitio de unión primario matS y une sitios distantes a través de un mecanismo que no depende de la tetramerización.
MukBEF
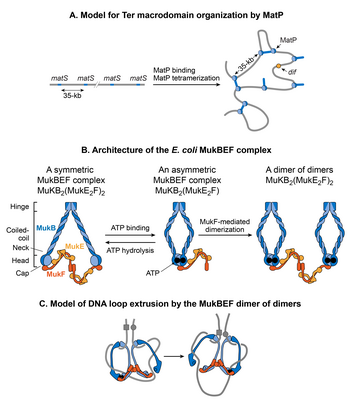
MukB pertenece a una familia de ATPasas llamadas proteínas de mantenimiento estructural de los cromosomas (SMC), que participan en la organización cromosómica de orden superior en eucariotas. Dos monómeros MukB se asocian mediante una interacción continua y antiparalela formando una varilla rígida de 100 nm de largo. En el centro de la varilla se produce una región de bisagra flexible. Debido a la flexibilidad de la región de la bisagra, MukB adopta una forma de V característica de la familia SMC. Las subunidades no SMC que se asocian con MukB son MukE y MukF. La asociación cierra la formación V, dando como resultado grandes estructuras en forma de anillos. MukE y MukF están codificados junto con MukB en el mismo operón en E. coli. La eliminación de cualquiera de las subunidades da como resultado el mismo fenotipo, lo que sugiere que el complejo MukBEF es la unidad funcional in vivo. Las actividades de unión al ADN del complejo residen en la subunidad MukB, mientras que MukE y MukF modulan la actividad de MukB.
El complejo MukBEF, junto con Topo IV, es necesario para la decatenación y reposicionamiento de oriC recién replicados. El papel de MukBEF no está restringido durante la replicación del ADN. Organiza y condensa el ADN incluso en células que no se replican. El reciente mapa de conformación cromosómica de alta resolución de E. coli revela que MukB participa en la formación de interacciones ADN-ADN en todo el cromosoma, excepto en el dominio Ter. ¿Cómo se impide que MukB actúe en el dominio Ter? MatP interactúa físicamente con MukB, evitando así que MukB se localice en el dominio Ter. Esto es evidente en la unión al ADN de MatP y MukB en el dominio Ter. La unión al ADN de MatP se enriquece en el dominio Ter, mientras que la unión al ADN de MukB se reduce en comparación con el resto del genoma. Además, en una cepa que ya carece de MatP, la ausencia de MukB provoca una reducción de los contactos del ADN en todo el cromosoma, incluido el dominio Ter. Este resultado concuerda con la opinión de que MatP desplaza a MukB del dominio Ter.
¿Cómo funciona el complejo MukBEF para organizar el E. coli cromosoma? Según la opinión actual, los complejos SMC organizan los cromosomas mediante la extrusión de bucles de ADN. Los complejos SMC se translocan a lo largo del ADN para extruir bucles de manera cis (en la misma molécula de ADN), donde el tamaño de los bucles depende de la procesividad del complejo. Los complejos SMC de diferentes organismos difieren en el mecanismo de extrusión del bucle. Microscopía de fluorescencia de molécula única de MukBEF en E. coli sugiere que la unidad funcional mínima in vivo es un dímero de dímeros. Esta unidad se forma mediante la unión de dos complejos MukBEF unidos a ATP mediante dimerización mediada por MukF. MukBEF se localiza en la célula como 1-3 grupos que se alargan paralelos al eje longitudinal de la célula. Cada grupo contiene un promedio de ~ 8-10 dímeros de dímeros. Según el modelo actual, MukBEF extruye bucles de ADN a modo de “escalada en roca”. Un dímero de dímeros libera un segmento de ADN y captura un nuevo segmento de ADN sin disociarse del cromosoma. Además del bucle de ADN, un vínculo entre el superenrollamiento negativo y la función MukBEF in vivo junto con la capacidad de la subunidad MukB para restringir los superenrollamientos negativos in vitro sugiere que MukBEF organiza el ADN generando superenrollamientos..
Papel de los NAP y naRNA
Además de contribuir a la compactación de los cromosomas al doblar, unir y formar bucles en el ADN a una escala más pequeña (~1 kb), los NAP participan en la condensación y organización del ADN al promover contactos de largo alcance entre ADN y ADN. Dos NAP, Fis y HU, surgieron como actores clave en la promoción de contactos ADN-ADN de largo alcance que ocurren en todo el cromosoma. Queda por estudiar cómo las actividades de organización del ADN de Fis y HU, que se comprenden bien a una escala más pequeña (~ 1 kb), dan como resultado la formación de interacciones ADN-ADN de largo alcance. No obstante, algunas de las interacciones de ADN mediadas por HU requieren la presencia de naRNA4. naRNA4 también participa en la realización de contactos de ADN de largo alcance. HU cataliza algunos de los contactos, no todos, lo que sugiere que el ARN participa con otros NAP en la formación de contactos de ADN. HU también parece actuar junto con MukB para promover interacciones ADN-ADN de largo alcance. Esta opinión se basa en observaciones de que la ausencia de HU o MukB provocó una reducción en los mismos contactos ADN-ADN. No está claro cómo MukB y HU actúan potencialmente juntos para promover las interacciones ADN-ADN. Es posible que las dos proteínas interactúen físicamente. Alternativamente, mientras MukBEF extruye grandes bucles de ADN, HU condensa y organiza esos bucles.
Papel de la relación funcional de los genes
Hay informes de que genes relacionados funcionalmente de E. coli están físicamente juntos en un espacio tridimensional dentro del cromosoma a pesar de que están muy separados por la distancia genética. La proximidad espacial de genes funcionalmente relacionados no sólo hace que las funciones biológicas sean más compartimentadas y eficientes, sino que también contribuiría al plegamiento y la organización espacial del nucleoide. Un estudio reciente que utilizó marcadores fluorescentes para la detección de loci de ADN específicos examinó las distancias físicas por pares entre los siete operones de ARNr que están genéticamente separados entre sí (hasta por dos millones de pb). Informó que todos los operones, excepto rrnC, estaban físicamente cerca. Sorprendentemente, los estudios 3C-seq no revelaron la agrupación física de los operones rrn, lo que contradice los resultados del estudio basado en fluorescencia. Por lo tanto, se requiere más investigación para resolver estas observaciones contradictorias. En otro ejemplo, GalR forma una red de interacción de sitios de unión de GalR que están dispersos por todo el cromosoma. GalR es un regulador transcripcional del regulón de galactosa compuesto por genes que codifican enzimas para el transporte y metabolismo del azúcar D-galactosa. GalR existe sólo en uno o dos focos en las células y puede autoensamblarse en grandes estructuras ordenadas. Por lo tanto, parece que GalR unido al ADN se multimeriza para formar interacciones a larga distancia.
Forma y estructura global
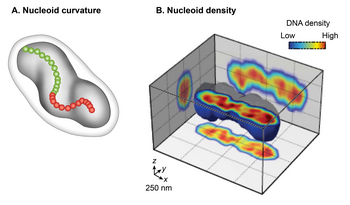
Microscopía electrónica de transmisión convencional (TEM) de E fijada químicamente. coli representaron el nucleoide como un orgánulo de forma irregular. Sin embargo, las imágenes de fluorescencia de campo amplio de nucleoides vivos en 3D revelaron una forma elipsoide discreta. La superposición de una imagen de contraste de fases de la célula y la imagen fluorescente del nucleoide mostró una yuxtaposición cercana sólo en la dimensión radial a lo largo de toda la longitud del nucleoide hasta la periferia de la célula. Este hallazgo indica confinamiento radial del nucleoide. Un examen detallado de la imagen de fluorescencia 3D después de un corte transversal perpendicular a su eje mayor reveló además dos características globales del nucleoide: curvatura y regiones longitudinales de alta densidad. Examinar la quiralidad de la línea central del nucleoide conectando el centro de intensidad de cada sección transversal mostró que la forma general del nucleoide es curva. La distribución de la intensidad de la fluorescencia en las secciones transversales reveló una subestructura de densidad, que consta de regiones o haces curvados de alta densidad en el núcleo central y regiones de baja densidad en la periferia. Una implicación del confinamiento radial es que determina la forma curva del nucleoide. Según un modelo, el nucleoide se ve obligado a doblarse porque está confinado en una E cilíndrica. coli cuyo radio es menor que su longitud flexible (longitud de persistencia). Este modelo fue respaldado por observaciones de que la eliminación de la pared celular o la inhibición de su síntesis aumentaba el radio de la célula y daba como resultado un aumento concomitante del radio helicoidal y una disminución del paso helicoidal en el nucleoide.
Conexiones nucleoide-membrana
Una fuerza de expansión debida a las conexiones de la membrana del ADN parece funcionar en oposición a las fuerzas de condensación para mantener un nivel óptimo de condensación del nucleoide. Los estudios de fraccionamiento celular y microscopía electrónica indicaron por primera vez la posibilidad de conexiones entre el ADN y la membrana. En la actualidad existen varios ejemplos conocidos de conexiones de membrana de ADN. La transerción es un mecanismo de transcripción, traducción e inserción concurrentes de proteínas de membrana nacientes que forman contactos transitorios entre el ADN y la membrana. Se ha demostrado que la transición de dos proteínas de membrana, LacY y TetA, provoca el reposicionamiento de loci cromosómicos hacia la membrana. Otro mecanismo de conexiones nucleoide-membrana es a través de un contacto directo entre los reguladores de la transcripción anclados a la membrana y sus sitios objetivo en el cromosoma. Un ejemplo de regulador de la transcripción en E. coli es CadC. CadC contiene un dominio sensorial periplásmico y un dominio de unión al ADN citoplasmático. La detección de un ambiente ácido por su dominio sensorial periplásmico estimula la actividad de unión al ADN de CadC, que luego activa la transcripción de sus genes diana. La localización en la membrana de genes regulados por un regulador de la transcripción anclado a la membrana aún no se ha demostrado. No obstante, se espera que la activación de genes diana en el cromosoma por parte de estos reguladores dé como resultado un contacto nucleoide-membrana, aunque sería un contacto dinámico. Además de estos ejemplos, el cromosoma también está anclado específicamente a la membrana celular a través de la interacción proteína-proteína entre proteínas unidas al ADN, por ejemplo, SlmA y MatP, y el divisoma. Dado que los genes que codifican las proteínas de la membrana se distribuyen por todo el genoma, los contactos dinámicos entre el ADN y la membrana a través de la transerción pueden actuar como una fuerza de expansión de nucleoides. Esta fuerza de expansión funcionaría en oposición a las fuerzas de condensación para mantener un nivel de condensación óptimo. La formación de nucleoides altamente condensados tras la exposición de E. coli al cloranfenicol, que bloquea la traducción, proporciona apoyo a la fuerza de expansión de los contactos transitorios entre el ADN y la membrana formados mediante la transerción. La forma redonda de los nucleoides excesivamente condensados después del tratamiento con cloranfenicol también sugiere un papel de los contactos de la membrana del ADN mediados por transerción en la definición de la forma elipsoide del nucleoide.
Visualización
El nucleoide se puede visualizar claramente en una micrografía electrónica con un aumento muy alto, donde, aunque su apariencia puede diferir, es claramente visible contra el citosol. A veces incluso son visibles hebras de lo que se cree que es ADN. Al teñir con la tinción de Feulgen, que tiñe específicamente el ADN, el nucleoide también se puede ver bajo un microscopio óptico. Las tinciones intercalantes de ADN DAPI y bromuro de etidio se utilizan ampliamente para la microscopía de fluorescencia de nucleoides. Tiene una forma irregular y se encuentra en las células procarióticas.
Daño y reparación del ADN
Se observan cambios en la estructura del nucleoide de bacterias y arqueas después de la exposición a condiciones que dañan el ADN. Los nucleoides de las bacterias Bacillus subtilis y Escherichia coli se vuelven significativamente más compactos después de la irradiación UV. Formación de la estructura compacta en E. coli requiere la activación de RecA a través de interacciones específicas entre RecA y ADN. La proteína RecA desempeña un papel clave en la reparación recombinante homóloga del daño del ADN.
Similar a B. subtilis y E. coli arriba, las exposiciones de la arqueona Haloferax volcanii a tensiones que dañan el ADN provocan la compactación y la reorganización del nucleoide. La compactación depende del complejo proteico Mre11-Rad50 que cataliza un paso temprano en la reparación recombinacional homóloga de roturas de doble hebra en el ADN. Se ha propuesto que la compactación de nucleoides es parte de una respuesta al daño del ADN que acelera la recuperación celular al ayudar a las proteínas reparadoras del ADN a localizar objetivos y al facilitar la búsqueda de secuencias de ADN intactas durante la recombinación homóloga.
Contenido relacionado
Ley de Fick
Híbrido (biología)
Evolución divergente