La expresion genica
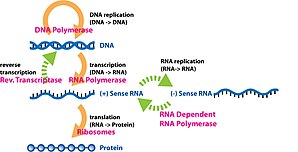
Expresión génica es el proceso mediante el cual la información de un gen se utiliza en la síntesis de un producto génico funcional que le permite producir productos finales, proteínas o ARN no codificante, y finalmente afectar a un fenotipo, como efecto final. Estos productos suelen ser proteínas, pero en genes que no codifican proteínas, como el ARN de transferencia (ARNt) y el ARN nuclear pequeño (ARNsn), el producto es un ARN no codificante funcional. La expresión génica se resume en el dogma central de la biología molecular formulado por primera vez por Francis Crick en 1958, más desarrollado en su artículo de 1970 y ampliado por los descubrimientos posteriores de la transcripción inversa y la replicación del ARN.
Todas las formas de vida conocidas (eucariotas (incluidos los organismos multicelulares), procariotas (bacterias y arqueas) y virus) utilizan el proceso de expresión génica para generar la maquinaria macromolecular necesaria para la vida.
En genética, la expresión génica es el nivel más fundamental en el que el genotipo da origen al fenotipo, es decir, rasgo observable. La información genética almacenada en el ADN representa el genotipo, mientras que el fenotipo resulta de la "interpretación" de esa información. Dichos fenotipos a menudo se muestran mediante la síntesis de proteínas que controlan la estructura y el desarrollo del organismo, o que actúan como enzimas que catalizan rutas metabólicas específicas.
Todos los pasos del proceso de expresión génica se pueden modular (regular), incluida la transcripción, el corte y empalme del ARN, la traducción y la modificación postraduccional de una proteína. La regulación de la expresión génica brinda control sobre el tiempo, la ubicación y la cantidad de un producto génico dado (proteína o ncRNA) presente en una célula y puede tener un efecto profundo en la estructura y función celular. La regulación de la expresión génica es la base para la diferenciación celular, el desarrollo, la morfogénesis y la versatilidad y adaptabilidad de cualquier organismo. Por lo tanto, la regulación génica puede servir como sustrato para el cambio evolutivo.
Mecanismo
Transcripción
La producción de una copia de ARN a partir de una cadena de ADN se denomina transcripción y la realizan las ARN polimerasas, que agregan un ribonucleótido a la vez a una cadena de ARN en crecimiento según la ley de complementariedad de las bases de nucleótidos. Este ARN es complementario a la cadena de ADN molde 3′ → 5′, con la excepción de que las timinas (T) se reemplazan con uracilos (U) en el ARN y posibles errores.
En las bacterias, la transcripción la lleva a cabo un solo tipo de polimerasa de ARN, que necesita unirse a una secuencia de ADN llamada caja de Pribnow con la ayuda de la proteína del factor sigma (factor σ) para comenzar la transcripción. En los eucariotas, la transcripción se realiza en el núcleo por tres tipos de polimerasas de ARN, cada una de las cuales necesita una secuencia de ADN especial llamada promotor y un conjunto de proteínas de unión al ADN (factores de transcripción) para iniciar el proceso (consulte la regulación de la transcripción a continuación). La ARN polimerasa I es responsable de la transcripción de los genes del ARN ribosómico (ARNr). La ARN polimerasa II (Pol II) transcribe todos los genes que codifican proteínas, pero también algunos ARN no codificantes (por ejemplo,, snRNA, snoRNA o ARN no codificantes largos). La ARN polimerasa III transcribe el ARNr 5S, los genes del ARN de transferencia (ARNt) y algunos ARN pequeños no codificantes (p. ej., 7SK). La transcripción termina cuando la polimerasa encuentra una secuencia llamada terminador.
Procesamiento de ARNm
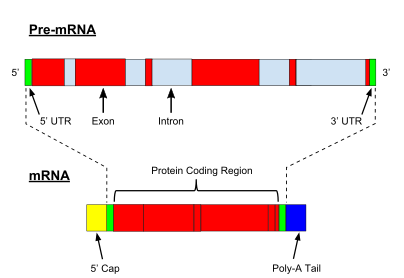
Mientras que la transcripción de genes codificantes de proteínas procarióticas crea ARN mensajero (ARNm) que está listo para traducirse en proteína, la transcripción de genes eucarióticos deja una transcripción primaria de ARN (pre-ARN), que primero tiene que sufrir una serie de modificaciones convertirse en un ARN maduro. Los tipos y pasos involucrados en los procesos de maduración varían entre preARN codificantes y no codificantes; es decir, aunque las moléculas de preARN tanto para el ARNm como para el ARNt se someten a corte y empalme, los pasos y la maquinaria involucrada son diferentes. El procesamiento del ARN no codificante se describe a continuación (maduración del ARN no codificante).
El procesamiento de pre-ARNm incluye el tapado de 5′, que es un conjunto de reacciones enzimáticas que agregan 7-metilguanosina (m7G) al extremo 5′ de pre-ARNm y así proteger el ARN de la degradación por exonucleasas. El casquete de m7G se une luego al heterodímero complejo de unión al casquete (CBC20/CBC80), que ayuda en la exportación del ARNm al citoplasma y también protege al ARN de la decapitación.
Otra modificación es la escisión y poliadenilación en 3′. Ocurren si la secuencia señal de poliadenilación (5′-AAUAAA-3′) está presente en el pre-ARNm, que generalmente se encuentra entre la secuencia codificadora de proteínas y el terminador. El pre-ARNm primero se escinde y luego se agrega una serie de ~200 adeninas (A) para formar una cola de poli(A), que protege al ARN de la degradación. La cola de poli(A) está unida por múltiples proteínas de unión a poli(A) (PABP) necesarias para la exportación de ARNm y el reinicio de la traducción. En el proceso inverso de deadenilación, las colas poli(A) se acortan por la exonucleasa CCR4-Not 3′-5′, que a menudo conduce a la descomposición completa de la transcripción.
Una modificación muy importante del pre-ARNm eucariótico es el empalme de ARN. La mayoría de los pre-ARNm eucariotas consisten en segmentos alternados llamados exones e intrones. Durante el proceso de empalme, un complejo catalítico de proteína de ARN conocido como spliceosome cataliza dos reacciones de transesterificación, que eliminan un intrón y lo liberan en forma de estructura lariat, y luego empalman los exones vecinos. En ciertos casos, algunos intrones o exones pueden eliminarse o retenerse en el ARNm maduro. Este llamado empalme alternativo crea una serie de diferentes transcritos que se originan en un solo gen. Debido a que estas transcripciones pueden traducirse potencialmente en diferentes proteínas, el empalme amplía la complejidad de la expresión génica eucariótica y el tamaño del proteoma de una especie.
El procesamiento extensivo del ARN puede ser una ventaja evolutiva posible gracias al núcleo de los eucariotas. En los procariotas, la transcripción y la traducción ocurren juntas, mientras que en los eucariotas, la membrana nuclear separa los dos procesos, dando tiempo para que ocurra el procesamiento del ARN.
Maduración del ARN no codificante
En la mayoría de los organismos, los genes no codificantes (ncRNA) se transcriben como precursores que se someten a un procesamiento posterior. En el caso de los ARN ribosómicos (ARNr), a menudo se transcriben como un pre-ARNr que contiene uno o más ARNr. El pre-ARNr se escinde y modifica (2′-O-metilación y formación de pseudouridina) en sitios específicos por aproximadamente 150 especies diferentes de ARN pequeño restringido al nucléolo, llamadas snoARN. Los SnoRNA se asocian con proteínas, formando snoRNP. Mientras que la parte del snoRNA se empareja con el ARN objetivo y, por lo tanto, coloca la modificación en un sitio preciso, la parte de la proteína realiza la reacción catalítica. En eucariotas, en particular una snoRNP llamada RNasa, MRP escinde el pre-rRNA 45S en los rRNA 28S, 5.8S y 18S. Los factores de procesamiento de ARNr y ARN forman grandes agregados llamados nucléolos.
En el caso del ARN de transferencia (ARNt), por ejemplo, la RNasa P elimina la secuencia 5′, mientras que la enzima tRNasa Z elimina el extremo 3′ y la cola CCA 3′ sin plantilla se agrega mediante una nucleotidil transferasa. En el caso del micro ARN (miARN), los miARN se transcriben primero como transcritos primarios o pri-miARN con una tapa y una cola de poli-A y se procesan en estructuras cortas de bucle de tallo de 70 nucleótidos conocidas como pre-miARN en el núcleo celular. por las enzimas Drosha y Pasha. Después de exportarse, se procesa para madurar miARN en el citoplasma mediante la interacción con la endonucleasa Dicer, que también inicia la formación del complejo de silenciamiento inducido por ARN (RISC), compuesto por la proteína Argonaute.
Incluso los snRNA y los snoRNA se someten a una serie de modificaciones antes de convertirse en parte del complejo RNP funcional. Esto se hace en el nucleoplasma o en los compartimentos especializados llamados cuerpos de Cajal. Sus bases están metiladas o pseudouridiniladas por un grupo de pequeños ARN específicos del cuerpo de Cajal (scaRNA), que son estructuralmente similares a los snoRNA.
Exportación de ARN
En los eucariotas, la mayor parte del ARN maduro debe exportarse al citoplasma desde el núcleo. Mientras que algunos ARN funcionan en el núcleo, muchos ARN se transportan a través de los poros nucleares hacia el citosol. La exportación de ARN requiere asociación con proteínas específicas conocidas como exportinas. Moléculas de exportina específicas son responsables de la exportación de un tipo de ARN determinado. El transporte de ARNm también requiere la asociación correcta con Exon Junction Complex (EJC), lo que garantiza que se complete el procesamiento correcto del ARNm antes de la exportación. En algunos casos, los ARN se transportan adicionalmente a una parte específica del citoplasma, como una sinapsis; luego son remolcados por proteínas motoras que se unen a través de proteínas conectoras a secuencias específicas (llamadas "códigos postales") en el ARN.
Traducción
Para algunos ARN (ARN no codificante), el ARN maduro es el producto génico final. En el caso del ARN mensajero (ARNm), el ARN es un portador de información que codifica para la síntesis de una o más proteínas. El ARNm que lleva una sola secuencia de proteína (común en eucariotas) es monocistrónico, mientras que el ARNm que lleva múltiples secuencias de proteína (común en procariotas) se conoce como policistrónico.
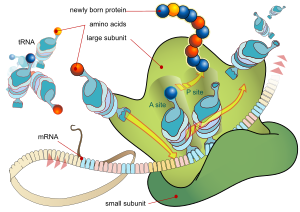
Todo ARNm consta de tres partes: una región no traducida en 5' (5'UTR), una región codificante de proteínas o marco de lectura abierto (ORF) y una región no traducida en 3' (3'UTR). La región codificante transporta información para la síntesis de proteínas codificada por el código genético para formar tripletes. Cada triplete de nucleótidos de la región codificante se denomina codón y corresponde a un sitio de unión complementario a un triplete de anticodón en el ARN de transferencia. Los ARN de transferencia con la misma secuencia de anticodón siempre llevan un tipo idéntico de aminoácido. A continuación, el ribosoma encadena los aminoácidos según el orden de los tripletes en la región codificante. El ribosoma ayuda al ARN de transferencia a unirse al ARN mensajero y toma el aminoácido de cada ARN de transferencia y lo convierte en una proteína sin estructura. Cada molécula de ARNm se traduce en muchas moléculas de proteína, en promedio ~2800 en mamíferos.
En los procariotas, la traducción generalmente ocurre en el punto de transcripción (co-transcripcionalmente), a menudo utilizando un ARN mensajero que aún está en proceso de creación. En eucariotas, la traducción puede ocurrir en una variedad de regiones de la célula dependiendo de dónde se supone que está la proteína que se escribe. Las ubicaciones principales son el citoplasma para las proteínas citoplasmáticas solubles y la membrana del retículo endoplásmico para las proteínas que se exportan desde la célula o se insertan en una membrana celular. Las proteínas que se supone que se producen en el retículo endoplásmico se reconocen a mitad del proceso de traducción. Esto se rige por la partícula de reconocimiento de señales, una proteína que se une al ribosoma y lo dirige al retículo endoplásmico cuando encuentra un péptido señal en la cadena de aminoácidos en crecimiento (naciente).
Plegado
Cada proteína existe como un polipéptido desplegado o una espiral aleatoria cuando se traduce de una secuencia de ARNm a una cadena lineal de aminoácidos. Este polipéptido carece de cualquier estructura tridimensional desarrollada (el lado izquierdo de la figura vecina). Luego, el polipéptido se pliega en su estructura tridimensional característica y funcional a partir de una espiral aleatoria. Los aminoácidos interactúan entre sí para producir una estructura tridimensional bien definida, la proteína plegada (el lado derecho de la figura) conocida como estado nativo. La estructura tridimensional resultante está determinada por la secuencia de aminoácidos (dogma de Anfinsen).
La estructura tridimensional correcta es esencial para funcionar, aunque algunas partes de las proteínas funcionales pueden permanecer desplegadas. El hecho de no plegarse en la forma deseada generalmente produce proteínas inactivas con diferentes propiedades, incluidos los priones tóxicos. Se cree que varias enfermedades neurodegenerativas y de otro tipo resultan de la acumulación de proteínas mal plegadas. Muchas alergias son causadas por el plegamiento de las proteínas, ya que el sistema inmunitario no produce anticuerpos para ciertas estructuras proteicas.
Las enzimas llamadas chaperonas ayudan a la proteína recién formada a alcanzar (plegarse en) la estructura tridimensional que necesita para funcionar. De manera similar, las chaperonas de ARN ayudan a los ARN a alcanzar sus formas funcionales. Ayudar al plegamiento de proteínas es una de las principales funciones del retículo endoplásmico en eucariotas.
Translocación
Las proteínas secretoras de eucariotas o procariotas deben translocarse para entrar en la vía secretora. Las proteínas recién sintetizadas se dirigen al canal de translocación eucariota Sec61 o procariota SecYEG mediante péptidos señal. La eficacia de la secreción de proteínas en eucariotas depende mucho del péptido señal que se haya utilizado.
Transporte de proteínas
Muchas proteínas están destinadas a otras partes de la célula además del citosol y se utiliza una amplia gama de secuencias de señalización o (péptidos de señalización) para dirigir las proteínas a donde se supone que deben estar. En procariotas, este es normalmente un proceso simple debido a la limitada compartimentación de la célula. Sin embargo, en los eucariotas existe una gran variedad de procesos de direccionamiento diferentes para garantizar que la proteína llegue al orgánulo correcto.
No todas las proteínas permanecen dentro de la célula y muchas se exportan, por ejemplo, enzimas digestivas, hormonas y proteínas de la matriz extracelular. En eucariotas, la ruta de exportación está bien desarrollada y el mecanismo principal para la exportación de estas proteínas es la translocación al retículo endoplásmico, seguido del transporte a través del aparato de Golgi.
Regulación de la expresión génica
La regulación de la expresión génica es el control de la cantidad y el momento de aparición del producto funcional de un gen. El control de la expresión es vital para permitir que una célula produzca los productos genéticos que necesita cuando los necesita; a su vez, esto le da a las células la flexibilidad para adaptarse a un entorno variable, señales externas, daño a la célula y otros estímulos. De manera más general, la regulación génica otorga a la célula el control sobre toda la estructura y función, y es la base para la diferenciación celular, la morfogénesis y la versatilidad y adaptabilidad de cualquier organismo.
Se utilizan numerosos términos para describir tipos de genes dependiendo de cómo estén regulados; éstas incluyen:
- A gen constitutivo es un gen que se transcribe continuamente en lugar de un gen facultativo, que sólo se transcribe cuando es necesario.
- A gen de limpieza es un gen que se requiere para mantener la función celular básica y así se expresa típicamente en todos los tipos de células de un organismo. Ejemplos incluyen actina, GAPDH y ubiquitina. Algunos genes de limpieza se transcriben a un ritmo relativamente constante y estos genes se pueden utilizar como punto de referencia en experimentos para medir las tasas de expresión de otros genes.
- A gen facultativo es un gen sólo transcribido cuando es necesario en lugar de un gen constitutivo.
- An gen inducible es un gen cuya expresión responde al cambio ambiental o depende de la posición del ciclo celular.
Se puede modular cualquier paso de la expresión génica, desde el paso de transcripción de ADN-ARN hasta la modificación postraduccional de una proteína. La estabilidad del producto génico final, ya sea ARN o proteína, también contribuye al nivel de expresión del gen: un producto inestable da como resultado un nivel de expresión bajo. En general, la expresión génica se regula a través de cambios en el número y tipo de interacciones entre moléculas que colectivamente influyen en la transcripción de ADN y la traducción de ARN.
Algunos ejemplos simples de dónde la expresión génica es importante son:
- Control de la expresión de la insulina por lo que da una señal para la regulación de la glucosa en sangre.
- Inactivación de cromosoma X en mamíferos femeninos para prevenir una "sobredosis" de los genes que contiene.
- Los niveles de expresión Cyclin controlan la progresión a través del ciclo celular eucarístico.
Regulación transcripcional
La regulación de la transcripción se puede dividir en tres rutas principales de influencia; genética (interacción directa de un factor de control con el gen), interacción de modulación de un factor de control con la maquinaria de transcripción y epigenética (cambios no secuenciales en la estructura del ADN que influyen en la transcripción).
La interacción directa con el ADN es el método más simple y directo mediante el cual una proteína cambia los niveles de transcripción. Los genes a menudo tienen varios sitios de unión a proteínas alrededor de la región codificante con la función específica de regular la transcripción. Hay muchas clases de sitios reguladores de unión al ADN conocidos como potenciadores, aislantes y silenciadores. Los mecanismos para regular la transcripción son variados, desde bloquear sitios de unión clave en el ADN para la ARN polimerasa hasta actuar como activador y promover la transcripción ayudando a la unión de la ARN polimerasa.
La actividad de los factores de transcripción se modula aún más mediante señales intracelulares que provocan la modificación postraduccional de las proteínas, incluida la fosforilación, la acetilación o la glicosilación. Estos cambios influyen en la capacidad de un factor de transcripción para unirse, directa o indirectamente, al ADN promotor, reclutar ARN polimerasa o favorecer la elongación de una molécula de ARN recién sintetizada.
La membrana nuclear en eucariotas permite una mayor regulación de los factores de transcripción por la duración de su presencia en el núcleo, que está regulada por cambios reversibles en su estructura y por la unión de otras proteínas. Los estímulos ambientales o las señales endocrinas pueden provocar la modificación de las proteínas reguladoras provocando cascadas de señales intracelulares, que dan como resultado la regulación de la expresión génica.
Se ha hecho evidente que existe una influencia significativa de los efectos específicos de secuencias no ADN en la transcripción. Estos efectos se denominan epigenéticos e implican la estructura de orden superior del ADN, las proteínas de unión al ADN no específicas de secuencia y la modificación química del ADN. En general, los efectos epigenéticos alteran la accesibilidad del ADN a las proteínas y así modulan la transcripción.
En eucariotas, la estructura de la cromatina, controlada por el código de histonas, regula el acceso al ADN con un impacto significativo en la expresión de genes en las áreas de eucromatina y heterocromatina.
Potenciadores, factores de transcripción, complejo mediador y bucles de ADN en la transcripción de mamíferos

La expresión génica en los mamíferos está regulada por muchos elementos reguladores en cis, incluidos los promotores centrales y los elementos proximales a los promotores que se encuentran cerca de los sitios de inicio de la transcripción de los genes, corriente arriba en el ADN (hacia la región 5' del sentido hebra). Otros módulos reguladores cis importantes se localizan en regiones de ADN que están distantes de los sitios de inicio de la transcripción. Estos incluyen potenciadores, silenciadores, aisladores y elementos de amarre. Los potenciadores y sus factores de transcripción asociados tienen un papel principal en la regulación de la expresión génica.
Los potenciadores son regiones del genoma que regulan los genes. Los potenciadores controlan los programas de expresión génica específicos del tipo de célula, la mayoría de las veces recorriendo largas distancias para acercarse físicamente a los promotores de sus genes objetivo. Múltiples potenciadores, cada uno a menudo decenas o cientos de miles de nucleótidos distantes de sus genes objetivo, enlazan con sus promotores de genes objetivo y se coordinan entre sí para controlar la expresión génica.
La ilustración muestra un potenciador dando vueltas para acercarse al promotor de un gen objetivo. El bucle está estabilizado por un dímero de una proteína conectora (por ejemplo, dímero de CTCF o YY1). Un miembro del dímero está anclado a su motivo de unión en el potenciador y el otro miembro está anclado a su motivo de unión en el promotor (representado por los zigzags rojos en la ilustración). Varios factores de transcripción específicos de la función celular (entre los aproximadamente 1600 factores de transcripción en una célula humana) generalmente se unen a motivos específicos en un potenciador. Una pequeña combinación de estos factores de transcripción unidos a potenciadores, cuando un bucle de ADN los acerca a un promotor, gobierna el nivel de transcripción del gen objetivo. El mediador (un complejo que generalmente consta de unas 26 proteínas en una estructura que interactúa) comunica las señales reguladoras de los factores de transcripción unidos al ADN potenciadores directamente a la enzima ARN polimerasa II (pol II) unida al promotor.
Los potenciadores, cuando están activos, generalmente se transcriben de ambas cadenas de ADN con ARN polimerasas que actúan en dos direcciones diferentes, produciendo dos ARNe como se ilustra en la figura. Un potenciador inactivo puede unirse a un factor de transcripción inactivo. La fosforilación del factor de transcripción puede activarlo y ese factor de transcripción activado puede entonces activar el potenciador al que está unido (ver la pequeña estrella roja que representa la fosforilación del factor de transcripción unido al potenciador en la ilustración). Un potenciador activado comienza la transcripción de su ARN antes de activar la transcripción del ARN mensajero de su gen objetivo.
Metilación y desmetilación del ADN en la regulación transcripcional

La metilación del ADN es un mecanismo generalizado para la influencia epigenética en la expresión génica y se observa en bacterias y eucariotas y tiene funciones en el silenciamiento de la transcripción hereditaria y la regulación de la transcripción. La metilación ocurre con mayor frecuencia en una citosina (ver Figura). La metilación de la citosina ocurre principalmente en secuencias de dinucleótidos donde una citosina es seguida por una guanina, un sitio CpG. El número de sitios CpG en el genoma humano es de unos 28 millones. Según el tipo de célula, alrededor del 70% de los sitios CpG tienen una citosina metilada.
La metilación de la citosina en el ADN tiene un papel importante en la regulación de la expresión génica. La metilación de CpG en una región promotora de un gen generalmente reprime la transcripción de genes, mientras que la metilación de CpG en el cuerpo de un gen aumenta la expresión. Las enzimas TET juegan un papel central en la desmetilación de citosinas metiladas. La desmetilación de CpG en un promotor de gen por la actividad de la enzima TET aumenta la transcripción del gen.
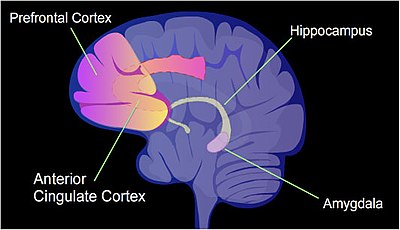
Regulación transcripcional en aprendizaje y memoria
En una rata, el condicionamiento del miedo contextual (CFC) es una experiencia de aprendizaje dolorosa. Solo un episodio de CFC puede resultar en un recuerdo aterrador para toda la vida. Después de un episodio de CFC, la metilación de la citosina se altera en las regiones promotoras de aproximadamente el 9,17% de todos los genes en el ADN de la neurona del hipocampo de una rata. El hipocampo es donde inicialmente se almacenan los nuevos recuerdos. Después de CFC, alrededor de 500 genes han aumentado la transcripción (a menudo debido a la desmetilación de los sitios CpG en una región promotora) y alrededor de 1000 genes han disminuido la transcripción (a menudo debido a la 5-metilcitosina recién formada en los sitios CpG en una región promotora). El patrón de genes inducidos y reprimidos dentro de las neuronas parece proporcionar una base molecular para formar el primer recuerdo transitorio de este evento de entrenamiento en el hipocampo del cerebro de la rata.
Se han establecido algunos mecanismos específicos que guían las nuevas metilaciones y desmetilaciones del ADN en el hipocampo durante el establecimiento de la memoria (consulte el resumen). Un mecanismo incluye guiar la isoforma corta de la enzima de desmetilación del ADN TET1, TET1s, a alrededor de 600 ubicaciones en el genoma. La guía se realiza mediante la asociación de TET1 con la proteína EGR1, un factor de transcripción importante en la formación de la memoria. Llevar TET1 a estos lugares inicia la desmetilación del ADN en esos sitios, regulando al alza los genes asociados. Un segundo mecanismo involucra a DNMT3A2, una isoforma de empalme de la ADN metiltransferasa DNMT3A, que agrega grupos metilo a las citosinas en el ADN. Esta isoforma es inducida por la actividad sináptica, y su ubicación de acción parece estar determinada por modificaciones postraduccionales de las histonas (un código de histonas). Los nuevos ARN mensajeros resultantes luego son transportados por partículas RNP mensajeras (gránulos neuronales) a las sinapsis de las neuronas, donde pueden traducirse en proteínas que afectan las actividades de las sinapsis.
En particular, el gen del factor neurotrófico derivado del cerebro (BDNF) se conoce como "gen del aprendizaje". Después de CFC hubo una regulación positiva de la expresión del gen BDNF, relacionada con la disminución de la metilación de CpG de ciertos promotores internos del gen, y esto se correlacionó con el aprendizaje.
Regulación transcripcional en cáncer
La mayoría de los promotores de genes contienen una isla CpG con numerosos sitios CpG. Cuando muchos de los sitios CpG del promotor de un gen están metilados, el gen se silencia. Los cánceres colorrectales suelen tener de 3 a 6 mutaciones de conductor y de 33 a 66 mutaciones de autoestopista o pasajero. Sin embargo, el silenciamiento transcripcional puede ser más importante que la mutación para provocar la progresión al cáncer. Por ejemplo, en los cánceres colorrectales, alrededor de 600 a 800 genes son silenciados transcripcionalmente por la metilación de la isla CpG (ver regulación de la transcripción en el cáncer). La represión transcripcional en el cáncer también puede ocurrir por otros mecanismos epigenéticos, como la expresión alterada de microARN. En el cáncer de mama, la represión transcripcional de BRCA1 puede ocurrir con más frecuencia por microARN-182 transcrito en exceso que por hipermetilación del promotor de BRCA1 (consulte Expresión baja de BRCA1 en cánceres de mama y de ovario).
Regulación postranscripcional
En eucariotas, donde se requiere la exportación de ARN antes de que sea posible la traducción, se cree que la exportación nuclear proporciona un control adicional sobre la expresión génica. Todo el transporte dentro y fuera del núcleo se realiza a través del poro nuclear y el transporte está controlado por una amplia gama de proteínas de importación y exportación.
La expresión de un gen que codifica una proteína solo es posible si el ARN mensajero que lleva el código sobrevive el tiempo suficiente para traducirse. En una célula típica, una molécula de ARN solo es estable si se protege específicamente contra la degradación. La degradación del ARN tiene una importancia particular en la regulación de la expresión en células eucariotas donde el ARNm tiene que viajar distancias significativas antes de ser traducido. En eucariotas, el ARN se estabiliza mediante ciertas modificaciones postranscripcionales, particularmente la tapa 5 'y la cola poliadenilada.
La degradación intencional del ARNm se utiliza no solo como un mecanismo de defensa contra el ARN extraño (normalmente de virus), sino también como una ruta de desestabilización del ARNm. Si una molécula de ARNm tiene una secuencia complementaria a un pequeño ARN de interferencia, entonces se dirige a la destrucción a través de la vía de interferencia del ARN.
Tres regiones principales no traducidas y microARN
Tres regiones principales no traducidas (3'UTR) de los ARN mensajeros (ARNm) a menudo contienen secuencias reguladoras que influyen en la expresión génica después de la transcripción. Tales 3′-UTR a menudo contienen tanto sitios de unión para microARN (miARN) como para proteínas reguladoras. Al unirse a sitios específicos dentro de la 3′-UTR, los miARN pueden disminuir la expresión génica de varios ARNm inhibiendo la traducción o provocando directamente la degradación de la transcripción. La 3′-UTR también puede tener regiones silenciadoras que se unen a proteínas represoras que inhiben la expresión de un ARNm.
La 3′-UTR a menudo contiene elementos de respuesta de microARN (MRE). Los MRE son secuencias a las que se unen los miARN. Estos son motivos predominantes dentro de 3′-UTR. Entre todos los motivos reguladores dentro de las 3′-UTR (por ejemplo, incluidas las regiones silenciadoras), los MRE constituyen aproximadamente la mitad de los motivos.
A partir de 2014, el sitio web miRBase, un archivo de secuencias y anotaciones de miARN, enumeró 28 645 entradas en 233 especies biológicas. De estos, 1.881 miARN estaban en loci de miARN humanos anotados. Se predijo que los miARN tenían un promedio de alrededor de cuatrocientos ARNm objetivo (que afectan la expresión de varios cientos de genes). Friedman et al. estima que >45 000 sitios diana de miARN dentro de los 3'UTR de ARNm humano se conservan por encima de los niveles de fondo, y >el 60 % de los genes codificadores de proteínas humanas han estado bajo presión selectiva para mantener el emparejamiento con los miARN.
Los experimentos directos muestran que un solo miARN puede reducir la estabilidad de cientos de ARNm únicos. Otros experimentos muestran que un solo miARN puede reprimir la producción de cientos de proteínas, pero que esta represión a menudo es relativamente leve (menos del doble).
Los efectos de la desregulación de la expresión génica de miARN parecen ser importantes en el cáncer. Por ejemplo, en los cánceres gastrointestinales, nueve miRNAs han sido identificados como epigenéticamente alterados y efectivos en la regulación a la baja de las enzimas de reparación del ADN.
Los efectos de la desregulación de miARN de la expresión génica también parecen ser importantes en los trastornos neuropsiquiátricos, como la esquizofrenia, el trastorno bipolar, la depresión mayor, la enfermedad de Parkinson, la enfermedad de Alzheimer y los trastornos del espectro autista.
Reglamento de traducción
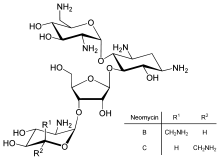
La regulación directa de la traducción es menos prevalente que el control de la transcripción o la estabilidad del ARNm, pero se usa ocasionalmente. La inhibición de la traducción de proteínas es un objetivo importante para las toxinas y los antibióticos, por lo que pueden matar una célula anulando su control normal de expresión génica. Los inhibidores de la síntesis de proteínas incluyen el antibiótico neomicina y la toxina ricina.
Modificaciones postraduccionales
Las modificaciones postraduccionales (PTM) son modificaciones covalentes de las proteínas. Al igual que el empalme de ARN, ayudan a diversificar significativamente el proteoma. Estas modificaciones suelen ser catalizadas por enzimas. Además, procesos como las adiciones covalentes a los residuos de la cadena lateral de aminoácidos a menudo pueden ser revertidos por otras enzimas. Sin embargo, algunos, como la escisión proteolítica del esqueleto de la proteína, son irreversibles.
Los PTM desempeñan muchas funciones importantes en la célula. Por ejemplo, la fosforilación está involucrada principalmente en la activación y desactivación de proteínas y en las vías de señalización. Los PTM participan en la regulación transcripcional: una función importante de la acetilación y la metilación es la modificación de la cola de las histonas, que altera la accesibilidad del ADN para la transcripción. También se pueden ver en el sistema inmunológico, donde la glicosilación juega un papel clave. Un tipo de PTM puede iniciar otro tipo de PTM, como se puede ver en cómo la ubiquitinación marca las proteínas para su degradación a través de la proteólisis. La proteólisis, además de participar en la descomposición de las proteínas, también es importante para activarlas y desactivarlas, y para regular procesos biológicos como la transcripción del ADN y la muerte celular.
Medición

La medición de la expresión génica es una parte importante de muchas ciencias de la vida, ya que la capacidad de cuantificar el nivel en el que se expresa un gen en particular dentro de una célula, tejido u organismo puede proporcionar mucha información valiosa. Por ejemplo, medir la expresión génica puede:
- Identificar la infección viral de una célula (expresión de proteína viral).
- Determinar la susceptibilidad de un individuo al cáncer (expresión del oncogene).
- Encuentre si una bacteria es resistente a la penicilina (expresión beta-lactamasa).
Del mismo modo, el análisis de la ubicación de la expresión de proteínas es una herramienta poderosa, y esto se puede hacer a escala de organismo o celular. La investigación de la localización es particularmente importante para el estudio del desarrollo en organismos multicelulares y como indicador de la función proteica en células individuales. Idealmente, la medición de la expresión se realiza detectando el producto génico final (para muchos genes, esta es la proteína); sin embargo, a menudo es más fácil detectar uno de los precursores, normalmente el ARNm, e inferir los niveles de expresión génica a partir de estas mediciones.
Cuantificación de ARNm
Los niveles de ARNm se pueden medir cuantitativamente mediante transferencia Northern, que proporciona información sobre el tamaño y la secuencia de las moléculas de ARNm. Una muestra de ARN se separa en un gel de agarosa y se hibrida con una sonda de ARN marcada radiactivamente que es complementaria a la secuencia diana. A continuación, el ARN radiomarcado se detecta mediante una autorradiografía. Debido a que el uso de reactivos radiactivos hace que el procedimiento lleve mucho tiempo y sea potencialmente peligroso, se han desarrollado métodos alternativos de etiquetado y detección, como las químicas de digoxigenina y biotina. Las desventajas percibidas de la transferencia de Northern son que se requieren grandes cantidades de ARN y que la cuantificación puede no ser completamente precisa, ya que implica medir la fuerza de la banda en una imagen de un gel. Por otro lado, la información adicional sobre el tamaño del ARNm del Northern blot permite la discriminación de transcritos empalmados alternativamente.
Otro enfoque para medir la abundancia de mRNA es RT-qPCR. En esta técnica, a la transcripción inversa le sigue una PCR cuantitativa. La transcripción inversa primero genera una plantilla de ADN a partir del ARNm; esta plantilla monocatenaria se llama cDNA. La plantilla de cDNA luego se amplifica en el paso cuantitativo, durante el cual la fluorescencia emitida por las sondas de hibridación marcadas o los colorantes intercalados cambia a medida que avanza el proceso de amplificación del ADN. Con una curva estándar cuidadosamente construida, qPCR puede producir una medición absoluta del número de copias del ARNm original, generalmente en unidades de copias por nanolitro de tejido homogeneizado o copias por célula. qPCR es muy sensible (la detección de una sola molécula de ARNm es teóricamente posible), pero puede ser costosa según el tipo de indicador utilizado; las sondas de oligonucleótidos marcadas con fluorescencia son más caras que los colorantes fluorescentes intercalados no específicos.
Para el perfil de expresión o el análisis de alto rendimiento de muchos genes dentro de una muestra, se puede realizar PCR cuantitativa para cientos de genes simultáneamente en el caso de matrices de baja densidad. Un segundo enfoque es el microarreglo de hibridación. Una sola matriz o "chip" puede contener sondas para determinar los niveles de transcripción de cada gen conocido en el genoma de uno o más organismos. Alternativamente, "basado en etiquetas" Se pueden utilizar tecnologías como el análisis en serie de la expresión génica (SAGE) y RNA-Seq, que pueden proporcionar una medida relativa de la concentración celular de diferentes ARNm. Una ventaja de los métodos basados en etiquetas es la 'arquitectura abierta', que permite la medición exacta de cualquier transcripción, con una secuencia conocida o desconocida. La secuenciación de próxima generación (NGS), como RNA-Seq, es otro enfoque, que produce grandes cantidades de datos de secuencia que se pueden comparar con un genoma de referencia. Aunque la NGS consume comparativamente mucho tiempo, es costosa y requiere muchos recursos, puede identificar polimorfismos de un solo nucleótido, variantes de corte y empalme y genes novedosos, y también puede usarse para perfilar la expresión en organismos para los que hay poca o ninguna información de secuencia disponible..
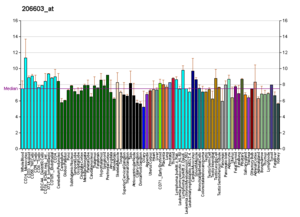
Perfiles de ARN en Wikipedia
Perfiles como estos se encuentran para casi todas las proteínas enumeradas en Wikipedia. Son generados por organizaciones como el Instituto de Genómica de la Fundación de Investigación Novartis y el Instituto Europeo de Bioinformática. Se puede encontrar información adicional buscando en sus bases de datos (para ver un ejemplo del transportador GLUT4 que se muestra aquí, consulte la cita). Estos perfiles indican el nivel de expresión de ADN (y, por lo tanto, de ARN producido) de una determinada proteína en un determinado tejido, y están codificados por colores en consecuencia en las imágenes ubicadas en el cuadro de proteínas en el lado derecho de cada página de Wikipedia.
Cuantificación de proteínas
Para los genes que codifican proteínas, el nivel de expresión se puede evaluar directamente mediante una serie de métodos con algunas claras analogías con las técnicas para la cuantificación del ARNm.
Uno de los métodos más utilizados es realizar un Western blot contra la proteína de interés. Esto da información sobre el tamaño de la proteína además de su identidad. Una muestra (a menudo lisado celular) se separa en un gel de poliacrilamida, se transfiere a una membrana y luego se sondea con un anticuerpo contra la proteína de interés. El anticuerpo puede conjugarse con un fluoróforo o con peroxidasa de rábano picante para obtener imágenes y/o cuantificación. La naturaleza basada en gel de este ensayo hace que la cuantificación sea menos precisa, pero tiene la ventaja de poder identificar modificaciones posteriores en la proteína, por ejemplo, proteólisis o ubiquitinación, a partir de cambios de tamaño.
Correlación ARN-proteína
Si bien la transcripción refleja directamente la expresión génica, el número de copias de las moléculas de ARNm no se correlaciona directamente con el número de moléculas de proteína traducidas del ARNm. La cuantificación de proteína y ARNm permite una correlación de los dos niveles. La regulación en cada paso de la expresión génica puede afectar la correlación, como se muestra para la regulación de la traducción o la estabilidad de la proteína. Los factores postraduccionales, como el transporte de proteínas en células altamente polares, también pueden influir en la correlación de ARNm-proteína medida.
Localización
El análisis de la expresión no se limita a la cuantificación; también se puede determinar la localización. El ARNm puede detectarse con una hebra de ARNm complementaria adecuadamente marcada y la proteína puede detectarse a través de anticuerpos marcados. Luego, la muestra analizada se observa por microscopía para identificar dónde se encuentra el ARNm o la proteína.
Al reemplazar el gen con una nueva versión fusionada con un marcador de proteína verde fluorescente (o similar), la expresión puede cuantificarse directamente en células vivas. Esto se hace mediante imágenes usando un microscopio de fluorescencia. Es muy difícil clonar una proteína fusionada con GFP en su ubicación nativa en el genoma sin afectar los niveles de expresión, por lo que este método a menudo no se puede usar para medir la expresión génica endógena. Sin embargo, se usa ampliamente para medir la expresión de un gen introducido artificialmente en la célula, por ejemplo, a través de un vector de expresión. Es importante tener en cuenta que al fusionar una proteína objetivo con un indicador fluorescente, el comportamiento de la proteína, incluida su localización celular y nivel de expresión, puede cambiar significativamente.
El ensayo inmunoabsorbente ligado a enzimas funciona mediante el uso de anticuerpos inmovilizados en una placa de microtitulación para capturar las proteínas de interés de las muestras añadidas al pocillo. Utilizando un anticuerpo de detección conjugado con una enzima o un fluoróforo, la cantidad de proteína unida se puede medir con precisión mediante detección fluorométrica o colorimétrica. El proceso de detección es muy similar al de una transferencia Western, pero al evitar los pasos de gel se puede lograr una cuantificación más precisa.
Sistema de expresión
Un sistema de expresión es un sistema diseñado específicamente para la producción de un producto génico de elección. Normalmente es una proteína, aunque también puede ser ARN, como ARNt o una ribozima. Un sistema de expresión consta de un gen, normalmente codificado por ADN, y la maquinaria molecular requerida para transcribir el ADN en ARNm y traducir el ARNm en proteína utilizando los reactivos provistos. En el sentido más amplio, esto incluye todas las células vivas, pero el término se usa más normalmente para referirse a la expresión como una herramienta de laboratorio. Por lo tanto, un sistema de expresión es a menudo artificial de alguna manera. Los sistemas de expresión son, sin embargo, un proceso fundamentalmente natural. Los virus son un excelente ejemplo en el que se replican utilizando la célula huésped como sistema de expresión para las proteínas virales y el genoma.
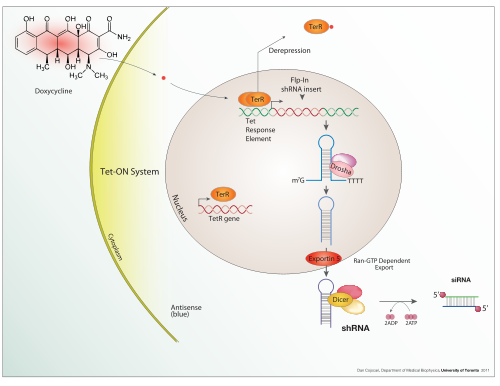
Expresión inducible
La doxiciclina también se usa en "Tet-on" y "Tet-off" Activación transcripcional controlada por tetraciclina para regular la expresión transgénica en organismos y cultivos celulares.
En la naturaleza
Además de estas herramientas biológicas, ciertas configuraciones de ADN observadas naturalmente (genes, promotores, potenciadores, represores) y la propia maquinaria asociada se denominan sistema de expresión. Este término se utiliza normalmente en el caso de que un gen o un conjunto de genes se active en condiciones bien definidas, por ejemplo, el sistema de expresión de interruptor represor simple en el fago lambda y el sistema operador lac en bacterias. Varios sistemas de expresión natural se usan o modifican directamente y se usan para sistemas de expresión artificial tales como el sistema de expresión Tet-on y Tet-off.
Redes de genes
En ocasiones, los genes se han considerado como nodos en una red, donde las entradas son proteínas, como factores de transcripción, y las salidas son el nivel de expresión génica. El nodo en sí realiza una función, y se ha interpretado que la operación de estas funciones realiza un tipo de procesamiento de información dentro de las células y determina el comportamiento celular.
Las redes de genes también se pueden construir sin formular un modelo causal explícito. Este suele ser el caso cuando se ensamblan redes a partir de grandes conjuntos de datos de expresión. La covariación y correlación de la expresión se calcula a través de una gran muestra de casos y mediciones (a menudo datos transcriptómicos o proteómicos). La fuente de variación puede ser experimental o natural (observacional). Hay varias formas de construir redes de expresión génica, pero un enfoque común es calcular una matriz de todas las correlaciones de expresión por pares entre condiciones, puntos de tiempo o individuos y convertir la matriz (después de establecer un umbral en algún valor de corte) en una representación gráfica en la que los nodos representan genes, transcripciones o proteínas y los bordes que conectan estos nodos representan la fuerza de la asociación (ver GeneNetwork GeneNetwork 2).
Técnicas y herramientas
Las siguientes técnicas experimentales se utilizan para medir la expresión génica y se enumeran en orden cronológico aproximado, comenzando con las tecnologías más antiguas y establecidas. Se dividen en dos grupos en función de su grado de multiplexidad.
- Técnicas de bajo a medio complejo:
- Reporter gene
- Parcela norte
- Western blot
- Hibridación fluorescente in situ
- Trascripción inversa PCR
- Técnicas de complejo superior:
- SAGE
- Microarrayo de ADN
- Tiling array
- RNA-Seq
Bases de datos de expresión génica
- Gene expression omnibus (GEO) at NCBI
- Expression Atlas en el EBI
- Mouse Gene Expression Database en el Laboratorio Jackson
- CollecTF: una base de datos de sitios de unión de factores validados experimentalmente en Bacteria.
- COLOMBOS: colección de compendia de expresión bacteriana.
- Muchos microbios Microarrays Base de datos: microbial Affymetrix data
Contenido relacionado
Kalmia latifolia
Homo habilis
Gram (desambiguación)