Factor de transcripcion
En biología molecular, un factor de transcripción (TF) (o factor de unión a ADN específico de secuencia) es una proteína que controla la tasa de transcripción de la información genética del ADN al ARN mensajero, mediante la unión a una secuencia específica de ADN. La función de los TF es regular (encender y apagar) los genes para asegurarse de que se expresen en las células deseadas en el momento adecuado y en la cantidad adecuada a lo largo de la vida de la célula y el organismo. Los grupos de TF funcionan de manera coordinada para dirigir la división celular, el crecimiento celular y la muerte celular a lo largo de la vida; migración y organización celular (plan corporal) durante el desarrollo embrionario; e intermitentemente en respuesta a señales del exterior de la célula, como una hormona. Hay 1500-1600 TF en el genoma humano. Los factores de transcripción son miembros tanto del proteoma como del reguloma.
Los TF funcionan solos o con otras proteínas en un complejo, promoviendo (como activador) o bloqueando (como represor) el reclutamiento de ARN polimerasa (la enzima que realiza la transcripción de la información genética del ADN al ARN) para genes específicos.
Una característica definitoria de los TF es que contienen al menos un dominio de unión al ADN (DBD), que se une a una secuencia específica de ADN adyacente a los genes que regulan. Los TF se agrupan en clases según sus DBD. Otras proteínas, como los coactivadores, los remodeladores de la cromatina, las histonas acetiltransferasas, las histonas desacetilasas, las quinasas y las metilasas también son esenciales para la regulación génica, pero carecen de dominios de unión al ADN y, por lo tanto, no son TF.
Los TF son de interés en medicina porque las mutaciones de TF pueden causar enfermedades específicas y los medicamentos pueden dirigirse potencialmente hacia ellas.
Número
Los factores de transcripción son esenciales para la regulación de la expresión génica y, como consecuencia, se encuentran en todos los organismos vivos. La cantidad de factores de transcripción que se encuentran dentro de un organismo aumenta con el tamaño del genoma, y los genomas más grandes tienden a tener más factores de transcripción por gen.
Hay aproximadamente 2800 proteínas en el genoma humano que contienen dominios de unión al ADN, y se supone que 1600 de ellas funcionan como factores de transcripción, aunque otros estudios indican que es un número menor. Por lo tanto, aproximadamente el 10% de los genes del genoma codifican factores de transcripción, lo que convierte a esta familia en la familia más grande de proteínas humanas. Además, los genes a menudo están flanqueados por varios sitios de unión para distintos factores de transcripción, y la expresión eficiente de cada uno de estos genes requiere la acción cooperativa de varios factores de transcripción diferentes (ver, por ejemplo, factores nucleares de hepatocitos). Por lo tanto, el uso combinatorio de un subconjunto de los aproximadamente 2000 factores de transcripción humanos explica fácilmente la regulación única de cada gen en el genoma humano durante el desarrollo.
Mecanismo
Los factores de transcripción se unen a las regiones potenciadoras o promotoras del ADN adyacentes a los genes que regulan. Dependiendo del factor de transcripción, la transcripción del gen adyacente se regula hacia arriba o hacia abajo. Los factores de transcripción utilizan una variedad de mecanismos para la regulación de la expresión génica. Estos mecanismos incluyen:
- estabilizar o bloquear la unión de la polimerasa RNA al ADN
- cataliza la acetilación o desacetilación de las proteínas de la piedra. El factor de transcripción puede hacer esto directamente o reclutar otras proteínas con esta actividad catalítica. Muchos factores de transcripción usan uno o el otro de dos mecanismos opuestos para regular la transcripción:
- actividad histone acetyltransferase (HAT) – acetila las proteínas histone, que debilita la asociación de ADN con histonas, lo que hace que el ADN sea más accesible a la transcripción, por lo que la transcripción está regulando
- actividad histone deacetylase (HDAC) – deacetiliza las proteínas histone, que fortalece la asociación de ADN con histonas, lo que hace que el ADN sea menos accesible a la transcripción, por lo que la transcripción de baja regulación
- reclutar coactivador o proteínas Corepressor al complejo de ADN factor de transcripción
Función
Los factores de transcripción son uno de los grupos de proteínas que leen e interpretan el "modelo" en el ADN. Se unen al ADN y ayudan a iniciar un programa de aumento o disminución de la transcripción de genes. Como tales, son vitales para muchos procesos celulares importantes. A continuación se presentan algunas de las funciones importantes y roles biológicos en los que están involucrados los factores de transcripción:
Regulación transcripcional basal
En los eucariotas, una clase importante de factores de transcripción llamados factores de transcripción generales (GTF) son necesarios para que se produzca la transcripción. Muchos de estos GTF en realidad no se unen al ADN, sino que son parte del gran complejo de preiniciación de la transcripción que interactúa directamente con la ARN polimerasa. Los GTF más comunes son TFIIA, TFIIB, TFIID (ver también proteína de unión a TATA), TFIIE, TFIIF y TFIIH. El complejo de preiniciación se une a las regiones promotoras del ADN aguas arriba del gen que regulan.
Mejora diferencial de la transcripción
Otros factores de transcripción regulan diferencialmente la expresión de varios genes al unirse a regiones potenciadoras de ADN adyacentes a genes regulados. Estos factores de transcripción son fundamentales para garantizar que los genes se expresen en la célula adecuada en el momento adecuado y en la cantidad adecuada, según los requisitos cambiantes del organismo.
Desarrollo
Muchos factores de transcripción en organismos multicelulares están involucrados en el desarrollo. En respuesta a los estímulos, estos factores de transcripción activan o desactivan la transcripción de los genes apropiados, lo que, a su vez, permite cambios en la morfología celular o las actividades necesarias para la determinación del destino celular y la diferenciación celular. La familia de factores de transcripción Hox, por ejemplo, es importante para la formación adecuada del patrón corporal en organismos tan diversos como las moscas de la fruta y los humanos. Otro ejemplo es el factor de transcripción codificado por el gen de la región Y determinante del sexo (SRY), que juega un papel importante en la determinación del sexo en humanos.
Respuesta a señales intercelulares
Las células pueden comunicarse entre sí mediante la liberación de moléculas que producen cascadas de señalización dentro de otra célula receptiva. Si la señal requiere una regulación positiva o negativa de los genes en la célula receptora, a menudo los factores de transcripción estarán aguas abajo en la cascada de señalización. La señalización de estrógeno es un ejemplo de una cascada de señalización bastante corta que involucra el factor de transcripción del receptor de estrógeno: el estrógeno es secretado por tejidos como los ovarios y la placenta, cruza la membrana celular de la célula receptora y se une al receptor de estrógeno en la célula. #39;s citoplasma. El receptor de estrógeno luego va al núcleo de la célula y se une a sus sitios de unión al ADN, cambiando la regulación transcripcional de los genes asociados.
Respuesta al entorno
Los factores de transcripción no solo actúan aguas abajo de las cascadas de señalización relacionadas con estímulos biológicos, sino que también pueden estar aguas abajo de las cascadas de señalización involucradas en estímulos ambientales. Los ejemplos incluyen el factor de choque térmico (HSF), que regula al alza los genes necesarios para la supervivencia a temperaturas más altas, el factor inducible por hipoxia (HIF), que regula al alza los genes necesarios para la supervivencia celular en entornos con poco oxígeno, y la proteína de unión al elemento regulador de esteroles (SREBP), que ayuda a mantener los niveles adecuados de lípidos en la célula.
Control del ciclo celular
Muchos factores de transcripción, especialmente algunos que son protooncogenes o supresores de tumores, ayudan a regular el ciclo celular y, como tales, determinan el tamaño de una célula y cuándo puede dividirse en dos células hijas. Un ejemplo es el oncogén Myc, que tiene funciones importantes en el crecimiento celular y la apoptosis.
Patogénesis
Los factores de transcripción también se pueden usar para alterar la expresión génica en una célula huésped para promover la patogénesis. Un ejemplo bien estudiado de esto son los efectores tipo activador de la transcripción (efectores TAL) secretados por la bacteria Xanthomonas. Cuando se inyectan en las plantas, estas proteínas pueden ingresar al núcleo de la célula vegetal, unir secuencias promotoras de plantas y activar la transcripción de genes vegetales que ayudan en la infección bacteriana. Los efectores TAL contienen una región de repetición central en la que existe una relación simple entre la identidad de dos residuos críticos en repeticiones secuenciales y bases de ADN secuenciales en el sitio objetivo del efector TAL. Es probable que esta propiedad facilite la evolución de estas proteínas para competir mejor con los mecanismos de defensa de la célula huésped.
Regulación
Es común en biología que los procesos importantes tengan múltiples capas de regulación y control. Esto también es cierto con los factores de transcripción: los factores de transcripción no solo controlan las tasas de transcripción para regular las cantidades de productos génicos (ARN y proteínas) disponibles para la célula, sino que los factores de transcripción mismos están regulados (a menudo por otros factores de transcripción). A continuación se muestra una breve sinopsis de algunas de las formas en que se puede regular la actividad de los factores de transcripción:
Síntesis
Los factores de transcripción (como todas las proteínas) se transcriben de un gen en un cromosoma a ARN, y luego el ARN se traduce a proteína. Cualquiera de estos pasos puede regularse para afectar la producción (y por lo tanto la actividad) de un factor de transcripción. Una implicación de esto es que los factores de transcripción pueden regularse a sí mismos. Por ejemplo, en un bucle de retroalimentación negativa, el factor de transcripción actúa como su propio represor: si la proteína del factor de transcripción se une al ADN de su propio gen, regula a la baja la producción de más de sí mismo. Este es un mecanismo para mantener niveles bajos de un factor de transcripción en una célula.
Localización nuclear
En los eucariotas, los factores de transcripción (como la mayoría de las proteínas) se transcriben en el núcleo pero luego se traducen en el citoplasma de la célula. Muchas proteínas que están activas en el núcleo contienen señales de localización nuclear que las dirigen al núcleo. Pero, para muchos factores de transcripción, este es un punto clave en su regulación. Las clases importantes de factores de transcripción, como algunos receptores nucleares, primero deben unirse a un ligando mientras están en el citoplasma antes de que puedan trasladarse al núcleo.
Activación
Los factores de transcripción pueden activarse (o desactivarse) a través de su dominio de detección de señales mediante una serie de mecanismos que incluyen:
- ligand binding – No sólo es ligand vinculante capaz de influenciar donde un factor de transcripción se encuentra dentro de una célula pero ligand unión también puede afectar si el factor de transcripción está en un estado activo y capaz de ligar ADN u otros cofactores (ver, por ejemplo, los receptores nucleares).
- fosforilación – Muchos factores de transcripción, como las proteínas STAT, deben ser fosforiladas antes de que puedan atar el ADN.
- interacción con otros factores de transcripción (Por ejemplo., homo- o hetero-dimerización) o proteínas coregulatorias
Accesibilidad del sitio de unión al ADN
En los eucariotas, el ADN se organiza con la ayuda de las histonas en partículas compactas llamadas nucleosomas, donde las secuencias de alrededor de 147 pares de bases de ADN dan ~1,65 vueltas alrededor de los octámeros de proteína histona. El ADN dentro de los nucleosomas es inaccesible para muchos factores de transcripción. Algunos factores de transcripción, los llamados factores pioneros, aún pueden unirse a sus sitios de unión al ADN en el ADN nucleosómico. Para la mayoría de los demás factores de transcripción, el nucleosoma debe ser desenrollado activamente por motores moleculares como los remodeladores de la cromatina. Alternativamente, el nucleosoma se puede desenvolver parcialmente por las fluctuaciones térmicas, lo que permite el acceso temporal al sitio de unión del factor de transcripción. En muchos casos, un factor de transcripción necesita competir para unirse a su sitio de unión al ADN con otros factores de transcripción e histonas o proteínas de cromatina no histonas. Los pares de factores de transcripción y otras proteínas pueden desempeñar funciones antagónicas (activador frente a represor) en la regulación del mismo gen.
Disponibilidad de otros cofactores/factores de transcripción
La mayoría de los factores de transcripción no funcionan solos. Muchas familias grandes de TF forman interacciones homotípicas o heterotípicas complejas a través de la dimerización. Para que se produzca la transcripción de genes, una serie de factores de transcripción deben unirse a las secuencias reguladoras del ADN. Esta colección de factores de transcripción, a su vez, recluta proteínas intermediarias como cofactores que permiten el reclutamiento eficiente del complejo de preiniciación y la ARN polimerasa. Por lo tanto, para que un solo factor de transcripción inicie la transcripción, todas estas otras proteínas también deben estar presentes, y el factor de transcripción debe estar en un estado en el que pueda unirse a ellas si es necesario. Los cofactores son proteínas que modulan los efectos de los factores de transcripción. Los cofactores son intercambiables entre promotores de genes específicos; el complejo proteico que ocupa el ADN promotor y la secuencia de aminoácidos del cofactor determinan su conformación espacial. Por ejemplo, ciertos receptores de esteroides pueden intercambiar cofactores con NF-κB, que es un cambio entre la inflamación y la diferenciación celular; por lo tanto, los esteroides pueden afectar la respuesta inflamatoria y la función de ciertos tejidos.
Interacción con citosina metilada
Los factores de transcripción y las citosinas metiladas en el ADN tienen funciones importantes en la regulación de la expresión génica. (La metilación de la citosina en el ADN ocurre principalmente cuando a la citosina le sigue la guanina en la secuencia de ADN 5' a 3', un sitio CpG). La metilación de los sitios CpG en una región promotora de un gen generalmente reprime la transcripción del gen, mientras que la metilación de CpG en el cuerpo de un gen aumenta la expresión. Las enzimas TET juegan un papel central en la desmetilación de citosinas metiladas. La desmetilación de CpG en un promotor de gen por la actividad de la enzima TET aumenta la transcripción del gen.
Se evaluaron los sitios de unión al ADN de 519 factores de transcripción. De estos, 169 factores de transcripción (33 %) no tenían dinucleótidos CpG en sus sitios de unión, y 33 factores de transcripción (6 %) podían unirse a un motivo que contenía CpG pero no mostraban preferencia por un sitio de unión con un motivo metilado o CpG no metilado. Hubo 117 factores de transcripción (23 %) cuya unión a su secuencia de unión se inhibió si contenía un sitio CpG metilado, 175 factores de transcripción (34 %) que mejoraron la unión si su secuencia de unión tenía un sitio CpG metilado y 25 factores de transcripción los factores (5%) se inhibieron o mejoraron la unión dependiendo de en qué parte de la secuencia de unión se encontraba la CpG metilada.
Las enzimas TET no se unen específicamente a la metilcitosina excepto cuando son reclutadas (ver desmetilación del ADN). Se ha demostrado que múltiples factores de transcripción importantes en la diferenciación celular y la especificación del linaje, incluidos NANOG, SALL4A, WT1, EBF1, PU.1 y E2A, reclutan enzimas TET en loci genómicos específicos (principalmente potenciadores) para actuar sobre la metilcitosina (mC) y convertirlo en hidroximetilcitosina hmC (y en la mayoría de los casos marcarlos para su posterior desmetilación completa a citosina). La conversión de mC a hmC mediada por TET parece interrumpir la unión de proteínas de unión a 5mC, incluidas las proteínas MECP2 y MBD (dominio de unión a metil-CpG), lo que facilita la remodelación del nucleosoma y la unión de factores de transcripción, lo que activa la transcripción de esos genes. EGR1 es un factor de transcripción importante en la formación de la memoria. Tiene un papel esencial en la reprogramación epigenética de las neuronas cerebrales. El factor de transcripción EGR1 recluta la proteína TET1 que inicia una vía de desmetilación del ADN. EGR1, junto con TET1, se emplea en la programación de la distribución de sitios de metilación en el ADN del cerebro durante el desarrollo del cerebro y el aprendizaje (ver Epigenética en el aprendizaje y la memoria).
Estructura


Los factores de transcripción tienen una estructura modular y contienen los siguientes dominios:
- dominio de unión de ADN ()DBD), que se adhiere a secuencias específicas de ADN (enhancer o promotor. Componente necesario para todos los vectores. Utilizado para conducir la transcripción de las secuencias de promotores transgene del vector) adyacentes a los genes regulados. secuencias de ADN que unen los factores de transcripción a menudo se denominan elementos de respuesta.
- Dominio de activación ()AD), que contiene sitios de unión para otras proteínas como los Coregulators de la transcripción. Estos sitios vinculantes se denominan frecuentemente funciones de activación ()AFs), Dominio de transactivación ()TAD) o Trans-activación de dominio TAD pero no mezcla con dominio asociativo topológicamente TAD.
- Un opcional dominio de señalización ()SSD)Por ejemplo., un dominio ligand-binding), que siente las señales externas y, en respuesta, transmite estas señales al resto del complejo de transcripción, dando lugar a la regulación de la expresión génica. Además, los dominios DBD y señalización pueden residir en proteínas separadas que se asocian dentro del complejo de transcripción para regular la expresión génica.
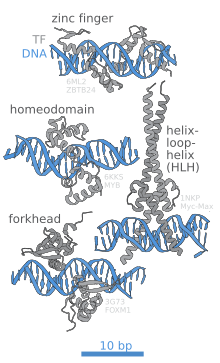
Dominio de unión al ADN
La porción (dominio) del factor de transcripción que se une al ADN se denomina dominio de unión al ADN. A continuación se muestra una lista parcial de algunas de las principales familias de dominios de unión al ADN/factores de transcripción:
| Familia | InterPro | Pfam | SCOP |
|---|---|---|---|
| básico helix-loop-helix | InterPro:IPR001092 | Pfam PF00010 | SCOP 47460 |
| cremallera básica-leucina (bZIP) | InterPro:IPR004827 | Pfam PF00170 | SCOP 57959 |
| Dominio de los reguladores de la respuesta bipartita | InterPro:IPR001789 | Pfam PF00072 | SCOP 46894 |
| AP2/ERF/GCC box | InterPro:IPR001471 | Pfam PF00847 | SCOP 54176 |
| helix-turn-helix | |||
| Las proteínas homeodomain, que son codificadas por genes homeobox, son factores de transcripción. Las proteínas del Homeodomain desempeñan funciones críticas en la regulación del desarrollo. | InterPro:IPR009057 | Pfam PF00046 | SCOP 46689 |
| represor de lambda | InterPro:IPR010982 | SCOP 47413 | |
| srf-like (factor de respuesta srum) | InterPro:IPR002100 | Pfam PF00319 | SCOP 55455 |
| caja emparejada | |||
| helix alado | InterPro:IPR013196 | Pfam PF08279 | SCOP 46785 |
| Dedos de zinc | |||
| * multidominio Cys2Su2 Dedos de zinc | InterPro:IPR007087 | Pfam PF00096 | SCOP 57667 |
| * Zn2/Cys6 | SCOP 57701 | ||
| * Zn2/Cys8 dedo de zinc del receptor nuclear | InterPro:IPR001628 | Pfam PF00105 | SCOP 57716 |
Elementos de respuesta
La secuencia de ADN a la que se une un factor de transcripción se denomina sitio de unión del factor de transcripción o elemento de respuesta.
Los factores de transcripción interactúan con sus sitios de unión mediante una combinación de fuerzas electrostáticas (de las cuales los enlaces de hidrógeno son un caso especial) y de Van der Waals. Debido a la naturaleza de estas interacciones químicas, la mayoría de los factores de transcripción se unen al ADN de una manera específica de secuencia. Sin embargo, no todas las bases en el sitio de unión del factor de transcripción pueden interactuar con el factor de transcripción. Además, algunas de estas interacciones pueden ser más débiles que otras. Por lo tanto, los factores de transcripción no se unen a una sola secuencia, sino que son capaces de unirse a un subconjunto de secuencias estrechamente relacionadas, cada una con una fuerza de interacción diferente.
Por ejemplo, aunque el sitio de unión consenso para la proteína de unión a TATA (TBP) es TATAAAA, el factor de transcripción TBP también puede unirse a secuencias similares como TATATAT o TATATAA.
Debido a que los factores de transcripción pueden unirse a un conjunto de secuencias relacionadas y estas secuencias tienden a ser cortas, los posibles sitios de unión del factor de transcripción pueden ocurrir por casualidad si la secuencia de ADN es lo suficientemente larga. Sin embargo, es poco probable que un factor de transcripción se una a todas las secuencias compatibles en el genoma de la célula. Otras restricciones, como la accesibilidad al ADN en la célula o la disponibilidad de cofactores, también pueden ayudar a determinar dónde se unirá realmente un factor de transcripción. Por lo tanto, dada la secuencia del genoma, todavía es difícil predecir dónde se unirá realmente un factor de transcripción en una célula viva.
Sin embargo, se puede obtener una especificidad de reconocimiento adicional mediante el uso de más de un dominio de unión al ADN (por ejemplo, DBD en tándem en el mismo factor de transcripción o mediante la dimerización de dos factores de transcripción) que se unen a dos o más secuencias adyacentes de ADN.
Importancia clínica
Los factores de transcripción tienen importancia clínica por al menos dos razones: (1) las mutaciones se pueden asociar con enfermedades específicas y (2) pueden ser objetivos de medicamentos.
Trastornos
Debido a sus funciones importantes en el desarrollo, la señalización intercelular y el ciclo celular, algunas enfermedades humanas se han asociado con mutaciones en los factores de transcripción.
Muchos factores de transcripción son supresores de tumores u oncogenes y, por lo tanto, las mutaciones o la regulación anómala de ellos se asocian con el cáncer. Se sabe que tres grupos de factores de transcripción son importantes en el cáncer humano: (1) las familias NF-kappaB y AP-1, (2) la familia STAT y (3) los receptores de esteroides.
A continuación se muestran algunos de los ejemplos mejor estudiados:
| Estado | Descripción | Locus |
|---|---|---|
| Síndrome de Rett | Las mutaciones en el factor de transcripción MECP2 están asociadas con el síndrome de Rett, un trastorno neurodesarrollado. | Xq28 |
| Diabetes | Una forma rara de diabetes llamada MODY (diabetes de aparición de la fuerza de los jóvenes) puede ser causada por mutaciones en factores nucleares hepatocitos (HNFs) o factor promotor de insulina-1 (IPF1/Pdx1). | múltiple |
| Dispraxia verbal de desarrollo | Las mutaciones en el factor de transcripción FOXP2 están asociadas con la dispraxia verbal de desarrollo, una enfermedad en la que los individuos no pueden producir los movimientos finamente coordinados requeridos para el discurso. | 7q31 |
| Enfermedades autoinmunitarias | Las mutaciones en el factor de transcripción FOXP3 causan una rara forma de enfermedad autoinmune llamada IPEX. | Xp11.23-q13.3 |
| Síndrome de Li-Fraumeni | Causado por mutaciones en el supresor tumoral p53. | 17p13.1 |
| Cáncer de mama | La familia STAT es relevante para el cáncer de mama. | múltiple |
| Cánceres múltiples | La familia HOX está involucrada en una variedad de cánceres. | múltiple |
| Osteoartritis | Mutación o actividad reducida de SOX9 |
Posibles objetivos farmacológicos
Aproximadamente el 10 % de los medicamentos recetados actualmente se dirigen directamente a la clase de receptores nucleares de factores de transcripción. Los ejemplos incluyen tamoxifeno y bicalutamida para el tratamiento del cáncer de mama y de próstata, respectivamente, y varios tipos de esteroides anabólicos y antiinflamatorios. Además, los factores de transcripción a menudo son modulados indirectamente por fármacos a través de cascadas de señalización. Podría ser posible dirigirse directamente a otros factores de transcripción menos explorados, como NF-κB, con fármacos. Se cree que los factores de transcripción fuera de la familia de receptores nucleares son más difíciles de atacar con terapias de moléculas pequeñas, ya que no está claro si son 'medicables'. pero se han hecho progresos en Pax2 y el camino de la muesca.
Papel en la evolución
Las duplicaciones de genes han jugado un papel crucial en la evolución de las especies. Esto se aplica particularmente a los factores de transcripción. Una vez que ocurren como duplicados, las mutaciones acumuladas que codifican para una copia pueden tener lugar sin afectar negativamente la regulación de los objetivos aguas abajo. Sin embargo, recientemente se han dilucidado los cambios de las especificidades de unión al ADN del factor de transcripción Leafy de copia única, que se produce en la mayoría de las plantas terrestres. En ese sentido, un factor de transcripción de una sola copia puede sufrir un cambio de especificidad a través de un intermediario promiscuo sin perder la función. Se han propuesto mecanismos similares en el contexto de todas las hipótesis filogenéticas alternativas y el papel de los factores de transcripción en la evolución de todas las especies.
Papel en la actividad de biocontrol
Los factores de transcripción tienen un papel en la actividad de resistencia que es importante para una actividad de biocontrol exitosa. La resistencia al estrés oxidativo y la detección de pH alcalino fueron aportados por el factor de transcripción Yap1 y Rim101 de Papiliotrema terrestris LS28 como herramientas moleculares que revelaron una comprensión de los mecanismos genéticos subyacentes a la actividad de biocontrol que respaldará los programas de manejo de enfermedades. basado en el control biológico e integrado.
Análisis
Existen diferentes tecnologías disponibles para analizar los factores de transcripción. A nivel genómico, se utilizan comúnmente la secuenciación del ADN y la investigación de bases de datos. La versión proteica del factor de transcripción es detectable mediante el uso de anticuerpos específicos. La muestra se detecta en un western blot. Mediante el uso del ensayo de cambio de movilidad electroforética (EMSA), se puede detectar el perfil de activación de los factores de transcripción. Un enfoque multiplex para la creación de perfiles de activación es un sistema de chip TF en el que se pueden detectar varios factores de transcripción diferentes en paralelo.
El método más utilizado para identificar los sitios de unión del factor de transcripción es la inmunoprecipitación de cromatina (ChIP). Esta técnica se basa en la fijación química de la cromatina con formaldehído, seguida de la coprecipitación del ADN y el factor de transcripción de interés mediante un anticuerpo que se dirige específicamente a esa proteína. Luego, las secuencias de ADN se pueden identificar mediante micromatrices o secuenciación de alto rendimiento (ChIP-seq) para determinar los sitios de unión del factor de transcripción. Si no hay anticuerpos disponibles para la proteína de interés, DamID puede ser una alternativa conveniente.
Clases
Como se describe con más detalle a continuación, los factores de transcripción se pueden clasificar por su (1) mecanismo de acción, (2) función reguladora o (3) homología de secuencia (y, por lo tanto, similitud estructural) en sus dominios de unión al ADN.
Mecanicista
Hay dos clases mecánicas de factores de transcripción:
- Los factores de transcripción generales están involucrados en la formación de un complejo de preiniciación. Los más comunes son abreviados como TFIIA, TFIIB, TFIID, TFIIE, TFIIF y TFIIH. Son ubicuos e interactúan con la región promotora central que rodea el sitio de inicio de la transcripción de todos los genes de clase II.
- Factores de transcripción son proteínas que se unen en algún lugar del sitio de iniciación para estimular o reprimir la transcripción. Estos son aproximadamente sinónimos con factores de transcripción específicos, porque varían considerablemente dependiendo de qué secuencias de reconocimiento están presentes en la proximidad del gen.
| Ejemplos de factores de transcripción específicos | |||
|---|---|---|---|
| Factor | Tipo estructural | Secuencia de reconocimiento | Binds as |
| SP1 | Dedo de zinc | 5'-GGGCG-3' | Monomer |
| AP-1 | Cierre básico | 5'-TGA(G/C)TCA-3 ' | Dimer |
| C/EBP | Cierre básico | 5'-ATTGCGCAAT-3 ' | Dimer |
| Factor de shock de calor | Cierre básico | 5'-XGAAX-3' | Trimer |
| ATF/CREB | Cierre básico | 5'-TGACGTCA-3 ' | Dimer |
| c-Myc | Helix-loop-helix básico | 5'-CACGTG-3' | Dimer |
| Oct-1 | Helix-turn-helix | 5'-ATGCAAAT-3' | Monomer |
| NF-1 | Novela | 5'-TTGGCXXXXXGCCAA-3 ' | Dimer |
| (G/C) = G o C X = A, T, G o C | |||
Funcional
Los factores de transcripción se han clasificado según su función reguladora:
- Yo. constitutivamente activa – presente en todas las células en todo momento – factores de transcripción general, Sp1, NF1, CCAAT
- II. condicionalmente activo – requiere activación
- II.A desarrollo (célula específica) – la expresión está controlada firmemente, pero, una vez expresada, no requiere ninguna activación adicional – GATA, HNF, PIT-1, MyoD, Myf5, Hox, Winged Helix
- II.B función de la señal – requiere señal externa para la activación
- II.B.1 ligando extracelular (endocrino o paracrina)-dependiente – receptores nucleares
- II.B.2 ligando intracelular (autocrina)-dependiente – activado por pequeñas moléculas intracelulares – SREBP, p53, receptores nucleares huérfanos
- II.B.3 Receptor de membrana celular dependiente – segundas cascadas de señalización de mensajero que resultan en la fosforilación del factor de transcripción
- II.B.3.a factores nucleares residentes – residir en el núcleo independientemente del estado de activación – CREB, AP-1, Mef2
- II.B.3.b factores citoplasmáticos latentes – la forma inactiva reside en el citoplasma, pero, cuando se activa, se translocaron en el núcleo – STAT, R-SMAD, NF-κB, Notch, TUBBY, NFAT
Estructurales
Los factores de transcripción a menudo se clasifican según la similitud de secuencia y, por lo tanto, la estructura terciaria de sus dominios de unión al ADN:
- 1 Superclase: Dominios básicos
- 1.1 Clase: Factores de cremallera Leucine (bZIP)
- 1.1.1 Familia: componentes AP-1 (como); incluye (c-Fos/c-Jun)
- 1.1.2 Familia: CREB
- 1.1.3 Familia: factores similares al C/EBP
- 1.1.4 Familia: bZIP / PAR
- 1.1.5 Familia: Factores de unión de la caja G de la planta
- 1.1.6 Familia: ZIP únicamente
- 1.2 Clase: Factores Helix-loop-helix (bHLH)
- 1.2.1 Familia: factores Ubiquitous (clase A)
- 1.2.2 Familia: Factores de transcripción miogénica (MyoD)
- 1.2.3 Familia: Achaete-Scute
- 1.2.4 Familia: Tal/Twist/Atonal/Hen
- 1.3 Clase: Helix-loop-helix / factores de cremallera de leucina (bHLH-ZIP)
- 1.3.1 Familia: Factores Ubiquitous bHLH-ZIP; incluye USF (USF1, USF2); SREBP (SREBP)
- 1.3.2 Familia: Factores de control del ciclo celular; incluye c-Myc
- 1.4 Clase: NF-1
- 1.4.1 Familia: NF-1 (A, B, C, X)
- 1,5 Clase: RF-X
- 1.5.1 Familia: RF-X (1, 2, 3, 4, 5, ANK)
- 1.6 Clase: bHSH
- 1.1 Clase: Factores de cremallera Leucine (bZIP)
- 2 Superclase: Dominios de unión de ADN que coordinan el zinc
- 2.1 Clase: Dedo cinc Cys4 del receptor nuclear
- 2.1.1 Familia: Receptores de hormonas esteroideas
- 2.1.2 Familia: Factores similares a los receptores de hormona tiroidea
- 2.2 Clase: diversos dedos Cys4 de zinc
- 2.2.1 Familia: GATA-Factors
- 2.3 Clase: Cis2 Su2 dominio de dedo de zinc
- 2.3.1 Familia: Factores Ubiquitos, incluye TFIIIA, Sp1
- 2.3.2 Familia: Reguladores de ciclo celular/desarrollo; incluye Krüppel
- 2.3.4 Familia: Grandes factores con propiedades vinculantes similares a NF-6B
- 2.4 Clase: Cys6 cysteine-zinc cluster
- 2.5 Clase: Dedos de zinc de composición alterna
- 2.1 Clase: Dedo cinc Cys4 del receptor nuclear
- 3 Superclass: Helix-turn-helix
- 3.1 Clase: Homeo dominio
- 3.1.1 Familia: Homeo dominio solamente; incluye Ubx
- 3.1.2 Familia: factores de dominio POU; incluye Oct
- 3.1.3 Familia: dominio Homeo con región LIM
- 3.1.4 Familia: dominio homeo más motivos de dedo de zinc
- 3.2 Clase: Caja de matrimonio
- 3.2.1 Familia: Pared más dominio homeo
- 3.2.2 Familia: Dominio pareado solamente
- 3.3 Clase: Cabeza de tenedor / helix alado
- 3.3.1 Familia: Reguladores de desarrollo; incluye cabeza de horquilla
- 3.3.2 Familia: Reguladores específicos del tejido
- 3.3.3 Familia: Factores de control del ciclo celular
- 3.3.0 Familia: Otros reguladores
- 3.4 Clase: Factores de choque de calor
- 3.4.1 Familia: FSM
- 3.5 Clase: Grupos triptófanos
- 3.5.1 Familia: Myb
- 3.5.2 Familia: tipo Ets
- 3.5.3 Familia: Factores reguladores de interferón
- 3.6 Clase: dominio TEA (factor de potenciador tradicional)
- 3.6.1 Familia: TEA (TEAD1, TEAD2, TEAD3, TEAD4)
- 3.1 Clase: Homeo dominio
- 4 Superclass: beta-Scaffold Factores con Contactos Menores
- 4.1 Clase: RHR (Rel homology region)
- 4.1.1 Familia: Rel/ankyrin; NF-kappaB
- 4.1.2 Familia: anquirin únicamente
- 4.1.3 Familia: NFATNuclear Factor de Acautivado T-cells) (NFATC1, NFATC2, NFATC3)
- 4.2 Clase: SITUACIÓN
- 4.2.1 Familia: SITU
- 4.3 Clase: p53
- 4.3.1 Familia: p53
- 4.4 Clase: caja MADS
- 4.4.1 Familia: Reguladores de diferenciación; incluye (Mef2)
- 4.4.2 Familia: Respuestas a las señales externas, SRF (factor de respuesta sérica) (SRF)
- 4.4.3 Familia: Reguladores metabólicos (ARG80)
- 4.5 Clase: factores de transcripción de beta-Barrel alpha-helix
- 4.6 Clase: Proteínas de unión TATA
- 4.6.1 Familia: TBP
- 4.7 Clase: buzón HMG
- 4.7.1 Familia: genes SOX, SRY
- 4.7.2 Familia: TCF-1 (TCF1)
- 4.7.3 Familia: HMG2, SSRP1
- 4.7.4 Familia: UBF
- 4.7.5 Familia: MATA
- 4.8 Clase: Factores de CCAAT heteroméricos
- 4.8.1 Familia: Factores heteroméricos del CCAAT
- 4.9 Clase: Grainyhead
- 4.9.1 Familia: Cabeza de parto
- 4.10 Clase: Factores de dominio de cuello frío
- 4.10.1 Familia: csd
- 4.11 Clase: Runt
- 4.11.1 Familia: Runt
- 4.1 Clase: RHR (Rel homology region)
- 0 Superclass: Otros factores de transcripción
- 0.1 Clase: Proteínas de puño de cobre
- 0,2 Clase: HMGI(Y) (HMGA1)
- 0.2.1 Familia: HMGI(Y)
- 0.3 Clase: Dominio de bolsillo
- 0,4 Clase: E1A-like factors
- 0.5 Clase: factores relacionados con AP2/EREBP
- 0,5.1 Familia: AP2
- 0,5.2 Familia: EREBP
- 0,5.3 Superfamilia: AP2/B3
- Familia: ARF
- 0,5.3.2 Familia: ABI
- 0,5.3.3 Familia: RAV
Contenido relacionado
Toronja
Félidos
Frailecillo