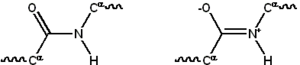Proteína

Proteínas são grandes biomoléculas e macromoléculas que compreendem uma ou mais longas cadeias de resíduos de aminoácidos. As proteínas desempenham uma vasta gama de funções dentro dos organismos, incluindo catalisar reações metabólicas, replicação do DNA, responder a estímulos, fornecer estrutura para células e organismos e transportar moléculas de um local para outro. As proteínas diferem umas das outras principalmente em sua sequência de aminoácidos, que é ditada pela sequência de nucleotídeos de seus genes e que geralmente resulta no dobramento da proteína em uma estrutura 3D específica que determina sua atividade.
Uma cadeia linear de resíduos de aminoácidos é chamada de polipeptídeo. Uma proteína contém pelo menos um longo polipeptídeo. Polipeptídeos curtos, contendo menos de 20 a 30 resíduos, raramente são considerados proteínas e são comumente chamados de peptídeos. Os resíduos de aminoácidos individuais são ligados por ligações peptídicas e resíduos de aminoácidos adjacentes. A sequência de resíduos de aminoácidos em uma proteína é definida pela sequência de um gene, que é codificada no código genético. Em geral, o código genético especifica 20 aminoácidos padrão; mas em certos organismos o código genético pode incluir selenocisteína e - em certas archaea - pirrolisina. Pouco depois ou mesmo durante a síntese, os resíduos de uma proteína são frequentemente modificados quimicamente por modificação pós-traducional, que altera as propriedades físicas e químicas, dobramento, estabilidade, atividade e, finalmente, a função das proteínas. Algumas proteínas possuem grupos não peptídicos ligados, que podem ser chamados de grupos prostéticos ou cofatores. As proteínas também podem trabalhar juntas para atingir uma função específica e, muitas vezes, se associam para formar complexos proteicos estáveis.
Uma vez formadas, as proteínas existem apenas por um determinado período e são então degradadas e recicladas pela maquinaria da célula através do processo de renovação de proteínas. O tempo de vida de uma proteína é medido em termos de sua meia-vida e abrange uma ampla gama. Eles podem existir por minutos ou anos com uma vida útil média de 1 a 2 dias em células de mamíferos. Proteínas anormais ou mal dobradas são degradadas mais rapidamente por serem alvo de destruição ou por serem instáveis.
Assim como outras macromoléculas biológicas, como polissacarídeos e ácidos nucléicos, as proteínas são partes essenciais dos organismos e participam de praticamente todos os processos dentro das células. Muitas proteínas são enzimas que catalisam reações bioquímicas e são vitais para o metabolismo. As proteínas também têm funções estruturais ou mecânicas, como a actina e a miosina no músculo e as proteínas no citoesqueleto, que formam um sistema de andaimes que mantém a forma da célula. Outras proteínas são importantes na sinalização celular, nas respostas imunes, na adesão celular e no ciclo celular. Nos animais, as proteínas são necessárias na dieta para fornecer os aminoácidos essenciais que não podem ser sintetizados. A digestão quebra as proteínas para uso metabólico.
As proteínas podem ser purificadas a partir de outros componentes celulares usando uma variedade de técnicas, como ultracentrifugação, precipitação, eletroforese e cromatografia; o advento da engenharia genética tornou possível uma série de métodos para facilitar a purificação. Os métodos comumente usados para estudar a estrutura e a função da proteína incluem imuno-histoquímica, mutagênese direcionada ao local, cristalografia de raios-X, ressonância magnética nuclear e espectrometria de massa.
História e etimologia
As proteínas foram reconhecidas como uma classe distinta de moléculas biológicas no século XVIII por Antoine Fourcroy e outros, distinguidas pelas propriedades das moléculas. capacidade de coagular ou flocular sob tratamentos com calor ou ácido. Exemplos notáveis na época incluíam albumina de clara de ovo, albumina de soro sanguíneo, fibrina e glúten de trigo.
As proteínas foram descritas pela primeira vez pelo químico holandês Gerardus Johannes Mulder e nomeadas pelo químico sueco Jöns Jacob Berzelius em 1838. Mulder realizou análises elementares de proteínas comuns e descobriu que quase todas as proteínas tinham a mesma fórmula empírica, C 400H620N100O120P1S1. Ele chegou à conclusão errônea de que eles poderiam ser compostos de um único tipo de molécula (muito grande). O termo "proteína" para descrever essas moléculas foi proposto pelo associado de Mulder, Berzelius; proteína é derivada da palavra grega πρώτειος (proteios), que significa "primário", "na liderança" ou "parado na frente", + -in. Mulder passou a identificar os produtos da degradação de proteínas, como o aminoácido leucina, para o qual encontrou um peso molecular (quase correto) de 131 Da. Antes de "proteína", outros nomes eram usados, como "albuminas" ou "materiais albuminosos" (Eiweisskörper, em alemão).
Os primeiros cientistas nutricionais, como o alemão Carl von Voit, acreditavam que a proteína era o nutriente mais importante para manter a estrutura do corpo, porque geralmente se acreditava que "a carne faz a carne." Karl Heinrich Ritthausen ampliou formas de proteínas conhecidas com a identificação do ácido glutâmico. Na Estação Experimental Agrícola de Connecticut, uma revisão detalhada das proteínas vegetais foi compilada por Thomas Burr Osborne. Trabalhando com Lafayette Mendel e aplicando a lei do mínimo de Liebig na alimentação de ratos de laboratório, os aminoácidos nutricionalmente essenciais foram estabelecidos. O trabalho foi continuado e comunicado por William Cumming Rose. A compreensão das proteínas como polipeptídeos veio através do trabalho de Franz Hofmeister e Hermann Emil Fischer em 1902. O papel central das proteínas como enzimas nos organismos vivos não foi totalmente apreciado até 1926, quando James B. Sumner mostrou que a enzima urease era de fato uma proteína.
A dificuldade em purificar proteínas em grandes quantidades tornou-as muito difíceis para os primeiros bioquímicos de proteínas estudarem. Assim, os primeiros estudos se concentraram em proteínas que poderiam ser purificadas em grandes quantidades, por exemplo, as de sangue, clara de ovo, várias toxinas e enzimas digestivas/metabólicas obtidas de matadouros. Na década de 1950, a Armor Hot Dog Co. purificou 1 kg de ribonuclease A pancreática bovina pura e a disponibilizou gratuitamente para os cientistas; esse gesto ajudou a ribonuclease A a se tornar um alvo importante para estudos bioquímicos nas décadas seguintes.
Linus Pauling é creditado com a previsão bem-sucedida de estruturas secundárias de proteínas regulares baseadas em pontes de hidrogênio, uma ideia apresentada pela primeira vez por William Astbury em 1933. Trabalho posterior de Walter Kauzmann sobre desnaturação, baseado em parte em estudos anteriores de Kaj Linderstrøm-Lang, contribuiu para a compreensão do enovelamento e estrutura de proteínas mediadas por interações hidrofóbicas.
A primeira proteína a ser sequenciada foi a insulina, por Frederick Sanger, em 1949. Sanger determinou corretamente a sequência de aminoácidos da insulina, demonstrando assim conclusivamente que as proteínas consistiam em polímeros lineares de aminoácidos em vez de cadeias ramificadas, colóides ou ciclois. Ele ganhou o Prêmio Nobel por essa conquista em 1958.
Com o desenvolvimento da cristalografia de raios X, tornou-se possível sequenciar estruturas de proteínas. As primeiras estruturas proteicas a serem resolvidas foram a hemoglobina por Max Perutz e a mioglobina por John Kendrew, em 1958. O uso de computadores e o aumento do poder computacional também apoiaram o sequenciamento de proteínas complexas. Em 1999, Roger Kornberg conseguiu sequenciar a estrutura altamente complexa da RNA polimerase usando raios X de alta intensidade de síncrotrons.
Desde então, a microscopia crioeletrônica (crio-EM) de grandes conjuntos macromoleculares foi desenvolvida. O Cryo-EM usa amostras de proteínas congeladas em vez de cristais e feixes de elétrons em vez de raios-x. Causa menos danos à amostra, permitindo que os cientistas obtenham mais informações e analisem estruturas maiores. A previsão computacional da estrutura proteica de pequenos domínios proteicos também ajudou os pesquisadores a abordar a resolução em nível atômico das estruturas proteicas. A partir de 2017, o Protein Data Bank possui mais de 126.060 estruturas de proteínas com resolução atômica.
Número de proteínas codificadas em genomas
O número de proteínas codificadas em um genoma corresponde aproximadamente ao número de genes (embora possa haver um número significativo de genes que codificam o RNA da proteína, por exemplo, RNAs ribossomais). Os vírus normalmente codificam de algumas a algumas centenas de proteínas, archaea e bactérias de algumas centenas a alguns milhares, enquanto os eucariotos geralmente codificam de alguns milhares a dezenas de milhares de proteínas (consulte o tamanho do genoma para obter uma lista de exemplos).
Bioquímica
A maioria das proteínas consiste em polímeros lineares construídos a partir de séries de até 20 L-α-aminoácidos diferentes. Todos os aminoácidos proteinogênicos possuem características estruturais comuns, incluindo um carbono α ao qual estão ligados um grupo amino, um grupo carboxila e uma cadeia lateral variável. Apenas a prolina difere dessa estrutura básica, pois contém um anel incomum para o grupo amina N-end, que força a porção CO-NH amida a uma conformação fixa. As cadeias laterais dos aminoácidos padrão, detalhadas na lista de aminoácidos padrão, possuem uma grande variedade de estruturas e propriedades químicas; é o efeito combinado de todas as cadeias laterais de aminoácidos em uma proteína que, em última análise, determina sua estrutura tridimensional e sua reatividade química. Os aminoácidos em uma cadeia polipeptídica são ligados por ligações peptídicas. Uma vez ligado na cadeia de proteína, um aminoácido individual é chamado de resíduo, e a série ligada de átomos de carbono, nitrogênio e oxigênio é conhecida como cadeia principal ou estrutura proteica.
A ligação peptídica tem duas formas de ressonância que contribuem com algum caráter de ligação dupla e inibem a rotação em torno de seu eixo, de modo que os carbonos alfa são aproximadamente coplanares. Os outros dois ângulos diedros na ligação peptídica determinam a forma local assumida pelo esqueleto da proteína. A extremidade com um grupo amino livre é conhecida como N-terminal ou aminoterminal, enquanto a extremidade da proteína com um grupo carboxila livre é conhecida como C-terminal ou carboxiterminal (a sequência da proteína é escrita de N- terminal para C-terminal, da esquerda para a direita).
As palavras proteína, polipeptídeo e peptídeo são um pouco ambíguas e podem se sobrepor em significado. Proteína é geralmente usado para se referir à molécula biológica completa em uma conformação estável, enquanto peptídeo é geralmente reservado para oligômeros de aminoácidos curtos, muitas vezes sem uma estrutura 3D estável. Mas o limite entre os dois não é bem definido e geralmente fica próximo de 20 a 30 resíduos. Polipeptídeo pode se referir a qualquer cadeia linear única de aminoácidos, geralmente independentemente do comprimento, mas geralmente implica na ausência de uma conformação definida.
Interações
As proteínas podem interagir com muitos tipos de moléculas, inclusive com outras proteínas, com lipídios, com carboidratos e com o DNA.
Abundância nas células
Estima-se que bactérias de tamanho médio contenham cerca de 2 milhões de proteínas por célula (por exemplo, E. coli e Staphylococcus aureus). Bactérias menores, como Mycoplasma ou espiroquetas, contêm menos moléculas, da ordem de 50.000 a 1 milhão. Em contraste, as células eucarióticas são maiores e, portanto, contêm muito mais proteínas. Por exemplo, estima-se que células de levedura contenham cerca de 50 milhões de proteínas e células humanas da ordem de 1 a 3 bilhões. A concentração de cópias de proteínas individuais varia de algumas moléculas por célula até 20 milhões. Nem todos os genes que codificam proteínas são expressos na maioria das células e seu número depende, por exemplo, do tipo de célula e de estímulos externos. Por exemplo, das cerca de 20.000 proteínas codificadas pelo genoma humano, apenas 6.000 são detectadas em células linfoblastóides.
Síntese
Biossíntese
As proteínas são montadas a partir de aminoácidos usando informações codificadas em genes. Cada proteína tem sua própria sequência única de aminoácidos que é especificada pela sequência de nucleotídeos do gene que codifica essa proteína. O código genético é um conjunto de conjuntos de três nucleotídeos chamados códons e cada combinação de três nucleotídeos designa um aminoácido, por exemplo AUG (adenina-uracil-guanina) é o código da metionina. Como o DNA contém quatro nucleotídeos, o número total de códons possíveis é 64; portanto, há alguma redundância no código genético, com alguns aminoácidos especificados por mais de um códon. Os genes codificados no DNA são primeiro transcritos em RNA pré-mensageiro (mRNA) por proteínas como a polimerase de RNA. A maioria dos organismos então processa o pré-mRNA (também conhecido como transcrito primário) usando várias formas de modificação pós-transcricional para formar o mRNA maduro, que é então usado como modelo para a síntese de proteínas pelo ribossomo. Em procariontes, o mRNA pode ser usado assim que é produzido ou ser ligado a um ribossomo após ter se afastado do nucleoide. Em contraste, os eucariotos produzem mRNA no núcleo da célula e depois o translocam através da membrana nuclear para o citoplasma, onde ocorre a síntese de proteínas. A taxa de síntese de proteínas é maior em procariotos do que em eucariotos e pode atingir até 20 aminoácidos por segundo.
O processo de síntese de uma proteína a partir de um molde de mRNA é conhecido como tradução. O mRNA é carregado no ribossomo e é lido três nucleotídeos de cada vez, combinando cada códon com seu anticódon de pareamento de bases localizado em uma molécula de RNA de transferência, que carrega o aminoácido correspondente ao códon que reconhece. A enzima aminoacil tRNA sintetase "carrega" as moléculas de tRNA com os aminoácidos corretos. O polipeptídeo em crescimento é geralmente chamado de cadeia nascente. As proteínas são sempre biossintetizadas do N-terminal para o C-terminal.
O tamanho de uma proteína sintetizada pode ser medido pelo número de aminoácidos que ela contém e por sua massa molecular total, que normalmente é expressa em unidades de daltons (sinônimo de unidades de massa atômica), ou a unidade derivada quilodalton (kDa). O tamanho médio de uma proteína aumenta de Archaea para Bacteria para Eukaryote (283, 311, 438 resíduos e 31, 34, 49 kDa respectivamente) devido a um maior número de domínios de proteína constituindo proteínas em organismos superiores. Por exemplo, as proteínas de levedura têm em média 466 aminoácidos de comprimento e 53 kDa em massa. As maiores proteínas conhecidas são as titinas, um componente do sarcômero muscular, com massa molecular de quase 3.000 kDa e comprimento total de quase 27.000 aminoácidos.
Síntese química
Proteínas curtas também podem ser sintetizadas quimicamente por uma família de métodos conhecidos como síntese de peptídeos, que se baseiam em técnicas de síntese orgânica, como a ligação química para produzir peptídeos com alto rendimento. A síntese química permite a introdução de aminoácidos não naturais em cadeias polipeptídicas, como a ligação de sondas fluorescentes a cadeias laterais de aminoácidos. Esses métodos são úteis em bioquímica de laboratório e biologia celular, embora geralmente não sejam para aplicações comerciais. A síntese química é ineficiente para polipeptídeos com mais de 300 aminoácidos, e as proteínas sintetizadas podem não assumir prontamente sua estrutura terciária nativa. A maioria dos métodos de síntese química procede do terminal C para o terminal N, oposto à reação biológica.
Estrutura

A maioria das proteínas se dobra em estruturas 3D únicas. A forma na qual uma proteína se dobra naturalmente é conhecida como sua conformação nativa. Embora muitas proteínas possam se enovelar sem ajuda, simplesmente por meio das propriedades químicas de seus aminoácidos, outras requerem a ajuda de chaperonas moleculares para se enovelar em seus estados nativos. Os bioquímicos geralmente se referem a quatro aspectos distintos da estrutura de uma proteína:
- Estrutura primária: a sequência de aminoácidos. Uma proteína é um poliamida.
- Estrutura secundária: repetir regularmente as estruturas locais estabilizadas por ligações de hidrogénio. Os exemplos mais comuns são o α-helix, β-sheet e voltas. Como as estruturas secundárias são locais, muitas regiões de diferentes estruturas secundárias podem estar presentes na mesma molécula de proteína.
- Estrutura terciária: a forma geral de uma única molécula proteica; a relação espacial das estruturas secundárias uns aos outros. A estrutura terciária é geralmente estabilizada por interações não-locais, mais comumente a formação de um núcleo hidrofóbico, mas também através de pontes de sal, ligações de hidrogênio, ligações de desulfeto e até mesmo modificações pós-translacionais. O termo "estrutura terciária" é frequentemente usado como sinônimo do termo fold. A estrutura terciária é o que controla a função básica da proteína.
- Estrutura quaternária: a estrutura formada por várias moléculas de proteína (correntes de polipeptídeo), geralmente chamada subunidades de proteína neste contexto, que funcionam como um único complexo proteico.
- Estrutura quinquenal: as assinaturas da superfície da proteína que organizam o interior celular lotado. A estrutura quinária é dependente de interações transientes, mas essenciais, macromoleculares que ocorrem dentro das células vivas.
As proteínas não são moléculas totalmente rígidas. Além desses níveis de estrutura, as proteínas podem alternar entre várias estruturas relacionadas enquanto desempenham suas funções. No contexto desses rearranjos funcionais, essas estruturas terciárias ou quaternárias são geralmente chamadas de "conformações", e as transições entre elas são chamadas de mudanças conformacionais. Tais mudanças são frequentemente induzidas pelo ligação de uma molécula de substrato ao sítio ativo de uma enzima, ou região física da proteína que participa da catálise química. Em solução, as proteínas também sofrem variação na estrutura por meio de vibração térmica e colisão com outras moléculas.
As proteínas podem ser informalmente divididas em três classes principais, que se correlacionam com estruturas terciárias típicas: proteínas globulares, proteínas fibrosas e proteínas de membrana. Quase todas as proteínas globulares são solúveis e muitas são enzimas. As proteínas fibrosas são frequentemente estruturais, como o colágeno, o principal componente do tecido conjuntivo, ou a queratina, o componente proteico do cabelo e das unhas. As proteínas de membrana geralmente servem como receptores ou fornecem canais para moléculas polares ou carregadas passarem pela membrana celular.
Um caso especial de pontes de hidrogênio intramoleculares dentro das proteínas, mal protegidas do ataque da água e, portanto, promovendo sua própria desidratação, são chamadas de dehidrons.
Domínios de proteína
Muitas proteínas são compostas por vários domínios proteicos, ou seja, segmentos de uma proteína que se dobram em unidades estruturais distintas. Os domínios geralmente também têm funções específicas, como atividades enzimáticas (por exemplo, quinase) ou servem como módulos de ligação (por exemplo, o domínio SH3 se liga a sequências ricas em prolina em outras proteínas).
Motivo de sequência
Sequências curtas de aminoácidos dentro de proteínas geralmente atuam como locais de reconhecimento para outras proteínas. Por exemplo, os domínios SH3 normalmente se ligam a motivos PxxP curtos (ou seja, 2 prolinas [P], separados por dois aminoácidos não especificados [x], embora os aminoácidos circundantes possam determinar a especificidade de ligação exata). Muitos desses motivos foram coletados no banco de dados Eukaryotic Linear Motif (ELM).
Topologia de proteína
A topologia de uma proteína descreve o emaranhado da espinha dorsal e o arranjo dos contatos dentro da cadeia dobrada. Duas estruturas teóricas de teoria de nós e topologia de circuito foram aplicadas para caracterizar a topologia de proteínas. Ser capaz de descrever a topologia de proteínas abre novos caminhos para a engenharia de proteínas e o desenvolvimento farmacêutico e aumenta nossa compreensão de doenças de dobramento incorreto de proteínas, como distúrbios neuromusculares e câncer.
Funções celulares
As proteínas são os principais atores dentro da célula, supostamente realizando as funções especificadas pela informação codificada nos genes. Com exceção de certos tipos de RNA, a maioria das outras moléculas biológicas são elementos relativamente inertes sobre os quais as proteínas agem. As proteínas representam metade do peso seco de uma célula de Escherichia coli, enquanto outras macromoléculas, como DNA e RNA, representam apenas 3% e 20%, respectivamente. O conjunto de proteínas expressas em uma determinada célula ou tipo celular é conhecido como seu proteoma.
A principal característica das proteínas que também permite seu conjunto diversificado de funções é sua capacidade de se ligar a outras moléculas de maneira específica e firme. A região da proteína responsável pela ligação de outra molécula é conhecida como sítio de ligação e geralmente é uma depressão ou "bolsão" na superfície molecular. Essa capacidade de ligação é mediada pela estrutura terciária da proteína, que define o local de ligação, e pelas propriedades químicas dos aminoácidos circundantes. correntes laterais. A ligação às proteínas pode ser extraordinariamente rígida e específica; por exemplo, a proteína inibidora da ribonuclease se liga à angiogenina humana com uma constante de dissociação subfemtomolar (<10−15 M), mas não se liga de forma alguma à sua onconase anfíbia homóloga (>1 M). Alterações químicas extremamente pequenas, como a adição de um único grupo metil a um parceiro de ligação, às vezes podem ser suficientes para quase eliminar a ligação; por exemplo, a aminoacil tRNA sintetase específica para o aminoácido valina discrimina a cadeia lateral muito semelhante do aminoácido isoleucina.
As proteínas podem se ligar a outras proteínas, bem como a substratos de moléculas pequenas. Quando as proteínas se ligam especificamente a outras cópias da mesma molécula, elas podem se oligomerizar para formar fibrilas; esse processo ocorre frequentemente em proteínas estruturais que consistem em monômeros globulares que se auto-associam para formar fibras rígidas. As interações proteína-proteína também regulam a atividade enzimática, controlam a progressão ao longo do ciclo celular e permitem a montagem de grandes complexos proteicos que realizam muitas reações intimamente relacionadas com uma função biológica comum. As proteínas também podem se ligar ou até mesmo ser integradas às membranas celulares. A capacidade dos parceiros de ligação de induzir mudanças conformacionais nas proteínas permite a construção de redes de sinalização extremamente complexas. Como as interações entre proteínas são reversíveis e dependem fortemente da disponibilidade de diferentes grupos de proteínas parceiras para formar agregados capazes de realizar conjuntos discretos de funções, o estudo das interações entre proteínas específicas é a chave para entender aspectos importantes da função celular e, finalmente, as propriedades que distinguem determinados tipos de células.
Enzimas
O papel mais conhecido das proteínas na célula é como enzimas, que catalisam reações químicas. As enzimas geralmente são altamente específicas e aceleram apenas uma ou algumas reações químicas. As enzimas realizam a maioria das reações envolvidas no metabolismo, bem como manipulam o DNA em processos como a replicação do DNA, o reparo do DNA e a transcrição. Algumas enzimas atuam sobre outras proteínas para adicionar ou remover grupos químicos em um processo conhecido como modificação pós-traducional. Cerca de 4.000 reações são conhecidas por serem catalisadas por enzimas. A taxa de aceleração conferida pela catálise enzimática é muitas vezes enorme - até 1017- aumento na taxa em relação à reação não catalisada no caso da orotato descarboxilase (78 milhões de anos sem a enzima, 18 milissegundos com a enzima).
As moléculas ligadas e submetidas à ação das enzimas são chamadas de substratos. Embora as enzimas possam consistir em centenas de aminoácidos, geralmente é apenas uma pequena fração dos resíduos que entra em contato com o substrato, e uma fração ainda menor – três a quatro resíduos em média – que estão diretamente envolvidos na catálise. A região da enzima que se liga ao substrato e contém os resíduos catalíticos é conhecida como sítio ativo.
As proteínas dirigentes são membros de uma classe de proteínas que ditam a estereoquímica de um composto sintetizado por outras enzimas.
Sinalização celular e ligação do ligante
Muitas proteínas estão envolvidas no processo de sinalização celular e transdução de sinal. Algumas proteínas, como a insulina, são proteínas extracelulares que transmitem um sinal da célula na qual foram sintetizadas para outras células em tecidos distantes. Outras são proteínas de membrana que atuam como receptores cuja principal função é ligar uma molécula sinalizadora e induzir uma resposta bioquímica na célula. Muitos receptores têm um sítio de ligação exposto na superfície celular e um domínio efetor dentro da célula, que pode ter atividade enzimática ou pode sofrer uma alteração conformacional detectada por outras proteínas dentro da célula.
Anticorpos são componentes proteicos de um sistema imunológico adaptativo cuja principal função é ligar antígenos, ou substâncias estranhas no corpo, e direcioná-los para destruição. Os anticorpos podem ser secretados no ambiente extracelular ou ancorados nas membranas de células B especializadas conhecidas como células plasmáticas. Enquanto as enzimas são limitadas em sua afinidade de ligação para seus substratos pela necessidade de conduzir sua reação, os anticorpos não têm tais restrições. A afinidade de ligação de um anticorpo ao seu alvo é extraordinariamente alta.
Muitas proteínas de transporte de ligantes se ligam a pequenas biomoléculas específicas e as transportam para outros locais no corpo de um organismo multicelular. Essas proteínas devem ter uma alta afinidade de ligação quando seu ligante está presente em altas concentrações, mas também devem liberar o ligante quando ele está presente em baixas concentrações nos tecidos-alvo. O exemplo canônico de uma proteína de ligação ao ligante é a hemoglobina, que transporta oxigênio dos pulmões para outros órgãos e tecidos em todos os vertebrados e tem homólogos próximos em todos os reinos biológicos. Lectinas são proteínas de ligação de açúcar que são altamente específicas para suas porções de açúcar. As lectinas normalmente desempenham um papel nos fenômenos de reconhecimento biológico envolvendo células e proteínas. Receptores e hormônios são proteínas de ligação altamente específicas.
As proteínas transmembrana também podem servir como proteínas de transporte de ligantes que alteram a permeabilidade da membrana celular a pequenas moléculas e íons. A membrana sozinha tem um núcleo hidrofóbico através do qual moléculas polares ou carregadas não podem se difundir. As proteínas de membrana contêm canais internos que permitem que essas moléculas entrem e saiam da célula. Muitas proteínas de canais iônicos são especializadas para selecionar apenas um determinado íon; por exemplo, os canais de potássio e sódio frequentemente discriminam apenas um dos dois íons.
Proteínas estruturais
As proteínas estruturais conferem rigidez e rigidez a componentes biológicos fluidos. A maioria das proteínas estruturais são proteínas fibrosas; por exemplo, o colágeno e a elastina são componentes críticos do tecido conjuntivo, como a cartilagem, e a queratina é encontrada em estruturas duras ou filamentosas, como cabelos, unhas, penas, cascos e algumas carapaças de animais. Algumas proteínas globulares também podem desempenhar funções estruturais, por exemplo, a actina e a tubulina são globulares e solúveis como monômeros, mas polimerizam para formar fibras longas e rígidas que compõem o citoesqueleto, o que permite que a célula mantenha sua forma e tamanho.
Outras proteínas que desempenham funções estruturais são as proteínas motoras, como miosina, cinesina e dineína, que são capazes de gerar forças mecânicas. Estas proteínas são cruciais para a motilidade celular de organismos unicelulares e o esperma de muitos organismos multicelulares que se reproduzem sexualmente. Eles também geram as forças exercidas pela contração dos músculos e desempenham papéis essenciais no transporte intracelular.
Evolução de proteínas
Uma questão chave na biologia molecular é como as proteínas evoluem, ou seja, como as mutações (ou melhor, mudanças na sequência de aminoácidos) levam a novas estruturas e funções? A maioria dos aminoácidos em uma proteína pode ser alterada sem interromper a atividade ou a função, como pode ser visto em várias proteínas homólogas entre as espécies (conforme coletado em bancos de dados especializados para famílias de proteínas, por exemplo, PFAM). Para evitar consequências dramáticas de mutações, um gene pode ser duplicado antes que possa sofrer mutações livremente. No entanto, isso também pode levar à perda completa da função do gene e, portanto, dos pseudogenes. Mais comumente, as alterações de um único aminoácido têm consequências limitadas, embora algumas possam alterar substancialmente a função da proteína, especialmente nas enzimas. Por exemplo, muitas enzimas podem alterar sua especificidade de substrato por uma ou algumas mutações. As alterações na especificidade do substrato são facilitadas pela promiscuidade do substrato, ou seja, a capacidade de muitas enzimas se ligarem e processarem vários substratos. Quando ocorrem mutações, a especificidade de uma enzima pode aumentar (ou diminuir) e, portanto, sua atividade enzimática. Assim, as bactérias (ou outros organismos) podem se adaptar a diferentes fontes de alimento, incluindo substratos não naturais, como o plástico.
Métodos de estudo
As atividades e estruturas das proteínas podem ser examinadas in vitro, in vivo e in silico. Estudos In vitro de proteínas purificadas em ambientes controlados são úteis para aprender como uma proteína desempenha sua função: por exemplo, estudos de cinética enzimática exploram o mecanismo químico de uma enzima's atividade catalítica e sua afinidade relativa para várias moléculas de substrato possíveis. Por outro lado, experimentos in vivo podem fornecer informações sobre o papel fisiológico de uma proteína no contexto de uma célula ou mesmo de um organismo inteiro. Estudos In silico usam métodos computacionais para estudar proteínas.
Purificação de proteínas
Para realizar a análise in vitro, uma proteína deve ser purificada de outros componentes celulares. Esse processo geralmente começa com a lise celular, na qual a membrana celular é rompida e seu conteúdo interno é liberado em uma solução conhecida como lisado bruto. A mistura resultante pode ser purificada por ultracentrifugação, que fraciona os vários componentes celulares em frações contendo proteínas solúveis; lipídios e proteínas de membrana; organelas celulares e ácidos nucléicos. A precipitação por um método conhecido como salting out pode concentrar as proteínas deste lisado. Vários tipos de cromatografia são então usados para isolar a proteína ou proteínas de interesse com base em propriedades como peso molecular, carga líquida e afinidade de ligação. O nível de purificação pode ser monitorado usando vários tipos de eletroforese em gel se o peso molecular e o ponto isoelétrico da proteína desejada forem conhecidos, por espectroscopia se a proteína tiver características espectroscópicas distinguíveis ou por ensaios enzimáticos se a proteína tiver atividade enzimática. Além disso, as proteínas podem ser isoladas de acordo com sua carga usando eletrofocalização.
Para proteínas naturais, uma série de etapas de purificação pode ser necessária para obter proteína suficientemente pura para aplicações laboratoriais. Para simplificar esse processo, a engenharia genética costuma ser usada para adicionar características químicas às proteínas que as tornam mais fáceis de purificar sem afetar sua estrutura ou atividade. Aqui, uma "tag" consistindo em uma sequência específica de aminoácidos, muitas vezes uma série de resíduos de histidina (um "His-tag"), está ligado a um terminal da proteína. Como resultado, quando o lisado é passado por uma coluna de cromatografia contendo níquel, os resíduos de histidina ligam o níquel e se ligam à coluna enquanto os componentes não marcados do lisado passam sem impedimentos. Várias marcas diferentes foram desenvolvidas para ajudar os pesquisadores a purificar proteínas específicas de misturas complexas.
Localização celular
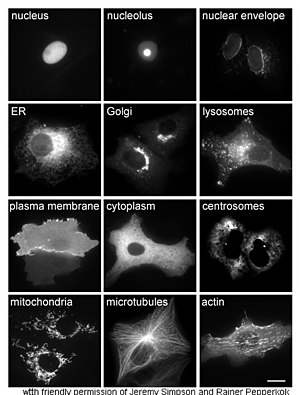
O estudo de proteínas in vivo geralmente se preocupa com a síntese e localização da proteína dentro da célula. Embora muitas proteínas intracelulares sejam sintetizadas no citoplasma e proteínas ligadas à membrana ou secretadas no retículo endoplasmático, as especificidades de como as proteínas são direcionadas para organelas ou estruturas celulares específicas geralmente não são claras. Uma técnica útil para avaliar a localização celular usa a engenharia genética para expressar em uma célula uma proteína de fusão ou quimera que consiste na proteína natural de interesse ligada a um "repórter" como a proteína verde fluorescente (GFP). A posição da proteína fundida dentro da célula pode ser visualizada de forma limpa e eficiente usando microscopia, conforme mostrado na figura ao lado.
Outros métodos para elucidar a localização celular de proteínas requerem o uso de marcadores compartimentais conhecidos para regiões como RE, Golgi, lisossomos ou vacúolos, mitocôndrias, cloroplastos, membrana plasmática, etc. desses marcadores ou de anticorpos para marcadores conhecidos, fica muito mais simples identificar a localização de uma proteína de interesse. Por exemplo, a imunofluorescência indireta permitirá a colocalização da fluorescência e a demonstração da localização. Corantes fluorescentes são usados para rotular compartimentos celulares para uma finalidade semelhante.
Também existem outras possibilidades. Por exemplo, a imuno-histoquímica geralmente usa um anticorpo para uma ou mais proteínas de interesse que são conjugadas a enzimas que produzem sinais luminescentes ou cromogênicos que podem ser comparados entre amostras, permitindo informações de localização. Outra técnica aplicável é o cofracionamento em gradientes de sacarose (ou outro material) usando centrifugação isopícnica. Embora essa técnica não comprove a colocalização de um compartimento de densidade conhecida e da proteína de interesse, ela aumenta a probabilidade e é mais passível de estudos em larga escala.
Finalmente, o método padrão-ouro de localização celular é a microscopia imunoeletrônica. Esta técnica também usa um anticorpo para a proteína de interesse, juntamente com técnicas clássicas de microscopia eletrônica. A amostra é preparada para exame de microscopia eletrônica normal, e então tratada com um anticorpo para a proteína de interesse que é conjugado a um material extremamente eletrodenso, geralmente ouro. Isso permite a localização de ambos os detalhes ultraestruturais, bem como a proteína de interesse.
Por meio de outra aplicação de engenharia genética conhecida como mutagênese direcionada ao local, os pesquisadores podem alterar a sequência da proteína e, portanto, sua estrutura, localização celular e suscetibilidade à regulação. Esta técnica ainda permite a incorporação de aminoácidos não naturais em proteínas, usando tRNAs modificados, e pode permitir o desenho racional de novas proteínas com novas propriedades.
Proteômica
O complemento total de proteínas presentes em uma célula ou tipo de célula é conhecido como seu proteoma, e o estudo de tais conjuntos de dados em larga escala define o campo da proteômica, nomeado por analogia com o campo relacionado da genômica. As principais técnicas experimentais em proteômica incluem eletroforese 2D, que permite a separação de muitas proteínas, espectrometria de massa, que permite a identificação rápida e de alto rendimento de proteínas e sequenciamento de peptídeos (mais frequentemente após a digestão em gel), microarranjos de proteínas, que permitem a detecção dos níveis relativos das várias proteínas presentes em uma célula, e triagem de dois híbridos, que permite a exploração sistemática de interações proteína-proteína. O complemento total de tais interações biologicamente possíveis é conhecido como interactoma. Uma tentativa sistemática de determinar as estruturas de proteínas que representam todas as dobras possíveis é conhecida como genômica estrutural.
Determinação da estrutura
Descobrir a estrutura terciária de uma proteína, ou a estrutura quaternária de seus complexos, pode fornecer pistas importantes sobre como a proteína desempenha sua função e como ela pode ser afetada, ou seja, no design de medicamentos. Como as proteínas são muito pequenas para serem vistas ao microscópio de luz, outros métodos devem ser empregados para determinar sua estrutura. Métodos experimentais comuns incluem cristalografia de raios-X e espectroscopia de RMN, os quais podem produzir informações estruturais em resolução atômica. No entanto, experimentos de RMN são capazes de fornecer informações a partir das quais um subconjunto de distâncias entre pares de átomos pode ser estimado, e as conformações finais possíveis para uma proteína são determinadas pela resolução de um problema de geometria de distância. A interferometria de dupla polarização é um método analítico quantitativo para medir a conformação geral da proteína e as alterações conformacionais devido a interações ou outros estímulos. O dicroísmo circular é outra técnica de laboratório para determinar a composição interna de folhas β / hélice α de proteínas. A microscopia crioeletrônica é usada para produzir informações estruturais de baixa resolução sobre complexos proteicos muito grandes, incluindo vírus montados; uma variante conhecida como cristalografia de elétrons também pode produzir informações de alta resolução em alguns casos, especialmente para cristais bidimensionais de proteínas de membrana. As estruturas resolvidas são geralmente depositadas no Protein Data Bank (PDB), um recurso disponível gratuitamente a partir do qual dados estruturais sobre milhares de proteínas podem ser obtidos na forma de coordenadas cartesianas para cada átomo na proteína.
São conhecidas muito mais sequências de genes do que estruturas de proteínas. Além disso, o conjunto de estruturas resolvidas é direcionado para proteínas que podem ser facilmente submetidas às condições exigidas na cristalografia de raios-X, um dos principais métodos de determinação de estrutura. Em particular, as proteínas globulares são comparativamente fáceis de cristalizar na preparação para a cristalografia de raios-X. Proteínas de membrana e grandes complexos de proteínas, por outro lado, são difíceis de cristalizar e estão sub-representados no PDB. Iniciativas de genômica estrutural tentaram remediar essas deficiências resolvendo sistematicamente estruturas representativas das principais classes de dobras. Os métodos de previsão da estrutura da proteína tentam fornecer um meio de gerar uma estrutura plausível para proteínas cujas estruturas não foram determinadas experimentalmente.
Previsão de estrutura
Complementar ao campo da genômica estrutural, a previsão da estrutura de proteínas desenvolve modelos matemáticos eficientes de proteínas para prever computacionalmente as formações moleculares em teoria, em vez de detectar estruturas com observação de laboratório. O tipo mais bem-sucedido de previsão de estrutura, conhecido como modelagem de homologia, depende da existência de um "template" estrutura com similaridade de sequência com a proteína sendo modelada; genômica estrutural' O objetivo é fornecer representação suficiente em estruturas resolvidas para modelar a maioria das que permanecem. Embora a produção de modelos precisos continue sendo um desafio quando apenas estruturas de modelo distantemente relacionadas estão disponíveis, foi sugerido que o alinhamento de sequência é o gargalo nesse processo, pois modelos bastante precisos podem ser produzidos se um modelo "perfeito" o alinhamento da sequência é conhecido. Muitos métodos de previsão de estrutura serviram para informar o campo emergente da engenharia de proteínas, no qual novas dobras de proteínas já foram projetadas. Além disso, as proteínas (em eucariotos ~33%) contêm grandes segmentos não estruturados, mas biologicamente funcionais, e podem ser classificadas como proteínas intrinsecamente desordenadas. Prever e analisar o distúrbio da proteína é, portanto, uma parte importante da caracterização da estrutura da proteína.
Bioinformática
Uma vasta gama de métodos computacionais foi desenvolvida para analisar a estrutura, função e evolução das proteínas. O desenvolvimento de tais ferramentas foi impulsionado pela grande quantidade de dados genômicos e proteômicos disponíveis para uma variedade de organismos, incluindo o genoma humano. É simplesmente impossível estudar todas as proteínas experimentalmente, portanto, apenas algumas são submetidas a experimentos de laboratório enquanto ferramentas computacionais são usadas para extrapolar para proteínas semelhantes. Tais proteínas homólogas podem ser identificadas eficientemente em organismos distantemente relacionados por alinhamento de sequência. As sequências do genoma e do gene podem ser pesquisadas por uma variedade de ferramentas para determinadas propriedades. As ferramentas de perfil de sequência podem encontrar sítios de enzimas de restrição, quadros de leitura abertos em sequências de nucleotídeos e prever estruturas secundárias. Árvores filogenéticas podem ser construídas e hipóteses evolutivas desenvolvidas usando software especial como ClustalW em relação à ancestralidade de organismos modernos e os genes que eles expressam. O campo da bioinformática é hoje indispensável para a análise de genes e proteínas.
Simulação in silico de processos dinâmicos
Um problema computacional mais complexo é a previsão de interações intermoleculares, como no acoplamento molecular, dobramento de proteínas, interação proteína-proteína e reatividade química. Modelos matemáticos para simular esses processos dinâmicos envolvem a mecânica molecular, em particular a dinâmica molecular. A esse respeito, simulações in silico descobriram o dobramento de pequenos domínios de proteínas α-helicoidais, como o headpiece do vilão, a proteína acessória do HIV e métodos híbridos que combinam dinâmica molecular padrão com matemática da mecânica quântica exploraram os estados eletrônicos de rodopsinas.
Além da dinâmica molecular clássica, os métodos de dinâmica quântica permitem a simulação de proteínas em detalhes atomísticos com uma descrição precisa dos efeitos da mecânica quântica. Os exemplos incluem o método Hartree dependente do tempo de configuração múltipla multicamada (MCTDH) e a abordagem de equações hierárquicas de movimento (HEOM), que foram aplicadas a criptocromos de plantas e complexos de colheita de luz de bactérias, respectivamente. As simulações de mecânica quântica e clássica de sistemas em escala biológica são extremamente exigentes computacionalmente, portanto, as iniciativas de computação distribuída (por exemplo, o projeto Folding@home) facilitam a modelagem molecular explorando os avanços no processamento paralelo de GPU e as técnicas de Monte Carlo.
Análise química
O conteúdo total de nitrogênio da matéria orgânica é formado principalmente pelos grupos amino nas proteínas. O Nitrogênio Total Kjeldahl (TKN) é uma medida de nitrogênio amplamente utilizada na análise de água (residual), solo, alimentos, rações e matéria orgânica em geral. Como o nome sugere, o método Kjeldahl é aplicado. Métodos mais sensíveis estão disponíveis.
Nutrição
A maioria dos microorganismos e plantas podem biossintetizar todos os 20 aminoácidos padrão, enquanto os animais (incluindo humanos) devem obter alguns dos aminoácidos da dieta. Os aminoácidos que um organismo não consegue sintetizar por conta própria são referidos como aminoácidos essenciais. As principais enzimas que sintetizam certos aminoácidos não estão presentes em animais, como a aspartoquinase, que catalisa a primeira etapa na síntese de lisina, metionina e treonina a partir do aspartato. Se os aminoácidos estiverem presentes no ambiente, os microorganismos podem conservar energia absorvendo os aminoácidos de seus arredores e regulando negativamente suas vias biossintéticas.
Nos animais, os aminoácidos são obtidos através do consumo de alimentos que contêm proteínas. As proteínas ingeridas são então decompostas em aminoácidos através da digestão, que normalmente envolve a desnaturação da proteína através da exposição ao ácido e hidrólise por enzimas chamadas proteases. Alguns aminoácidos ingeridos são usados para a biossíntese de proteínas, enquanto outros são convertidos em glicose por meio da gliconeogênese ou inseridos no ciclo do ácido cítrico. Esse uso de proteína como combustível é particularmente importante em condições de fome, pois permite que as próprias proteínas do corpo sejam usadas para sustentar a vida, principalmente aquelas encontradas nos músculos.
Em animais como cães e gatos, a proteína mantém a saúde e a qualidade da pele, promovendo o crescimento e a queratinização do folículo piloso, reduzindo assim a probabilidade de problemas de pele que produzem maus odores. Proteínas de baixa qualidade também desempenham um papel na saúde gastrointestinal, aumentando o potencial de flatulência e compostos odoríferos em cães, porque quando as proteínas atingem o cólon em um estado não digerido, elas são fermentadas produzindo gás sulfídrico, indol e escatol. Cães e gatos digerem melhor as proteínas animais do que as vegetais, mas os produtos de origem animal de baixa qualidade são mal digeridos, incluindo pele, penas e tecido conjuntivo.
Contenido relacionado
Ciência agrícola
Colágeno
Combustão espontânea (desambiguação)
Cardiologia
Polimorfismo