Proteassoma
Proteassomas são complexos proteicos que degradam proteínas desnecessárias ou danificadas por proteólise, uma reação química que quebra as ligações peptídicas. As enzimas que ajudam nessas reações são chamadas de proteases.
Os proteassomas fazem parte de um mecanismo principal pelo qual as células regulam a concentração de proteínas específicas e degradam proteínas mal dobradas. As proteínas são marcadas para degradação com uma pequena proteína chamada ubiquitina. A reação de marcação é catalisada por enzimas chamadas ubiquitina ligases. Uma vez que uma proteína é marcada com uma única molécula de ubiquitina, isso é um sinal para outras ligases anexarem moléculas adicionais de ubiquitina. O resultado é uma cadeia de poliubiquitina que é ligada ao proteassoma, permitindo que ele degrade a proteína marcada. O processo de degradação produz peptídeos com cerca de sete a oito aminoácidos de comprimento, que podem então ser degradados em sequências de aminoácidos mais curtas e usados na síntese de novas proteínas.
Proteasomes são encontrados dentro de todos os eucariontes e archaea, e em algumas bactérias. Nos eucariotos, os proteassomas estão localizados tanto no núcleo quanto no citoplasma.
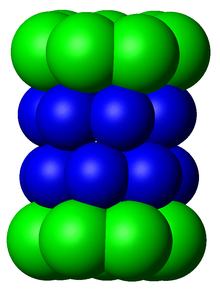
Na estrutura, o proteassoma é um complexo cilíndrico contendo um "núcleo" de quatro anéis empilhados formando um poro central. Cada anel é composto por sete proteínas individuais. Os dois anéis internos são feitos de sete subunidades β que contêm de três a sete sítios ativos de protease. Esses sítios estão localizados na superfície interna dos anéis, de modo que a proteína-alvo deve entrar no poro central antes de ser degradada. Cada um dos dois anéis externos contém sete subunidades α cuja função é manter um "portão" através do qual as proteínas entram no barril. Essas subunidades α são controladas pela ligação a "cap" estruturas ou partículas reguladoras que reconhecem marcadores de poliubiquitina ligados a substratos proteicos e iniciam o processo de degradação. O sistema geral de ubiquitinação e degradação proteassômica é conhecido como sistema ubiquitina-proteassoma.
A via de degradação proteossomal é essencial para muitos processos celulares, incluindo o ciclo celular, a regulação da expressão gênica e as respostas ao estresse oxidativo. A importância da degradação proteolítica dentro das células e o papel da ubiquitina nas vias proteolíticas foi reconhecida na premiação do Prêmio Nobel de Química de 2004 para Aaron Ciechanover, Avram Hershko e Irwin Rose.
Descoberta
Antes da descoberta do sistema ubiquitina-proteassoma, pensava-se que a degradação de proteínas nas células dependia principalmente de lisossomos, organelas ligadas à membrana com interiores ácidos e cheios de protease que podem degradar e depois reciclar proteínas exógenas e organelas envelhecidas ou danificadas. No entanto, o trabalho de Joseph Etlinger e Alfred L. Goldberg em 1977 sobre a degradação de proteínas dependente de ATP em reticulócitos, que não possuem lisossomos, sugeriu a presença de um segundo mecanismo de degradação intracelular. Isso foi demonstrado em 1978 como sendo composto de várias cadeias de proteínas distintas, uma novidade entre as proteases da época. Trabalhos posteriores sobre modificação de histonas levaram à identificação de uma modificação covalente inesperada da proteína histona por uma ligação entre uma cadeia lateral de lisina da histona e o resíduo de glicina C-terminal da ubiquitina, uma proteína que não tinha função conhecida. Descobriu-se então que uma proteína previamente identificada associada à degradação proteolítica, conhecida como fator de proteólise dependente de ATP 1 (APF-1), era a mesma proteína da ubiquitina. As atividades proteolíticas deste sistema foram isoladas como um complexo multiproteico originalmente chamado de complexo proteinase multicatalítico por Sherwin Wilk e Marion Orlowski. Mais tarde, o complexo proteolítico dependente de ATP, responsável pela degradação proteica dependente de ubiquitina, foi descoberto e foi denominado proteassoma 26S.
Grande parte do trabalho inicial que levou à descoberta do sistema proteassoma da ubiquitina ocorreu no final dos anos 1970 e início dos anos 1980 no Technion, no laboratório de Avram Hershko, onde Aaron Ciechanover trabalhou como estudante de pós-graduação. O ano sabático de Hershko no laboratório de Irwin Rose no Fox Chase Cancer Center forneceu insights conceituais importantes, embora Rose posteriormente tenha minimizado seu papel na descoberta. Os três dividiram o Prêmio Nobel de Química de 2004 por seu trabalho na descoberta desse sistema.
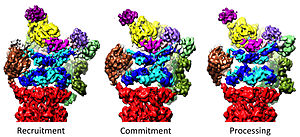
Embora os dados de microscopia eletrônica revelando a estrutura de anéis empilhados do proteassoma tenham se tornado disponíveis em meados da década de 1980, a primeira estrutura da partícula central do proteassoma não foi resolvida por cristalografia de raios-X até 1994. Em 2018, as primeiras estruturas atômicas da holoenzima do proteassoma 26S humano em complexo com um substrato proteico poliubiquitilado foram resolvidos por microscopia eletrônica criogênica, revelando mecanismos pelos quais o substrato é reconhecido, deubiquitilado, desenovelado e degradado pelo proteassoma 26S humano.
Estrutura e organização
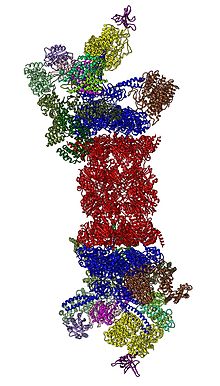
Os subcomponentes do proteassoma são muitas vezes referidos pelo seu coeficiente de sedimentação de Svedberg (denotado S). O proteassoma mais usado exclusivamente em mamíferos é o proteassoma 26S citosólico, que tem cerca de 2000 kilodaltons (kDa) em massa molecular contendo uma subunidade de proteína 20S e duas subunidades cap regulatórias 19S. O núcleo é oco e fornece uma cavidade fechada na qual as proteínas são degradadas; aberturas nas duas extremidades do núcleo permitem a entrada da proteína-alvo. Cada extremidade da partícula central associa-se a uma subunidade reguladora 19S que contém vários locais ativos de ATPase e locais de ligação de ubiquitina; é esta estrutura que reconhece as proteínas poliubiquitinadas e as transfere para o núcleo catalítico. Uma forma alternativa de subunidade reguladora chamada partícula 11S pode se associar ao núcleo essencialmente da mesma maneira que a partícula 19S; o 11S pode desempenhar um papel na degradação de peptídeos estranhos, como os produzidos após a infecção por um vírus.
Partícula de núcleo 20S
O número e a diversidade de subunidades contidas na partícula central 20S dependem do organismo; o número de subunidades distintas e especializadas é maior em organismos multicelulares do que em organismos unicelulares e maior em eucariotos do que em procariotos. Todas as partículas 20S consistem em quatro estruturas de anéis heptaméricas empilhadas que são compostas por dois tipos diferentes de subunidades; As subunidades α são de natureza estrutural, enquanto as subunidades β são predominantemente catalíticas. As subunidades α são pseudoenzimas homólogas às subunidades β. Eles são montados com seus terminais N adjacentes aos das subunidades β. Os dois anéis externos na pilha consistem em sete subunidades α cada, que servem como domínios de ancoragem para as partículas reguladoras e as subunidades alfa N-termini (Pfam PF10584) formam uma porta que bloqueia o acesso desregulado de substratos à cavidade interna. Os dois anéis internos consistem cada um em sete subunidades β e em seus terminais N contêm os sítios ativos de protease que realizam as reações de proteólise. Três atividades catalíticas distintas foram identificadas no complexo purificado: quimotripsina-símile, tripsina-símile e hidrólise de peptidilglutamil-peptídeo. O tamanho do proteassoma é relativamente conservado e é de cerca de 150 angstroms (Å) por 115 Å. A câmara interna tem no máximo 53 Å de largura, embora a entrada possa ser tão estreita quanto 13 Å, sugerindo que as proteínas do substrato devem ser pelo menos parcialmente desdobradas para entrar.
Em archaea, como Thermoplasma acidophilum, todas as subunidades α e β são idênticas, enquanto proteassomas eucarióticas, como as da levedura, contêm sete tipos distintos de cada subunidade. Nos mamíferos, as subunidades β1, β2 e β5 são catalíticas; embora eles compartilhem um mecanismo comum, eles têm três especificidades de substrato distintas consideradas semelhantes a quimotripsina, semelhantes a tripsina e hidrólise de peptídeos peptidil-glutamil (PHGH). Formas β alternativas denotadas β1i, β2i e β5i podem ser expressas em células hematopoiéticas em resposta à exposição a sinais pró-inflamatórios, como citocinas, em particular, interferon gama. O proteassoma montado com essas subunidades alternativas é conhecido como imunoproteassoma, cuja especificidade de substrato é alterada em relação ao proteassoma normal. Recentemente, um proteassoma alternativo foi identificado em células humanas que não possuem a subunidade central α3. Esses proteassomas (conhecidos como proteassomas α4-α4) formam partículas centrais 20S contendo uma subunidade α4 adicional no lugar da subunidade α3 ausente. Essas alternativas 'α4-α4' a existência de proteassomas foi previamente conhecida em leveduras. Embora a função precisa dessas isoformas de proteassoma ainda seja amplamente desconhecida, as células que expressam esses proteassomas apresentam maior resistência à toxicidade induzida por íons metálicos, como o cádmio.
Partícula reguladora 19S
A partícula 19S em eucariotos consiste em 19 proteínas individuais e é divisível em dois subconjuntos, uma base de 9 subunidades que se liga diretamente ao anel α da partícula central 20S e uma tampa de 10 subunidades. Seis das nove proteínas básicas são subunidades ATPase da família AAA, e um homólogo evolutivo dessas ATPases existe em archaea, chamado PAN (nucleotidase ativadora de proteassoma). A associação das partículas 19S e 20S requer a ligação do ATP às subunidades 19S ATPase, e a hidrólise do ATP é necessária para que o complexo montado degrade as proteínas enoveladas e ubiquitinadas. Observe que apenas a etapa de desdobramento do substrato requer energia da hidrólise do ATP, enquanto a ligação do ATP sozinha pode suportar todas as outras etapas necessárias para a degradação da proteína (por exemplo, montagem complexa, abertura do portão, translocação e proteólise). Na verdade, a ligação do ATP às ATPases por si só suporta a rápida degradação de proteínas não dobradas. No entanto, embora a hidrólise do ATP seja necessária apenas para o desdobramento, ainda não está claro se essa energia pode ser usada no acoplamento de algumas dessas etapas.
Em 2012, dois esforços independentes elucidaram a arquitetura molecular do proteassoma 26S por microscopia eletrônica de partícula única. Em 2016, três esforços independentes determinaram a primeira estrutura de resolução quase atômica do proteassoma humano 26S na ausência de substratos por crio-EM. Em 2018, um grande esforço elucidou os mecanismos detalhados de deubiquitylation, iniciação de translocação e desdobramento processivo de substratos determinando simultaneamente sete estruturas atômicas de proteassoma 26S acoplado a substrato. No coração do 19S, diretamente adjacente ao 20S, estão as AAA-ATPases (proteínas AAA) que se agrupam em um anel heterohexamérico da ordem Rpt1/Rpt2/Rpt6/Rpt3/Rpt4/Rpt5. Este anel é um trímero de dímeros: Rpt1/Rpt2, Rpt6/Rpt3 e Rpt4/Rpt5 dimerizam por meio de suas bobinas N-terminais. Essas espirais se projetam do anel hexamérico. As maiores partículas reguladoras não-ATPases Rpn1 e Rpn2 se ligam às pontas de Rpt1/2 e Rpt6/3, respectivamente. O receptor de ubiquitina Rpn13 liga-se ao Rpn2 e completa o subcomplexo de base. A tampa cobre metade do hexâmero AAA-ATPase (Rpt6/Rpt3/Rpt4) e, inesperadamente, contata diretamente o 20S via Rpn6 e, em menor extensão, Rpn5. As subunidades Rpn9, Rpn5, Rpn6, Rpn7, Rpn3 e Rpn12, que estão estruturalmente relacionadas entre si e com as subunidades do complexo COP9 e eIF3 (daí chamadas de subunidades PCI) formam uma estrutura semelhante a uma ferradura envolvendo o heterodímero Rpn8/Rpn11. Rpn11, a enzima deubiquitinadora, é colocada na boca do hexâmero AAA-ATPase, idealmente posicionada para remover porções de ubiquitina imediatamente antes da translocação de substratos para o 20S. O segundo receptor de ubiquitina identificado até o momento, Rpn10, está posicionado na periferia da pálpebra, próximo às subunidades Rpn8 e Rpn9.
Mudanças conformacionais de 19S
A partícula reguladora 19S dentro da holoenzima do proteassoma 26S foi observada em seis estados conformacionais fortemente diferentes na ausência de substratos até o momento. Uma característica da configuração AAA-ATPase neste estado predominante de baixa energia é um arranjo semelhante a uma escada ou arruela dos domínios AAA. Na presença de ATP, mas ausência de substrato três alternativas, conformações menos abundantes do 19S são adotadas diferindo principalmente no posicionamento da tampa em relação ao módulo AAA-ATPase. Na presença de ATP-γS ou de um substrato, foram observadas consideravelmente mais conformações exibindo mudanças estruturais dramáticas do módulo AAA-ATPase. Algumas das conformações ligadas ao substrato têm alta similaridade com as sem substrato, mas não são totalmente idênticas, particularmente no módulo AAA-ATPase. Antes da montagem 26S, a partícula reguladora 19S em uma forma livre também foi observada em sete estados conformacionais. Notavelmente, todos esses conformers são um pouco diferentes e apresentam características distintas. Assim, a partícula reguladora 19S pode amostrar pelo menos 20 estados conformacionais sob diferentes condições fisiológicas.
Regulação dos anos 20 pelos anos 19
A partícula reguladora 19S é responsável por estimular o 20S a degradar proteínas. Uma função primária das ATPases regulatórias 19S é abrir o portão no 20S que bloqueia a entrada de substratos na câmara de degradação. O mecanismo pelo qual a ATPase proteassômica abre esse portão foi recentemente elucidado. A abertura do portão 20S e, portanto, a degradação do substrato, requer os terminais C das ATPases proteassômicas, que contém um motivo específico (ou seja, motivo HbYX). As ATPases C-terminais ligam-se a bolsos no topo do 20S e prendem o complexo ATPase ao complexo proteolítico 20S, unindo assim o equipamento de desdobramento do substrato com a maquinaria de degradação do 20S. A ligação desses terminais C nesses bolsos 20S por si só estimula a abertura do portão no 20S da mesma maneira que uma "chave na fechadura" abre uma porta. O mecanismo preciso pelo qual esta "chave na fechadura" As funções do mecanismo foram estruturalmente elucidadas no contexto do proteassoma 26S humano em resolução quase atômica, sugerindo que a inserção de cinco terminais C das subunidades ATPase Rpt1/2/3/5/6 nos bolsos da superfície 20S é necessária para abrir completamente o portão 20S.
Outras partículas reguladoras
Os proteassomas 20S também podem associar-se a um segundo tipo de partícula reguladora, a partícula reguladora 11S, uma estrutura heptamérica que não contém nenhuma ATPase e pode promover a degradação de peptídeos curtos, mas não de proteínas completas. Presume-se que isso ocorre porque o complexo não pode desdobrar substratos maiores. Essa estrutura também é conhecida como PA28, REG ou PA26. Os mecanismos pelos quais ela se liga à partícula central através das caudas C-terminais de suas subunidades e induz alterações conformacionais do anel α para abrir o portão 20S sugerem um mecanismo semelhante para a partícula 19S. A expressão da partícula 11S é induzida pelo interferon gama e é responsável, em conjunto com as subunidades β do imunoproteassoma, pela geração de peptídeos que se ligam ao complexo principal de histocompatibilidade.
Outro tipo de partícula reguladora não ATPase é o Blm10 (levedura) ou PA200/PSME4 (humano). Ele abre apenas uma subunidade α no portão 20S e se dobra em uma cúpula com um poro muito pequeno sobre ele.
Montagem
A montagem do proteassoma é um processo complexo devido ao número de subunidades que devem associar-se para formar um complexo ativo. As subunidades β são sintetizadas com "propeptídeos" que são modificados pós-traducionalmente durante a montagem da partícula 20S para expor o sítio ativo proteolítico. A partícula 20S é montada a partir de dois meio-proteassomas, cada um dos quais consiste em um anel pró-β de sete membros ligado a um anel α de sete membros. A associação dos anéis β dos dois meio-proteassomas desencadeia a autólise dependente de treonina dos pró-peptídeos para expor o sítio ativo. Essas interações β são mediadas principalmente por pontes salinas e interações hidrofóbicas entre hélices alfa conservadas, cuja interrupção por mutação danifica a capacidade de montagem do proteassoma. A montagem dos semi-proteassomas, por sua vez, é iniciada pela montagem das subunidades α em seu anel heptamérico, formando um molde para a associação do anel pró-β correspondente. A montagem das subunidades α não foi caracterizada.
Apenas recentemente, o processo de montagem da partícula reguladora 19S foi elucidado de forma considerável. A partícula reguladora 19S é montada como dois subcomponentes distintos, a base e a tampa. A montagem do complexo de base é facilitada por quatro acompanhantes de montagem, Hsm3/S5b, Nas2/p27, Rpn14/PAAF1 e Nas6/gankyrin (nomes para leveduras/mamíferos). Essas chaperonas de montagem ligam-se às subunidades AAA-ATPase e sua principal função parece ser garantir a montagem adequada do anel heterohexamérico AAA-ATPase. Até o momento, ainda está em debate se o complexo de base se monta separadamente, se a montagem é modelada pela partícula central 20S ou se existem caminhos alternativos de montagem. Além das quatro chaperonas de montagem, a enzima deubiquitinadora Ubp6/Usp14 também promove a montagem de bases, mas não é essencial. A tampa é montada separadamente em uma ordem específica e não requer acompanhantes de montagem.
Processo de degradação de proteínas
Ubiquitinação e segmentação
As proteínas são alvo de degradação pelo proteassoma com modificação covalente de um resíduo de lisina que requer as reações coordenadas de três enzimas. Na primeira etapa, uma enzima ativadora de ubiquitina (conhecida como E1) hidrolisa ATP e adenila uma molécula de ubiquitina. Isso é então transferido para o resíduo de cisteína do sítio ativo de E1 em conjunto com a adenilação de uma segunda ubiquitina. Essa ubiquitina adenilada é então transferida para uma cisteína de uma segunda enzima, a enzima conjugadora de ubiquitina (E2). Na última etapa, um membro de uma classe altamente diversa de enzimas conhecidas como ubiquitina ligases (E3) reconhece a proteína específica a ser ubiquitinada e catalisa a transferência da ubiquitina de E2 para esta proteína alvo. Uma proteína-alvo deve ser marcada com pelo menos quatro monômeros de ubiquitina (na forma de uma cadeia de poliubiquitina) antes de ser reconhecida pela tampa do proteassoma. É, portanto, o E3 que confere especificidade de substrato a este sistema. O número de proteínas E1, E2 e E3 expressas depende do organismo e do tipo de célula, mas existem muitas enzimas E3 diferentes presentes em humanos, indicando que existe um grande número de alvos para o sistema ubiquitina proteassoma.
O mecanismo pelo qual uma proteína poliubiquitinada é direcionada para o proteassoma não é totalmente compreendido. Alguns instantâneos de alta resolução do proteassoma ligado a uma proteína poliubiquitinada sugerem que os receptores de ubiquitina podem ser coordenados com deubiquitinase Rpn11 para direcionamento e envolvimento inicial do substrato. As proteínas receptoras de ubiquitina têm um domínio N-terminal semelhante à ubiquitina (UBL) e um ou mais domínios associados à ubiquitina (UBA). Os domínios UBL são reconhecidos pelas tampas do proteassoma 19S e os domínios UBA se ligam à ubiquitina por meio de feixes de três hélices. Essas proteínas receptoras podem escoltar proteínas poliubiquitinadas para o proteassoma, embora as especificidades dessa interação e sua regulação não sejam claras.
A própria proteína ubiquitina tem 76 aminoácidos de comprimento e foi nomeada devido à sua natureza ubíqua, pois possui uma sequência altamente conservada e é encontrada em todos os organismos eucarióticos conhecidos. Os genes que codificam a ubiquitina em eucariotos são organizados em repetições em tandem, possivelmente devido às demandas pesadas de transcrição desses genes para produzir ubiquitina suficiente para a célula. Foi proposto que a ubiquitina é a proteína de evolução mais lenta identificada até o momento. A ubiquitina contém sete resíduos de lisina aos quais outra ubiquitina pode ser ligada, resultando em diferentes tipos de cadeias de poliubiquitina. As cadeias nas quais cada ubiquitina adicional está ligada à lisina 48 da ubiquitina anterior têm um papel no direcionamento do proteassoma, enquanto outros tipos de cadeias podem estar envolvidos em outros processos.
Desubiquitilação
As cadeias de ubiquitina conjugadas a uma proteína direcionada para a degradação proteassômica são normalmente removidas por qualquer uma das três enzimas deubiquitilantes associadas ao proteassoma (DUBs), que são Rpn11, Ubp6/USP14 e UCH37. Este processo recicla a ubiquitina e é essencial para manter o reservatório de ubiquitina nas células. Rpn11 é uma subunidade estequiométrica intrínseca da partícula reguladora 19S e é essencial para a função do proteassoma 26S. A atividade DUB de Rpn11 é aumentada no proteassoma em comparação com sua forma monomérica. Como Rpn11 remove uma cadeia de ubiquitina em bloco de um substrato de proteína foi capturado por uma estrutura atômica do proteassoma humano acoplado ao substrato em uma conformação denominada EB. Curiosamente, esta estrutura também mostra como a atividade DUB está acoplada ao reconhecimento do substrato pela proteassômica AAA-ATPase. Ao contrário de Rpn11, USP14 e UCH37 são os DUBs que nem sempre estão associados ao proteassoma. Nas células, descobriu-se que cerca de 10-40% dos proteossomas têm USP14 associado. Ambos Ubp6/USP14 e UCH37 são amplamente ativados pelo proteassoma e exibem uma atividade DUB muito baixa sozinha. Uma vez ativado, descobriu-se que o USP14 suprime a função do proteassoma por sua atividade DUB e induzindo vias paralelas de transições conformacionais do proteassoma, uma das quais proibiu diretamente a inserção do substrato na AAA-ATPase, conforme observado intuitivamente por microscopia eletrônica criogênica resolvida no tempo. Parece que o USP14 regula a função do proteassoma em vários pontos de controle, competindo cataliticamente com Rpn11 e reprogramando alostericamente os estados AAA-ATPase, o que é bastante inesperado para um DUB. Estas observações implicam que a regulação do proteassoma pode depender de suas transições dinâmicas de estados conformacionais.
Desdobramento e translocação
Depois que uma proteína foi ubiquitinada, ela é reconhecida pela partícula reguladora 19S em uma etapa de ligação dependente de ATP. A proteína substrato deve então entrar no interior da subunidade 20S para entrar em contato com os sítios proteolíticos ativos. Como o canal central da partícula 20S é estreito e fechado pelas caudas N-terminais das subunidades do anel α, os substratos devem ser pelo menos parcialmente desdobrados antes de entrarem no núcleo. A passagem do substrato desdobrado para o núcleo é chamada de translocação e ocorre necessariamente após a deubiquitinação. No entanto, a ordem em que os substratos são deubiquitinados e desdobrados ainda não está clara. Qual desses processos é a etapa limitante da velocidade na reação global de proteólise depende do substrato específico; para algumas proteínas, o processo de desdobramento é limitante, enquanto a deubiquitinação é a etapa mais lenta para outras proteínas. Sugere-se que a extensão em que os substratos devem ser desdobrados antes da translocação seja de cerca de 20 resíduos de aminoácidos pela estrutura atômica do proteassoma 26S acoplado ao substrato no estado compatível com a deubiquitilação, mas estrutura terciária substancial e, em particular, interações não locais, como dissulfeto ligações, são suficientes para inibir a degradação. A presença de segmentos proteicos intrinsecamente desordenados de tamanho suficiente, seja no terminal da proteína ou internamente, também foi proposta para facilitar a iniciação eficiente da degradação.
O portão formado pelas subunidades α impede que peptídeos com mais de quatro resíduos entrem no interior da partícula 20S. As moléculas de ATP ligadas antes da etapa inicial de reconhecimento são hidrolisadas antes da translocação. Embora a energia seja necessária para o desdobramento do substrato, ela não é necessária para a translocação. O proteassoma 26S montado pode degradar proteínas desdobradas na presença de um análogo de ATP não hidrolisável, mas não pode degradar proteínas dobradas, indicando que a energia da hidrólise de ATP é usada para o desdobramento do substrato. A passagem do substrato desdobrado através do portão aberto ocorre via difusão facilitada se o cap 19S estiver no estado ligado ao ATP.
O mecanismo de desdobramento das proteínas globulares é necessariamente geral, mas um tanto dependente da sequência de aminoácidos. Sequências longas de glicina e alanina alternadas mostraram inibir o desdobramento do substrato, diminuindo a eficiência da degradação proteassômica; isso resulta na liberação de subprodutos parcialmente degradados, possivelmente devido ao desacoplamento da hidrólise do ATP e etapas de desdobramento. Essas repetições de glicina-alanina também são encontradas na natureza, por exemplo, na fibroína da seda; em particular, certos produtos gênicos do vírus Epstein-Barr com essa sequência podem interromper o proteassoma, ajudando o vírus a se propagar ao impedir a apresentação do antígeno no complexo principal de histocompatibilidade.

Proteólise
O proteassoma funciona como uma endoprotease. O mecanismo de proteólise pelas subunidades β da partícula central 20S é através de um ataque nucleofílico dependente de treonina. Este mecanismo pode depender de uma molécula de água associada para a desprotonação da hidroxila reativa da treonina. A degradação ocorre dentro da câmara central formada pela associação dos dois anéis β e normalmente não libera produtos parcialmente degradados, em vez disso, reduz o substrato a polipeptídeos curtos tipicamente de 7 a 9 resíduos de comprimento, embora possam variar de 4 a 25 resíduos, dependendo organismo e substrato. O mecanismo bioquímico que determina o comprimento do produto não está totalmente caracterizado. Embora as três subunidades β catalíticas tenham um mecanismo comum, elas têm especificidades de substrato ligeiramente diferentes, que são consideradas semelhantes a quimotripsina, semelhantes a tripsina e hidrolisadoras de peptídeos peptidil-glutamil (PHGH). Essas variações na especificidade são o resultado de contatos interatômicos com resíduos locais próximos aos sítios ativos de cada subunidade. Cada subunidade β catalítica também possui um resíduo de lisina conservado necessário para a proteólise.
Embora o proteassoma normalmente produza fragmentos peptídicos muito curtos, em alguns casos esses produtos são eles próprios moléculas biologicamente ativas e funcionais. Certos fatores de transcrição que regulam a expressão de genes específicos, incluindo um componente do complexo mamífero NF-κB, são sintetizados como precursores inativos cuja ubiquitinação e subsequente degradação proteassômica os converte em uma forma ativa. Essa atividade requer que o proteassoma clive internamente a proteína substrato, em vez de degradá-la processualmente de um terminal. Sugeriu-se que loops longos nessas proteínas' As superfícies servem como substratos proteassômicos e entram na cavidade central, enquanto a maior parte da proteína permanece do lado de fora. Efeitos semelhantes foram observados em proteínas de levedura; esse mecanismo de degradação seletiva é conhecido como processamento dependente de ubiquitina/proteassoma regulado (RUP).
Degradação independente de ubiquitina
Embora a maioria dos substratos proteassômicos devam ser ubiquitinados antes de serem degradados, há algumas exceções a essa regra geral, especialmente quando o proteassoma desempenha um papel normal no processamento pós-traducional da proteína. A ativação proteassômica de NF-κB pelo processamento de p105 em p50 via proteólise interna é um exemplo importante. Algumas proteínas consideradas instáveis devido a regiões intrinsecamente não estruturadas são degradadas de maneira independente da ubiquitina. O exemplo mais conhecido de um substrato de proteassoma independente de ubiquitina é a enzima ornitina descarboxilase. Mecanismos independentes de ubiquitina direcionados aos principais reguladores do ciclo celular, como p53, também foram relatados, embora p53 também esteja sujeito à degradação dependente de ubiquitina. Finalmente, proteínas estruturalmente anormais, mal dobradas ou altamente oxidadas também estão sujeitas à degradação independente de ubiquitina e independente de 19S sob condições de estresse celular.
Evolução
O proteassoma 20S é onipresente e essencial em eucariotos e archaea. A ordem bacteriana Actinomycetales também compartilha homólogos do proteassoma 20S, enquanto a maioria das bactérias possui genes de choque térmico hslV e hslU, cujos produtos gênicos são uma protease multimérica disposta em um anel de duas camadas e uma ATPase. A proteína hslV foi levantada a hipótese de se assemelhar ao provável ancestral do proteassoma 20S. Em geral, o HslV não é essencial nas bactérias e nem todas as bactérias o possuem, enquanto alguns protistas possuem os sistemas 20S e hslV. Muitas bactérias também possuem outros homólogos do proteassoma e uma ATPase associada, principalmente ClpP e ClpX. Essa redundância explica por que o sistema HslUV não é essencial.
A análise da sequência sugere que as subunidades β catalíticas divergiram mais cedo na evolução do que as subunidades α predominantemente estruturais. Em bactérias que expressam um proteassoma 20S, as subunidades β têm alta identidade de sequência com subunidades β archaeais e eucarióticas, enquanto a identidade de sequência α é muito menor. A presença de proteassomas 20S em bactérias pode resultar de transferência lateral de genes, enquanto a diversificação de subunidades entre eucariotos é atribuída a múltiplos eventos de duplicação de genes.
Controle do ciclo celular
A progressão do ciclo celular é controlada pela ação ordenada de quinases dependentes de ciclina (CDKs), ativadas por ciclinas específicas que demarcam as fases do ciclo celular. As ciclinas mitóticas, que persistem na célula por apenas alguns minutos, têm um dos períodos de vida mais curtos de todas as proteínas intracelulares. Após um complexo CDK-ciclina ter desempenhado sua função, a ciclina associada é poliubiquitinada e destruída pelo proteassoma, que fornece direcionalidade para o ciclo celular. Em particular, a saída da mitose requer a dissociação dependente do proteassoma do componente regulador ciclina B do complexo do fator promotor da mitose. Em células de vertebrados, "deslizamento" através do checkpoint mitótico levando à saída prematura da fase M pode ocorrer apesar do atraso desta saída pelo checkpoint do fuso.
Os pontos de verificação anteriores do ciclo celular, como a verificação do ponto pós-restrição entre a fase G1 e a fase S, envolvem de forma semelhante a degradação proteassômica da ciclina A, cuja ubiquitinação é promovida pelo complexo promotor da anáfase (APC), uma ubiquitina ligase E3. O APC e o complexo proteico Skp1/Cul1/F-box (complexo SCF) são os dois principais reguladores da degradação da ciclina e do controle do ponto de controle; o próprio SCF é regulado pelo APC por meio da ubiquitinação da proteína adaptadora, Skp2, que impede a atividade do SCF antes da transição G1-S.Componentes individuais da partícula 19S têm suas próprias funções reguladoras. A ganquirina, uma oncoproteína recentemente identificada, é um dos subcomponentes 19S que também se liga fortemente à quinase dependente de ciclina CDK4 e desempenha um papel fundamental no reconhecimento da p53 ubiquitinada, por meio de sua afinidade pela ubiquitina ligase MDM2. A ganquirina é antiapoptótica e demonstrou ser superexpressa em alguns tipos de células tumorais, como o carcinoma hepatocelular.
Tal como os eucariotas, alguns archaea também usam o proteassoma para controlar o ciclo celular, especificamente controlando a divisão celular mediada por ESCRT-III.
Regulação do crescimento vegetal
Nas plantas, a sinalização por auxinas, ou fitohormônios que ordenam a direção e o tropismo do crescimento vegetal, induz o direcionamento de uma classe de repressores de fatores de transcrição conhecidos como proteínas Aux/IAA para degradação proteassômica. Essas proteínas são ubiquitinadas por SCFTIR1, ou SCF em complexo com o receptor de auxina TIR1. A degradação das proteínas Aux/IAA desreprime os fatores de transcrição na família do fator de resposta a auxina (ARF) e induz a expressão gênica dirigida por ARF. As consequências celulares da ativação do ARF dependem do tipo de planta e do estágio de desenvolvimento, mas estão envolvidas no direcionamento do crescimento nas raízes e nas nervuras das folhas. Acredita-se que a resposta específica à desrepressão de ARF seja mediada pela especificidade no pareamento de proteínas ARF e Aux/IAA individuais.
Apoptose
Ambos os sinais internos e externos podem levar à indução de apoptose, ou morte celular programada. A desconstrução resultante dos componentes celulares é realizada principalmente por proteases especializadas conhecidas como caspases, mas o proteassoma também desempenha papéis importantes e diversos no processo apoptótico. O envolvimento do proteassoma neste processo é indicado pelo aumento da ubiquitinação de proteínas e das enzimas E1, E2 e E3 que é observado bem antes da apoptose. Durante a apoptose, proteassomas localizados no núcleo também foram observados translocados para bolhas da membrana externa características da apoptose.
A inibição do proteassoma tem efeitos diferentes na indução da apoptose em diferentes tipos de células. Em geral, o proteassoma não é necessário para a apoptose, embora a inibição seja pró-apoptótica na maioria dos tipos celulares estudados. A apoptose é mediada pela interrupção da degradação regulada de proteínas pró-crescimento do ciclo celular. No entanto, algumas linhagens celulares - em particular, culturas primárias de células quiescentes e diferenciadas, como timócitos e neurônios - são impedidas de sofrer apoptose na exposição a inibidores de proteassoma. O mecanismo para este efeito não é claro, mas supõe-se que seja específico para células em estados quiescentes, ou que resulte da atividade diferencial da quinase pró-apoptótica JNK. A capacidade dos inibidores de proteassoma de induzir apoptose em células que se dividem rapidamente foi explorada em vários agentes quimioterápicos desenvolvidos recentemente, como bortezomibe e salinosporamida A.
Resposta ao estresse celular
Em resposta ao estresse celular - como infecção, choque térmico ou dano oxidativo - são expressas proteínas de choque térmico que identificam proteínas mal dobradas ou desdobradas e as direcionam para a degradação proteassômica. As proteínas chaperonas Hsp27 e Hsp90 têm sido implicadas no aumento da atividade do sistema ubiquitina-proteassoma, embora não sejam participantes diretos do processo. Hsp70, por outro lado, se liga a manchas hidrofóbicas expostas na superfície de proteínas mal dobradas e recruta ligases de ubiquitina E3, como CHIP, para marcar as proteínas para degradação proteassômica. A proteína CHIP (terminal carboxílico da proteína que interage com Hsp70) é regulada por meio da inibição de interações entre a enzima E3 CHIP e seu parceiro de ligação E2.
Existem mecanismos semelhantes para promover a degradação de proteínas danificadas oxidativamente através do sistema de proteassoma. Em particular, proteassomas localizados no núcleo são regulados por PARP e degradam ativamente histonas inapropriadamente oxidadas. Proteínas oxidadas, que muitas vezes formam grandes agregados amorfos na célula, podem ser degradadas diretamente pela partícula central 20S sem a tampa regulatória 19S e não requerem hidrólise de ATP ou marcação com ubiquitina. No entanto, altos níveis de dano oxidativo aumentam o grau de reticulação entre os fragmentos de proteínas, tornando os agregados resistentes à proteólise. Números e tamanhos maiores desses agregados altamente oxidados estão associados ao envelhecimento.
A desregulação do sistema de proteassoma da ubiquitina pode contribuir para várias doenças neurais. Pode levar a tumores cerebrais, como astrocitomas. Em algumas das doenças neurodegenerativas de início tardio que compartilham a agregação de proteínas mal dobradas como uma característica comum, como a doença de Parkinson e a doença de Alzheimer, grandes agregados insolúveis de proteínas mal dobradas podem se formar e resultar em neurotoxicidade, através de mecanismos que ainda não são bem compreendidos. A diminuição da atividade do proteassoma foi sugerida como causa da agregação e formação de corpos de Lewy no Parkinson. Esta hipótese é apoiada pela observação de que modelos de levedura de Parkinson são mais suscetíveis à toxicidade da α-sinucleína, o principal componente proteico dos corpos de Lewy, sob condições de baixa atividade do proteassoma. A atividade proteassômica prejudicada pode estar subjacente a distúrbios cognitivos, como os distúrbios do espectro do autismo e doenças musculares e nervosas, como a miopatia por corpos de inclusão.
Papel no sistema imunológico
O proteassoma desempenha um papel direto, mas crítico, na função do sistema imunológico adaptativo. Os antígenos peptídicos são exibidos pelas proteínas do complexo principal de histocompatibilidade classe I (MHC) na superfície das células apresentadoras de antígenos. Esses peptídeos são produtos da degradação proteassômica de proteínas originadas pelo patógeno invasor. Embora proteassomas constitutivamente expressos possam participar desse processo, um complexo especializado composto de proteínas, cuja expressão é induzida pelo interferon gama, são os principais produtores de peptídeos com tamanho e composição ótimos para ligação ao MHC. Essas proteínas cuja expressão aumenta durante a resposta imune incluem a partícula reguladora 11S, cujo principal papel biológico conhecido é a regulação da produção de ligantes do MHC, e subunidades β especializadas denominadas β1i, β2i e β5i com especificidade de substrato alterada. O complexo formado com as subunidades β especializadas é conhecido como imunoproteassoma. Outra subunidade variante β5i, β5t, é expressa no timo, levando a um "timoproteassoma" cuja função ainda não está clara.
A força da ligação do ligante MHC de classe I depende da composição do ligante C-terminal, pois os peptídeos se ligam por ligações de hidrogênio e por contatos próximos com uma região chamada "B pocket" na superfície do MHC. Muitos alelos de classe I do MHC preferem resíduos C-terminais hidrofóbicos, e o complexo imunoproteassoma é mais provável de gerar C-terminais hidrofóbicos.
Devido ao seu papel na geração da forma ativada de NF-κB, um regulador antiapoptótico e pró-inflamatório da expressão de citocinas, a atividade proteassômica tem sido associada a doenças inflamatórias e autoimunes. Níveis aumentados de atividade de proteassoma correlacionam-se com a atividade da doença e têm sido implicados em doenças autoimunes, incluindo lúpus eritematoso sistêmico e artrite reumatóide.
O proteassoma também está envolvido na proteólise mediada por anticorpos intracelulares de vírions ligados a anticorpos. Nesta via de neutralização, TRIM21 (uma proteína da família do motivo tripartido) liga-se à imunoglobulina G para direcionar o virião para o proteassoma onde é degradado.
Inibidores de proteassoma

Os inibidores de proteassoma têm atividade antitumoral eficaz na cultura celular, induzindo a apoptose ao interromper a degradação regulada das proteínas do ciclo celular pró-crescimento. Esta abordagem de indução seletiva de apoptose em células tumorais provou ser eficaz em modelos animais e testes em humanos.
ALactacistina, um produto natural sintetizado pela bactéria Streptomyces, foi o primeiro inibidor de proteassoma não peptídico descoberto e é amplamente utilizado como ferramenta de pesquisa em bioquímica e biologia celular. A lactacistina foi licenciada para a Myogenics/Proscript, que foi adquirida pela Millennium Pharmaceuticals, agora parte da Takeda Pharmaceuticals. A lactacistina modifica covalentemente a treonina amino-terminal das subunidades β catalíticas do proteassoma, particularmente a subunidade β5 responsável pela atividade semelhante à quimotripsina do proteassoma. Esta descoberta ajudou a estabelecer o proteassoma como uma classe mecanisticamente nova de protease: uma protease de treonina amino-terminal.
O Bortezomibe (Boronated MG132), uma molécula desenvolvida pela Millennium Pharmaceuticals e comercializada como Velcade, é o primeiro inibidor de proteassoma a alcançar uso clínico como agente quimioterápico. O bortezomib é utilizado no tratamento do mieloma múltiplo. Notavelmente, observou-se que o mieloma múltiplo resulta em níveis aumentados de peptídeos derivados de proteassoma no soro sanguíneo que diminuem para níveis normais em resposta à quimioterapia bem-sucedida. Estudos em animais indicaram que o bortezomibe também pode ter efeitos clinicamente significativos no câncer pancreático. Estudos pré-clínicos e clínicos iniciais foram iniciados para examinar a eficácia do bortezomibe no tratamento de outros cânceres relacionados a células B, particularmente alguns tipos de linfoma não-Hodgkin. Os resultados clínicos também parecem justificar o uso de inibidor de proteassoma combinado com quimioterapia, para leucemia linfoblástica aguda de células B Os inibidores de proteassoma podem matar alguns tipos de células de leucemia cultivadas que são resistentes a glicocorticóides.
A molécula ritonavir, comercializada como Norvir, foi desenvolvida como um inibidor de protease e usada para combater a infecção pelo HIV. No entanto, demonstrou inibir proteassomas, bem como proteases livres; para ser específico, a atividade semelhante à quimotripsina do proteassoma é inibida pelo ritonavir, enquanto a atividade semelhante à tripsina é um pouco aumentada. Estudos em modelos animais sugerem que o ritonavir pode ter efeitos inibitórios no crescimento de células de glioma.
Os inibidores de proteassoma também se mostraram promissores no tratamento de doenças autoimunes em modelos animais. Por exemplo, estudos em camundongos com enxertos de pele humana encontraram uma redução no tamanho das lesões de psoríase após o tratamento com um inibidor de proteassoma. Os inibidores também mostram efeitos positivos em modelos de asma em roedores.
A marcação e inibição do proteassoma também é de interesse em configurações de laboratório para estudos in vitro e in vivo da atividade proteassômica em células. Os inibidores laboratoriais mais comumente usados são a lactcistina e o peptídeo aldeído MG132 inicialmente desenvolvido pelo laboratório Goldberg. Os inibidores fluorescentes também foram desenvolvidos para marcar especificamente os locais ativos do proteassoma montado.
Significado clínico
O proteassoma e suas subunidades são de importância clínica por pelo menos dois motivos: (1) uma montagem complexa comprometida ou um proteassoma disfuncional pode estar associado à fisiopatologia subjacente de doenças específicas e (2) eles podem ser explorados como drogas alvos de intervenções terapêuticas. Mais recentemente, mais esforços foram feitos para considerar o proteassoma para o desenvolvimento de novos marcadores e estratégias de diagnóstico. Uma compreensão melhorada e abrangente da fisiopatologia do proteassoma deve levar a aplicações clínicas no futuro.
Os proteassomas formam um componente fundamental para o sistema ubiquitina-proteassoma (UPS) e correspondente Controle de Qualidade de Proteína celular (PQC). A ubiquitinação de proteínas e subsequente proteólise e degradação pelo proteassoma são mecanismos importantes na regulação do ciclo celular, crescimento e diferenciação celular, transcrição gênica, transdução de sinal e apoptose. Os defeitos do proteassoma levam à redução da atividade proteolítica e ao acúmulo de proteínas danificadas ou mal dobradas, o que pode contribuir para doenças neurodegenerativas, doenças cardiovasculares, respostas inflamatórias e doenças autoimunes e respostas sistêmicas de dano ao DNA levando a malignidades.
A pesquisa implicou os defeitos do UPS na patogênese de distúrbios neurodegenerativos e miodegenerativos, incluindo doença de Alzheimer, doença de Parkinson e doença de Pick, esclerose lateral amiotrófica (ALS), doença de Huntington e doença de Huntington. s, doença de Creutzfeldt-Jakob e doenças do neurônio motor, doenças de poliglutamina (PolyQ), distrofias musculares e várias formas raras de doenças neurodegenerativas associadas à demência. Como parte do sistema ubiquitina-proteassoma (UPS), o proteassoma mantém a homeostase da proteína cardíaca e, portanto, desempenha um papel significativo na lesão isquêmica cardíaca, hipertrofia ventricular e insuficiência cardíaca. Além disso, estão se acumulando evidências de que o UPS desempenha um papel essencial na transformação maligna. A proteólise UPS desempenha um papel importante nas respostas das células cancerígenas aos sinais estimuladores que são críticos para o desenvolvimento do câncer. Consequentemente, a expressão gênica por degradação de fatores de transcrição, como p53, c-jun, c-Fos, NF-κB, c-Myc, HIF-1α, MATα2, STAT3, proteínas de ligação a elementos regulados por esteróis e receptores de andrógenos são todos controlado pela UPS e, portanto, envolvido no desenvolvimento de várias malignidades. Além disso, o UPS regula a degradação de produtos gênicos supressores de tumor, como polipose adenomatosa coli (APC) no câncer colorretal, retinoblastoma (Rb). e supressor de tumor von Hippel-Lindau (VHL), bem como vários proto-oncogenes (Raf, Myc, Myb, Rel, Src, Mos, ABL). O UPS também está envolvido na regulação de respostas inflamatórias. Esta atividade é geralmente atribuída ao papel dos proteassomas na ativação de NF-κB que regula ainda mais a expressão de citocinas pró-inflamatórias como TNF-α, IL-β, IL-8, moléculas de adesão (ICAM-1, VCAM-1, P-selectina) e prostaglandinas e óxido nítrico (NO). Além disso, o UPS também desempenha um papel nas respostas inflamatórias como reguladores da proliferação de leucócitos, principalmente por meio da proteólise de ciclinas e da degradação de inibidores de CDK. Por fim, pacientes com doenças autoimunes com LES, síndrome de Sjögren e artrite reumatoide (AR) exibem predominantemente proteassomas circulantes que podem ser aplicados como biomarcadores clínicos.
Contenido relacionado
Ângela Vicente
Antianginal
Agarose