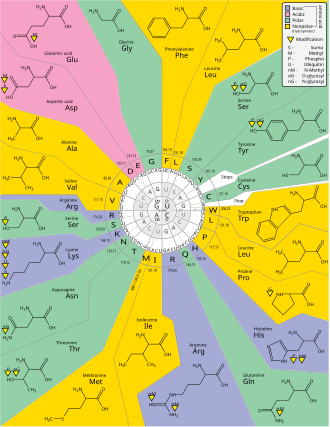
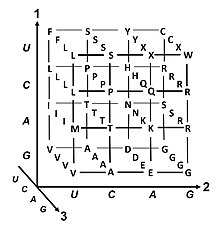
Código genético

O código genético é o conjunto de regras usadas pelas células vivas para traduzir as informações codificadas no material genético (sequências de DNA ou RNA de trigêmeos de nucleotídeos, ou códons) em proteínas. A tradução é realizada pelo ribossomo, que liga os aminoácidos proteinogênicos em uma ordem especificada pelo RNA mensageiro (mRNA), usando moléculas de RNA de transferência (tRNA) para transportar aminoácidos e para ler o mRNA três nucleotídeos por vez. O código genético é muito semelhante entre todos os organismos e pode ser expresso em uma tabela simples com 64 entradas.
Os códons especificam qual aminoácido será adicionado a seguir durante a biossíntese de proteínas. Com algumas exceções, um códon de três nucleotídeos em uma sequência de ácido nucleico especifica um único aminoácido. A grande maioria dos genes é codificada com um único esquema (consulte a tabela de códons de RNA). Esse esquema costuma ser chamado de código genético canônico ou padrão, ou simplesmente o código genético, embora existam códigos variantes (como nas mitocôndrias).
História
Esforços para entender como as proteínas são codificadas começaram depois que a estrutura do DNA foi descoberta em 1953. Os principais descobridores, o biofísico inglês Francis Crick e o biólogo americano James Watson, trabalhando juntos no Laboratório Cavendish da Universidade de Cambridge, levantaram a hipótese que a informação flui do DNA e que existe uma ligação entre o DNA e as proteínas. O físico soviético-americano George Gamow foi o primeiro a apresentar um esquema viável para a síntese de proteínas a partir do DNA. Ele postulou que conjuntos de três bases (trigêmeos) devem ser empregados para codificar os 20 aminoácidos padrão usados pelas células vivas para construir proteínas, o que permitiria um máximo de 43 = 64 aminoácidos. Ele nomeou essa interação DNA-proteína (o código genético original) como o "código diamante."
Em 1954, Gamow criou uma organização científica informal, o RNA Tie Club, conforme sugerido por Watson, para cientistas de diferentes convicções interessados em como as proteínas eram sintetizadas a partir dos genes. No entanto, o clube poderia ter apenas 20 membros permanentes para representar cada um dos 20 aminoácidos; e quatro membros honorários adicionais para representar os quatro nucleotídeos do DNA.
A primeira contribuição científica do clube, posteriormente registrada como "um dos artigos inéditos mais importantes da história da ciência" e "o artigo inédito mais famoso nos anais da biologia molecular" foi feito por Crick. Crick apresentou um artigo datilografado intitulado "Sobre modelos degenerados e a hipótese do adaptador: uma nota para o clube de gravatas de RNA" aos membros do clube em janeiro de 1955, que "mudaram totalmente a maneira como pensávamos sobre a síntese de proteínas", como lembrou Watson. A hipótese afirma que o código trigêmeo não foi passado para os aminoácidos como Gamow pensava, mas carregado por uma molécula diferente, um adaptador, que interage com os aminoácidos. O adaptador foi posteriormente identificado como tRNA.
Códons
O experimento de Crick, Brenner, Barnett e Watts-Tobin demonstrou pela primeira vez que os códons consistem em três bases de DNA. Marshall Nirenberg e Heinrich J. Matthaei foram os primeiros a revelar a natureza de um códon em 1961.
Eles usaram um sistema livre de células para traduzir uma sequência de RNA de poliuracila (ou seja, UUUUU...) e descobriram que o polipeptídeo que eles sintetizaram consistia apenas no aminoácido fenilalanina. Assim, eles deduziram que o códon UUU especificava o aminoácido fenilalanina.
Isso foi seguido por experimentos no laboratório de Severo Ochoa que demonstraram que a sequência de RNA de poliadenina (AAAAA...) codificava o polipeptídeo polilisina e que a sequência de RNA de policitosina (CCCCC...) codificado para o polipeptídeo poliprolina. Portanto, o códon AAA especifica o aminoácido lisina, e o códon CCC especifica o aminoácido prolina. Usando vários copolímeros, a maioria dos códons restantes foram então determinados.
Trabalhos subsequentes de Har Gobind Khorana identificaram o restante do código genético. Pouco tempo depois, Robert W. Holley determinou a estrutura do RNA de transferência (tRNA), a molécula adaptadora que facilita o processo de tradução do RNA em proteína. Este trabalho foi baseado nos estudos anteriores de Ochoa, rendendo a este último o Prêmio Nobel de Fisiologia ou Medicina em 1959 pelo trabalho na enzimologia da síntese de RNA.
Estendendo este trabalho, Nirenberg e Philip Leder revelaram a natureza triplete do código e decifraram seus códons. Nesses experimentos, várias combinações de mRNA foram passadas por um filtro que continha ribossomos, os componentes das células que traduzem o RNA em proteína. Trigêmeos únicos promoveram a ligação de tRNAs específicos ao ribossomo. Leder e Nirenberg foram capazes de determinar as sequências de 54 dos 64 códons em seus experimentos. Khorana, Holley e Nirenberg receberam o Nobel de 1968 por seu trabalho.
Os três stop códons foram nomeados pelos descobridores Richard Epstein e Charles Steinberg. "Âmbar" recebeu o nome de seu amigo Harris Bernstein, cujo sobrenome significa "âmbar" em alemão. Os outros dois códons de parada foram denominados "ochre" e "opala" para manter os "nomes das cores" tema.
Códigos genéticos expandidos (biologia sintética)
Em um amplo público acadêmico, o conceito da evolução do código genético do código genético original e ambíguo para um código bem definido ("congelado") com o repertório de 20 (+2) aminoácidos canônicos é amplamente aceito. No entanto, existem diferentes opiniões, conceitos, abordagens e ideias, qual é a melhor maneira de mudá-lo experimentalmente. Mesmo modelos são propostos que prevêem "pontos de entrada" para invasão de aminoácidos sintéticos do código genético.
Desde 2001, 40 aminoácidos não naturais foram adicionados às proteínas, criando um códon único (recodificação) e um correspondente par de transferência-RNA:aminoacil - tRNA-sintetase para codificá-lo com diversas propriedades físico-químicas e biológicas, a fim de ser usado como uma ferramenta para explorar a estrutura e a função da proteína ou para criar proteínas novas ou aprimoradas.
H. Murakami e M. Sisido estenderam alguns códons para ter quatro e cinco bases. Steven A. Benner construiu um 65º códon funcional (in vivo).
Em 2015, N. Budisa, D. Söll e colaboradores relataram a substituição completa de todos os 20.899 resíduos de triptofano (códons UGG) por tienopirrol-alanina não natural no código genético da bactéria Escherichia coli.
Em 2016 foi criado o primeiro organismo semissintético estável. Era uma bactéria (unicelular) com duas bases sintéticas (chamadas X e Y). As bases sobreviveram à divisão celular.
Em 2017, pesquisadores na Coreia do Sul relataram que haviam desenvolvido um camundongo com um código genético estendido que pode produzir proteínas com aminoácidos não naturais.
Em maio de 2019, os pesquisadores relataram a criação de um novo "Syn61" cepa da bactéria Escherichia coli. Esta cepa tem um genoma totalmente sintético que é refatorado (todas as sobreposições expandidas), recodificado (removendo completamente o uso de três dos 64 códons) e ainda modificado para remover os agora desnecessários tRNAs e fatores de liberação. É totalmente viável e cresce 1,6 × mais devagar que sua contraparte do tipo selvagem "MDS42".
Recursos
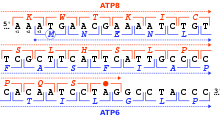
Frame de leitura
Um quadro de leitura é definido pelo trio inicial de nucleotídeos a partir do qual a tradução começa. Ele define o quadro para uma sequência de códons sucessivos e não sobrepostos, o que é conhecido como "quadro de leitura aberta" (ORF). Por exemplo, a string 5'-AAATGAACG-3' (veja a figura), se lido da primeira posição, contém os códons AAA, TGA e ACG; se lido a partir da segunda posição, contém os códons AAT e GAA; e se lido a partir da terceira posição, contém os códons ATG e AAC. Cada sequência pode, assim, ser lida em seu 5' → 3' direção em três quadros de leitura, cada um produzindo uma sequência de aminoácidos possivelmente distinta: no exemplo dado, Lys (K)-Trp (W)-Thr (T), Asn (N)-Glu (E) ou Met (M) -Asn (N), respectivamente (ao traduzir com o código mitocondrial dos vertebrados). Quando o DNA é de fita dupla, seis quadros de leitura possíveis são definidos, três na orientação direta em uma fita e três na direção oposta na fita oposta. Os quadros de codificação de proteínas são definidos por um códon de início, geralmente o primeiro códon AUG (ATG) na sequência de RNA (DNA).
Em eucariotos, ORFs em éxons são frequentemente interrompidos por íntrons.
Códons de início e fim
A tradução começa com um códon de iniciação de cadeia ou códon de início. O códon de início sozinho não é suficiente para iniciar o processo. Sequências próximas, como a sequência Shine-Dalgarno em E. coli e fatores de iniciação também são necessários para iniciar a tradução. O códon de iniciação mais comum é AUG, que é lido como metionina ou formilmetionina (em bactérias, mitocôndrias e plastídios). Códons de início alternativos dependendo do organismo incluem "GUG" ou "UUG"; esses códons normalmente representam valina e leucina, respectivamente, mas como códons iniciais eles são traduzidos como metionina ou formilmetionina.
Os três códons de parada têm nomes: UAG é amber, UGA é opal (às vezes também chamado de umber) e UAA é ocre. Os códons de parada também são chamados de "terminação" ou "absurdo" códons. Eles sinalizam a liberação do polipeptídeo nascente do ribossomo porque nenhum tRNA cognato possui anticódons complementares a esses sinais de parada, permitindo que um fator de liberação se ligue ao ribossomo.
Efeito das mutações
Durante o processo de replicação do DNA, ocasionalmente ocorrem erros na polimerização da segunda fita. Esses erros, mutações, podem afetar o fenótipo de um organismo, especialmente se ocorrerem dentro da sequência de codificação da proteína de um gene. As taxas de erro são normalmente de 1 erro em cada 10 a 100 milhões de bases - devido à "revisão" capacidade das DNA polimerases.
Mutações sem sentido e mutações sem sentido são exemplos de mutações pontuais que podem causar doenças genéticas, como doença falciforme e talassemia, respectivamente. As mutações sem sentido clinicamente importantes geralmente alteram as propriedades do resíduo de aminoácido codificado entre os estados básico, ácido, polar ou não polar, enquanto as mutações sem sentido resultam em um códon de parada.
Mutações que interrompem a sequência do quadro de leitura por indels (inserções ou deleções) de um não múltiplo de 3 bases de nucleotídeos são conhecidas como mutações de desvio de quadro. Essas mutações geralmente resultam em uma tradução completamente diferente da original e provavelmente causam a leitura de um códon de parada, que trunca a proteína. Essas mutações podem prejudicar a função da proteína e, portanto, são raras em sequências de codificação de proteínas in vivo. Uma razão pela qual a herança de mutações frameshift é rara é que, se a proteína que está sendo traduzida é essencial para o crescimento sob as pressões seletivas que o organismo enfrenta, a ausência de uma proteína funcional pode causar a morte antes que o organismo se torne viável. As mutações frameshift podem resultar em doenças genéticas graves, como a doença de Tay-Sachs.
Embora a maioria das mutações que alteram as sequências de proteínas sejam prejudiciais ou neutras, algumas mutações têm benefícios. Essas mutações podem permitir que o organismo mutante resista a determinados estresses ambientais melhor do que os organismos do tipo selvagem ou se reproduza mais rapidamente. Nesses casos, uma mutação tenderá a se tornar mais comum em uma população por meio da seleção natural. Os vírus que usam o RNA como material genético têm taxas de mutação rápidas, o que pode ser uma vantagem, pois esses vírus evoluem rapidamente e, assim, evitam as respostas defensivas do sistema imunológico. Em grandes populações de organismos de reprodução assexuada, por exemplo, E. coli, múltiplas mutações benéficas podem ocorrer simultaneamente. Esse fenômeno é chamado de interferência clonal e causa competição entre as mutações.
Degeneração
Degenerescência é a redundância do código genético. Este termo foi dado por Bernfield e Nirenberg. O código genético tem redundância, mas sem ambigüidade (consulte as tabelas de códons abaixo para obter a correlação completa). Por exemplo, embora os códons GAA e GAG especifiquem o ácido glutâmico (redundância), nenhum deles especifica outro aminoácido (sem ambigüidade). Os códons que codificam um aminoácido podem diferir em qualquer uma de suas três posições. Por exemplo, o aminoácido leucina é especificado por YUR ou CUN (UUA, UUG, CUU, CUC, CUA ou CUG) códons (diferença na primeira ou terceira posição indicada usando a notação IUPAC), enquanto o aminoácido serina é especificado por UCN ou AGY (UCA, UCG, UCC, códons UCU, AGU ou AGC) (diferença na primeira, segunda ou terceira posição). Uma consequência prática da redundância é que erros na terceira posição do códon triplete causam apenas uma mutação silenciosa ou um erro que não afetaria a proteína porque a hidrofilicidade ou hidrofobicidade é mantida pela substituição equivalente de aminoácidos; por exemplo, um códon de NUN (onde N = qualquer nucleotídeo) tende a codificar aminoácidos hidrofóbicos. NCN produz resíduos de aminoácidos que são pequenos em tamanho e moderados em hidropaticidade; NAN codifica resíduos hidrofílicos de tamanho médio. O código genético é tão bem estruturado para hidropaticidade que uma análise matemática (decomposição de valor singular) de 12 variáveis (4 nucleotídeos x 3 posições) produz uma correlação notável (C = 0,95) para prever a hidropaticidade do aminoácido codificado diretamente do sequência nucleotídica triplete, sem tradução. Observe na tabela abaixo que oito aminoácidos não são afetados por mutações na terceira posição do códon, enquanto na figura acima, uma mutação na segunda posição provavelmente causará uma mudança radical nas propriedades físico-químicas do aminoácido codificado. No entanto, mudanças na primeira posição dos códons são mais importantes do que mudanças na segunda posição em escala global. A razão pode ser que a reversão de carga (de uma carga positiva para negativa ou vice-versa) só pode ocorrer após mutações na primeira posição de certos códons, mas não após mudanças na segunda posição de qualquer códon. Tal reversão de carga pode ter consequências dramáticas para a estrutura ou função de uma proteína. Este aspecto pode ter sido amplamente subestimado por estudos anteriores.
Viés de uso de códons
A frequência de códons, também conhecida como viés de uso de códons, pode variar de espécie para espécie com implicações funcionais para o controle da tradução. O códon varia de acordo com o organismo; por exemplo, o códon de prolina mais comum em E. coli é CCG, enquanto em humanos este é o códon de prolina menos usado.
Tabela de frequência de códon do genoma humano | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Códigos genéticos alternativos
Aminoácidos não padronizados
Em algumas proteínas, aminoácidos não padrão são substituídos por códons de parada padrão, dependendo das sequências de sinal associadas no RNA mensageiro. Por exemplo, UGA pode codificar selenocisteína e UAG pode codificar pirrolisina. A selenocisteína passou a ser vista como o 21º aminoácido e a pirrolisina como o 22º. Ao contrário da selenocisteína, o UAG codificado por pirrolisina é traduzido com a participação de uma aminoacil-tRNA sintetase dedicada. Tanto a selenocisteína quanto a pirrolisina podem estar presentes no mesmo organismo. Embora o código genético seja normalmente fixo em um organismo, o procarioto achaeal Acetohalobium arabaticum pode expandir seu código genético de 20 para 21 aminoácidos (incluindo pirrolisina) sob diferentes condições de crescimento.
Variações

Havia originalmente um argumento simples e amplamente aceito de que o código genético deveria ser universal: ou seja, que qualquer variação no código genético seria letal para o organismo (embora Crick tivesse afirmado que os vírus eram uma exceção). Isso é conhecido como "acidente congelado" argumento para a universalidade do código genético. No entanto, em seu artigo seminal sobre as origens do código genético em 1968, Francis Crick ainda afirmou que a universalidade do código genético em todos os organismos era uma suposição não comprovada e provavelmente não era verdadeira em alguns casos. Ele previu que "O código é universal (o mesmo em todos os organismos) ou quase isso". A primeira variação foi descoberta em 1979, por pesquisadores que estudavam genes mitocondriais humanos. Muitas pequenas variantes foram descobertas depois disso, incluindo vários códigos mitocondriais alternativos. Essas variantes secundárias, por exemplo, envolvem a tradução do códon UGA como triptofano em espécies de Mycoplasma e a tradução de CUG como uma serina em vez de leucina em leveduras do "CTG clade" (como Candida albicans). Como os vírus devem usar o mesmo código genético de seus hospedeiros, as modificações no código genético padrão podem interferir na síntese ou no funcionamento da proteína viral. No entanto, vírus como os totivírus se adaptaram à modificação do código genético do hospedeiro. Em bactérias e archaea, GUG e UUG são códons de iniciação comuns. Em casos raros, certas proteínas podem usar códons de início alternativos. Surpreendentemente, existem variações na interpretação do código genético também em genes humanos codificados no núcleo: em 2016, pesquisadores que estudam a tradução da malato desidrogenase descobriram que em cerca de 4% dos mRNAs que codificam essa enzima, o códon de parada é usado naturalmente para codificar o aminoácidos triptofano e arginina. Esse tipo de recodificação é induzido por um contexto de códon de parada de alta leitura e é referido como leitura translacional funcional.
Apesar dessas diferenças, todos os códigos de ocorrência natural conhecidos são muito semelhantes. O mecanismo de codificação é o mesmo para todos os organismos: códons de três bases, tRNA, ribossomos, leitura de direção única e tradução de códons únicos em aminoácidos únicos. As variações mais extremas ocorrem em certos ciliados, onde o significado dos stop códons depende de sua posição dentro do mRNA. Quando perto do 3' No final, eles agem como terminadores enquanto em posições internas eles codificam aminoácidos como em Condylostoma magnum ou desencadeiam frameshifting ribossomal como em Euplotes.
As origens e variações do código genético, incluindo os mecanismos por trás da evolubilidade do código genético, têm sido amplamente estudadas, e alguns estudos foram feitos evoluindo experimentalmente o código genético de alguns organismos.
Inferência
Códigos genéticos variantes usados por um organismo podem ser inferidos identificando genes altamente conservados codificados nesse genoma e comparando seu uso de códons com os aminoácidos em proteínas homólogas de outros organismos. Por exemplo, o programa FACIL infere um código genético pesquisando quais aminoácidos em domínios de proteínas homólogas são mais frequentemente alinhados a cada códon. As probabilidades de aminoácidos resultantes (ou códon de parada) para cada códon são exibidas em um logotipo de código genético.
A partir de janeiro de 2022, a pesquisa mais completa de códigos genéticos é feita por Shulgina e Eddy, que examinaram 250.000 genomas procarióticos usando sua ferramenta Codetta. Esta ferramenta usa uma abordagem semelhante ao FACIL com um banco de dados Pfam maior. Apesar do NCBI já fornecer 33 tabelas de tradução, os autores foram capazes de encontrar novas 5 variações de código genético (corroboradas por mutações de tRNA) e corrigir várias atribuições incorretas.
Origem
O código genético é uma parte fundamental da história da vida, de acordo com uma versão de que moléculas de RNA autorreplicantes precederam a vida como a conhecemos. Esta é a hipótese do mundo do RNA. Nessa hipótese, qualquer modelo de surgimento do código genético está intimamente relacionado a um modelo de transferência de ribozimas (enzimas de RNA) para proteínas como as principais enzimas nas células. De acordo com a hipótese do mundo do RNA, as moléculas de RNA de transferência parecem ter evoluído antes das modernas aminoacil-tRNA sintetases, de modo que este último não pode fazer parte da explicação de seus padrões.
Um código genético hipotético desenvolvido aleatoriamente motiva ainda mais um modelo bioquímico ou evolutivo para sua origem. Se os aminoácidos fossem atribuídos aleatoriamente a códons tripletos, haveria 1,5 × 1084 códigos genéticos possíveis. Esse número é encontrado calculando o número de maneiras pelas quais 21 itens (20 aminoácidos mais uma parada) podem ser colocados em 64 compartimentos, em que cada item é usado pelo menos uma vez. No entanto, a distribuição de atribuições de códons no código genético não é aleatória. Em particular, o código genético agrupa certas atribuições de aminoácidos.
Os aminoácidos que compartilham a mesma via biossintética tendem a ter a mesma primeira base em seus códons. Isso pode ser uma relíquia evolutiva de um código genético inicial mais simples, com menos aminoácidos, que mais tarde evoluiu para codificar um conjunto maior de aminoácidos. Também pode refletir propriedades estéricas e químicas que tiveram outro efeito no códon durante sua evolução. Aminoácidos com propriedades físicas semelhantes também tendem a ter códons semelhantes, reduzindo os problemas causados por mutações pontuais e erros de tradução.
Dado o esquema de codificação de tripleto genético não aleatório, uma hipótese sustentável para a origem do código genético poderia abordar vários aspectos da tabela de códons, como ausência de códons para D-aminoácidos, padrões de códons secundários para alguns aminoácidos, confinamento de posições sinônimas na terceira posição, o pequeno conjunto de apenas 20 aminoácidos (em vez de um número que se aproxima de 64) e a relação dos padrões de códons de parada com os padrões de codificação de aminoácidos.
Três hipóteses principais abordam a origem do código genético. Muitos modelos pertencem a um deles ou a um híbrido:
- congelamento aleatório: o código genético foi criado aleatoriamente. Por exemplo, os ribozymes iniciais do tipo tRNA podem ter tido diferentes afinidades para aminoácidos, com bacalhau surgindo de outra parte do ribozyme que exibiu variabilidade aleatória. Uma vez que os peptídeos suficientes foram codificados, qualquer grande mudança aleatória no código genético teria sido letal; daí tornou-se "congelado".
- Afinidade estereoquímica: o código genético é resultado de uma alta afinidade entre cada aminoácido e seu códon ou anti-codon; a última opção implica que as moléculas pré-tRNA combinaram seus aminoácidos correspondentes por esta afinidade. Mais tarde durante a evolução, essa correspondência foi gradualmente substituída por correspondência por sintetases de aminoacyl-tRNA.
- Otimidade: o código genético continuou a evoluir após sua criação inicial, de modo que o código atual maximiza alguma função de fitness, geralmente algum tipo de minimização de erros.
As hipóteses abordaram uma variedade de cenários:
- Os princípios químicos governam a interação específica do RNA com aminoácidos. Experimentos com aptamers mostraram que alguns aminoácidos têm uma afinidade química seletiva para seus codões. Os experimentos mostraram que de 8 aminoácidos testados, 6 mostram alguma associação de RNA triplet-amino ácido.
- Expansão biossintética. O código genético cresceu de um código anterior mais simples através de um processo de "expansão biossintética". A vida Primordial "descobriu" novos aminoácidos (por exemplo, como subprodutos do metabolismo) e mais tarde incorporou alguns destes na maquinaria da codificação genética. Embora muitas evidências circunstanciais tenham sido encontradas para sugerir que menos tipos de aminoácidos foram usados no passado, hipóteses precisas e detalhadas sobre as quais os aminoácidos entraram no código em que ordem são controversos. No entanto, vários estudos sugeriram que Gly, Ala, Asp, Val, Ser, Pro, Glu, Leu, Thr pode pertencer a um grupo de aminoácidos de adição precoce, enquanto Cys, Met, Tyr, Trp, His, Phe pode pertencer a um grupo de aminoácidos de posterior adição.
- A seleção natural levou a tarefas de codon do código genético que minimizam os efeitos das mutações. Uma hipótese recente sugere que o código trigêmeo foi derivado de códigos que usavam mais do que os códigos trigêmeos (como os codões quadruplet). Mais tempo do que a decodificação tripla aumentaria a redundância de codon e seria mais resistente a erros. Esta característica poderia permitir a decodificação precisa de máquinas translacionais complexas ausentes, como o ribossoma, como antes das células começaram a fazer ribossomas.
- Canais de informação: As abordagens teóricas da informação modelam o processo de tradução do código genético em aminoácidos correspondentes como um canal de informação propenso a erros. O ruído inerente (isto é, o erro) no canal coloca o organismo com uma pergunta fundamental: como pode um código genético ser construído para suportar o ruído, ao mesmo tempo que traduz informações com precisão e eficiência? Estes modelos de "distorção de taxa" sugerem que o código genético originou-se como resultado da interação das três forças evolucionárias conflitantes: as necessidades de aminoácidos diversos, para tolerância a erros e para o custo mínimo de recursos. O código emerge em uma transição quando o mapeamento de bacalhau para aminoácidos se torna não aleatório. A emergência do código é governada pela topologia definida pelos erros prováveis e está relacionada com o problema de coloração do mapa.
- Teoria do jogo: Modelos baseados em jogos de sinalização combinam elementos da teoria do jogo, seleção natural e canais de informação. Tais modelos têm sido usados para sugerir que os primeiros polipeptídeos eram provavelmente curtos e tinham função não-enzima. Os modelos teóricos do jogo sugeriram que a organização de cadeias de RNA em células pode ter sido necessária para evitar o uso "deceptivo" do código genético, ou seja, impedir o antigo equivalente de vírus de esmagar o mundo do RNA.
- Pare de bacalhau. Os codons para paradas translacionais também são um aspecto interessante para o problema da origem do código genético. Como um exemplo para abordar a evolução do codon de parada, sugeriu-se que os codons de parada são tais que eles são mais propensos a encerrar a tradução no início do caso de um erro de mudança de quadro. Em contraste, alguns modelos moleculares estereoquímicos explicam a origem de bacalhau de parada como "insignável".