Virus del mosaico de la coliflor
virus del mosaico de la coliflor (CaMV) es un miembro del género Caulimovirus, uno de los seis géneros de la familia Caulimoviridae. , que son pararetrovirus que infectan las plantas. Los pararetrovirus se replican mediante transcripción inversa al igual que los retrovirus, pero las partículas virales contienen ADN en lugar de ARN.
Definición

El virus del mosaico de la coliflor (CaMV) es un miembro de la familia Caulimoviridae. Esta familia se agrupa junto con los Belpaoviridae, Metaviridae, Pseudoviridae y Retroviridae (todos los cuales tienen en cambio una Genoma de ARN replicado a través de un intermediario de ADN) en el orden Ortervirales; los Hepadnaviridae, a pesar de tener un genoma de ADN replicado a través de un intermediario de ARN (como los Caulimoviridae), están relacionados más lejanamente y pertenecen al orden separado Blubervirales > (ambas órdenes pertenecen a la misma clase, las Revtraviricetes).
CaMV infecta principalmente plantas de la familia Brassicaceae (como la coliflor y el nabo), pero algunas cepas de CaMV (D4 y W260) también pueden infectar especies de Solanaceae de los géneros Datura y Nicotiana. . CaMV induce una variedad de síntomas sistémicos como mosaico, lesiones necróticas en la superficie de las hojas, retraso en el crecimiento y deformación de la estructura general de la planta. Los síntomas mostrados varían según la cepa viral, el ecotipo del huésped y las condiciones ambientales.
CaMV se transmite de forma no circulatoria por especies de pulgones como Myzus persicae. Una vez introducidos dentro de una célula vegetal huésped, los viriones migran a la envoltura nuclear de la célula vegetal.
Estructura
La partícula CaMV es un icosaedro con un diámetro de 52 nm construido a partir de 420 subunidades de proteína de la cápside (CP) dispuestas con una triangulación T = 7, que rodea una cavidad central llena de disolvente.
CaMV contiene una molécula de ADN circular de doble cadena de aproximadamente 8,0 kilobases, interrumpida por mellas que resultan de las acciones de la ARNasa H durante la transcripción inversa. Estas mellas provienen del Met-tRNA y de dos cebadores de ARN utilizados en la transcripción inversa. Después de ingresar a la célula huésped, estos "nicks" en el ADN viral se reparan, formando una molécula superenrollada que se une a las histonas. Este ADN se transcribe en un ARN 35S de longitud completa, terminalmente redundante, y un ARN 19S subgenómico.
Genoma
El promotor del ARN 35S es un promotor constitutivo muy fuerte responsable de la transcripción de todo el genoma CaMV. Es bien conocido por su uso en la transformación de plantas. Provoca altos niveles de expresión genética en plantas dicotiledóneas. Sin embargo, es menos eficaz en monocotiledóneas, especialmente en cereales. Las diferencias de comportamiento probablemente se deben a diferencias en la calidad y/o cantidad de factores regulatorios. Un estudio reciente ha indicado que el promotor CaMV 35S también es funcional en algunas células animales, aunque los elementos promotores utilizados son diferentes de los de las plantas. Si bien este promotor tenía una actividad baja en comparación con los promotores animales canónicos, los niveles de productos indicadores fueron significativos. Esta observación sugiere que el promotor 35S puede tener potencial para su uso en animales.
El promotor se denominó promotor CaMV 35S ("promotor 35S") porque el coeficiente de sedimentación del transcrito viral, cuya expresión es impulsada naturalmente por este promotor, es 35S. Es uno de los promotores constitutivos de propósito general más utilizados. Fue descubierto a principios de la década de 1980 por Chua y colaboradores de la Universidad Rockefeller.
El ARN 35S es particularmente complejo y contiene una secuencia líder altamente estructurada de 600 nucleótidos de largo con seis a ocho marcos de lectura abiertos (ORF) cortos.
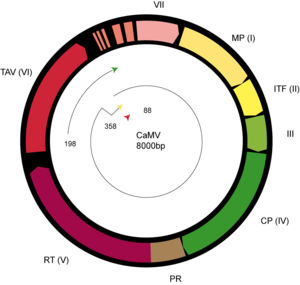
A este líder le siguen siete ORF más largos y estrechamente dispuestos que codifican todas las proteínas virales. El mecanismo de expresión de estas proteínas es único, ya que la proteína ORF VI (codificada por el ARN 19S) controla el reinicio de la traducción de los principales marcos de lectura abiertos en el ARN policistrónico 35S, un proceso que normalmente sólo ocurre en los ARNm bacterianos. La función de TAV depende de su asociación con polisomas y el factor de iniciación eucariota eIF3.
- ORF I – P1: proteína de movimientoP03545)
- ORF II – P2: factor de transmisión de áfido/insecto (P03548)
- ORF III – P3: proteína asociada al virión (VAP, P03551): proteína estructural, capacidades de unión de ADN
- ORF IV – P4: proteína capsida (CP, P03542)
- ORF V – P5: pro-polP03554): proteasa, transcripción inversa bifuncional y RNase H
- ORF VI – P6: transactivador/viroplasmin ()P03559): cuerpo de inclusión Formación/trata; posiblemente otras funciones (ver texto)
- ORF VII/VIII – desconocido (aparece nota que se requiere para la infección, Q83163, Q83164)
- Contiene un sitio de unión tRNA-Met
Además de sus funciones relacionadas con la activación traslacional y la formación de cuerpos de inclusión, se ha demostrado que P6 interactúa con otras proteínas CaMV, como P2 y P3, lo que sugiere que también puede contribuir en cierto grado al ensamblaje viral y Transmisión mediada por pulgones. Además, se ha demostrado que P6 se une a P7; Investigar las interacciones entre los dos puede ayudar a dilucidar la función aún desconocida de P7.
Otra función de P6 implica la modificación del NO EXPRESOR DE PATOGENIA RELACIONADO 1 (NPR1) del huésped durante el curso de la infección. NPR1 es un regulador importante de la señalización dependiente del ácido salicílico (SA) y del ácido jasmónico (JA), y está más estrechamente asociado con la diafonía entre los dos. La modificación de NPR1 sirve para inhibir las respuestas defensivas de las células vegetales al prevenir la señalización dependiente de SA; NPR1 modificado puede transportarse adecuadamente al núcleo y unirse al promotor PR-1, pero no puede iniciar la transcripción. Debido a que se requiere NPR1 activo para la acumulación de SA, esto conduce a un mayor agotamiento de SA. Mientras que la regulación de la señalización dependiente de SA por NPR1 modificado con P6 se localiza en el núcleo, la regulación de la señalización dependiente de JA es de naturaleza citoplasmática e involucra la vía COI1. A diferencia de SA, la señalización dependiente de JA aumenta en presencia de NPR1 modificado.
Replicación

CaMV se replica mediante transcripción inversa:
- Las partículas virales entran en una célula vegetal y no están encapsidadas. En esta etapa el ADN viral consta de tres fragmentos, uno en el – hilo (α) y dos en el + hilo (β y γ) que se montan imperfectamente en un genoma circular con tres huecos o discontinuidades (D1, D2, y D3).
- El ADN viral entra en el núcleo donde se llenan las discontinuidades. En este punto el ADN viral también se asocia con histonas anfitrionas, formando un minicromoso (no se muestra).
- La polimerasa RNA dependiente del ADN anfitriones transscribe del promotor 35S todo el camino alrededor del genoma viral, superando al promotor 35S. (Esto crea dos copias del promotor 35S en el ARN resultante.) La transcripción también inicia en el promotor 19S (no se muestra).
- Los ARN virales pasan al citoplasma host donde se transcriben.
- El 3′ extremo de un tRNAf Met anneals to a site corresponding to discontinuity 1 (D1) near the 5′ end of the 35S RNA.
- El tRNAf Met síntesis de primos, por la transcripción inversa viral (codificada por ORF V), de una nueva cadena α.
- RNase H quita el ARN del dúplex ADN-RNA, dejando detrás del ADN.
- Este nuevo ADN une al promotor de 35S en el extremo 3′ de la plantilla de ARN y la síntesis del hilo α del ADN continúa y RNase H continúa degradando el ARN complejo al ADN.
- Sintesis del hilo α completa. La actividad RNase H expone regiones ricas en purina en la posición de discontinuidad 3 (D3), que prepara la síntesis de la cadena de ADN γ.
- La actividad de RNase H expone regiones ricas en purina en la posición de discontinuidad 2 (D2), que prepara la síntesis de la cadena de ADN β. Cuando la nueva cadena γ de ADN alcanza el extremo 5′ de la nueva cadena α cambia al extremo 5′ del nuevo hilo α, recreando la discontinuidad 1 (D1). Cuando la nueva cadena γ de ADN alcanza el extremo 5′ de la nueva cadena β, desplaza la imprimación y algunos de la nueva cadena β sintetizada, dando lugar a la recreación de la discontinuidad 2 (D2). Cuando la nueva cadena β de ADN alcanza el extremo 5′ del nuevo hilo γ, desplaza la imprimación y algunos de los hilos γ recién sintetizados, resultando en la recreación de la discontinuidad 3 (D3).
En este punto, el nuevo genoma viral puede empaquetarse en cápsides y liberarse de la célula o pueden transportarse mediante proteínas de movimiento a una célula adyacente no infectada.
El promotor del virus del mosaico de la coliflor (CaMV 35S) se utiliza en la mayoría de los cultivos transgénicos para activar genes extraños que se han insertado artificialmente en la planta huésped. Se inserta en plantas transgénicas en una forma diferente de la que se encuentra cuando está presente en sus plantas huéspedes naturales Brassica. Esto le permite operar en una amplia gama de entornos de organismos huéspedes que de otro modo no serían posibles.
CaMV contiene aproximadamente 8 kb de genoma de ADN de doble hebra y produce partículas esféricas. Las infecciones por CaMV son sistémicas e incluso su ADN es infeccioso cuando se inocula en superficies desgastadas de plantas. El genoma del CaMV tiene ocho genes muy agrupados, de los cuales sólo dos genes pequeños, los genes II y VII, no son esenciales; como resultado, sólo estos dos genes pueden reemplazarse o eliminarse sin pérdida de infectividad. Además, los genomas de CaMV modificados que exceden el tamaño del genoma natural (8024 pb) incluso en unos pocos cientos de pb no están empaquetados en viriones. Estos dos factores limitan seriamente el tamaño del inserto de ADN clonable en CaMV. El gen bacteriano de la dihidrofolato reductasa DHFR se ha clonado con éxito en el genoma CaMV, en lugar del gen II, y se ha expresado con éxito en plantas.
Mecanismos moleculares de transmisión de CaMV mediada por vectores
El virus se adquiere de un huésped infectado durante la alimentación del pulgón vector. Para que se produzca, un complejo transmisible está compuesto por viriones y proteína P2 ubicada en los estiletes del vector. El dominio P2 N-terminal reconoce un receptor proteico ubicado en la punta del estilete y el dominio P2 C-terminal se une a los viriones decorados con P3.
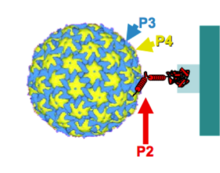
El modo de adquisición por el vector es controlado por el tejido y localización intracelular específica de P2. Esta proteína sólo se encuentra en las células epidermis y parenquima. Además, en estas células, P2 se localiza en cuerpos de inclusión electron-lucent únicos virales (ELIB). En las células anfitrionas, las proteínas virales P2 y P3 se producen por primera vez en numerosas fábricas virales (órganos de inclusión condensados por electrón), y posteriormente se exportan y colocalizan con microtúbulos, antes de concentrarse en ELIB. CaMV utiliza específicamente los microtúbulos para formar el cuerpo transmisible y así permitir la transmisión vectorial. La caracterización molecular completa y el estudio de este virus no se llevaron más lejos.
Evasión de las defensas de las plantas
El virus del mosaico de la coliflor posee una serie de mecanismos que le permiten contrarrestar las defensas de las células de la planta huésped. Si bien el ARN pregenómico 35S es responsable de la replicación del genoma mediante la transcriptasa inversa, también contiene una secuencia líder no codificante de 600 pares de bases que sirve como un ARNm importante para la producción de factores implicados en la contradefensa viral. Varios huéspedes de CaMV poseen pequeños mecanismos de silenciamiento viral basados en ARN que sirven para limitar la infección viral. Los productos de la secuencia de 600 pb antes mencionada son pequeños ARN virales (vsRNA) de 21, 22 y 24 nucleótidos de longitud que sirven como señuelos, efectores de unión e inactivación de la maquinaria silenciadora del huésped, como Argonaute 1 (AGO1). Como prueba de principio, la sobreexpresión experimental de estos vsRNA permite una mayor acumulación viral en plantas infectadas.
Preocupaciones sobre el uso del promotor CaMV 35S en plantas transgénicas
A principios de la década de 2010, surgieron algunas preocupaciones sobre el uso del promotor CaMV 35S para la expresión en plantas transgénicas porque existe una superposición de secuencias entre este promotor y las secuencias codificantes de P6. Cincuenta y cuatro eventos transgénicos certificados para su liberación en los EE. UU. contienen hasta 528 pb de ORF VI (que codifica los dominios C-terminales de P6). Como P6 es una proteína multifuncional cuya gama completa de funciones se desconoce, existe cierta preocupación de que la expresión de uno o más de sus dominios pueda tener consecuencias imprevistas en los organismos transgénicos. Estudios recientes han intentado determinar qué longitud del promotor CaMV 35S tiene la menor probabilidad de producir inadvertidamente dominios P6, manteniendo al mismo tiempo la actividad total del promotor. Como era de esperar, el uso de longitudes de promotor más cortas disminuye la cantidad de dominios P6 incluidos y también disminuye la probabilidad de efectos no deseados.
Contenido relacionado
Ley de Fick
Precisión y exactitud
Evidencia empírica