Virus del herpes simple
Virus del herpes simple 1 y 2 (HSV-1 y HSV-2), también conocidos por sus nombres taxonómicos Alfaherpesvirus humano 1 y Alfaherpesvirus humano 2, son dos miembros de la familia Herpesviridae humana, un conjunto de virus que producen infecciones virales en la mayoría de los humanos. Tanto el HSV-1 como el HSV-2 son muy comunes y contagiosos. Pueden transmitirse cuando una persona infectada comienza a eliminar el virus.
En 2016, alrededor del 67% de la población mundial menor de 50 años tenía HSV-1. En los Estados Unidos, se estima que alrededor del 47,8 % y el 11,9 % tienen HSV-1 y HSV-2, respectivamente, aunque la prevalencia real puede ser mucho mayor. Debido a que puede transmitirse a través de cualquier contacto íntimo, es una de las infecciones de transmisión sexual más comunes.
Síntomas
Muchos de los infectados nunca desarrollan síntomas. Los síntomas, cuando ocurren, pueden incluir ampollas acuosas en la piel de cualquier parte del cuerpo o en las membranas mucosas de la boca, los labios, la nariz, los genitales o los ojos (queratitis por herpes simple). Las lesiones cicatrizan con una costra característica de la enfermedad herpética. A veces, los virus causan síntomas leves o atípicos durante los brotes. Sin embargo, también pueden causar formas más problemáticas de herpes simple. Como virus neurotrópicos y neuroinvasivos, HSV-1 y -2 persisten en el cuerpo ocultándose del sistema inmunológico en los cuerpos celulares de las neuronas, particularmente en los ganglios sensoriales. Después de la infección inicial o primaria, algunas personas infectadas experimentan episodios esporádicos de reactivación o brotes virales. En un brote, el virus en una célula nerviosa se activa y se transporta a través del axón de la neurona a la piel, donde se produce la replicación y eliminación del virus y puede causar nuevas llagas.
Transmisión
HSV-1 y HSV-2 se transmiten por contacto con una persona infectada que tiene reactivaciones del virus. HSV 1 y HSV-2 se eliminan periódicamente, con mayor frecuencia de forma asintomática.
En un estudio de personas con un primer episodio de infección genital por HSV-1 realizado en 2022, la excreción genital de HSV-1 se detectó en el 12 % de los días a los 2 meses y disminuyó significativamente al 7 % de los días a los 11 meses. La mayor parte de la muda genital fue asintomática; las lesiones genitales y orales y la excreción oral fueron raras.
La mayoría de las transmisiones sexuales del HSV-2 ocurren durante períodos de excreción asintomática. La reactivación asintomática significa que el virus causa síntomas atípicos, sutiles o difíciles de notar que no se identifican como una infección activa por herpes, por lo que es posible contraer el virus incluso si no hay ampollas o llagas activas por HSV. En un estudio, las muestras diarias de hisopos genitales detectaron HSV-2 en una mediana de 12 a 28 % de los días entre aquellos que tuvieron un brote y 10 % de los días entre aquellos con infección asintomática (sin brotes previos), siendo muchos de estos episodios que ocurre sin brote visible ("eliminación subclínica").
En otro estudio, 73 sujetos fueron asignados al azar para recibir valaciclovir 1 g diario o placebo durante 60 días cada uno en un diseño cruzado de dos vías. Se recogió un hisopo diario del área genital para la detección del HSV-2 mediante reacción en cadena de la polimerasa, para comparar el efecto de valaciclovir versus placebo sobre la diseminación viral asintomática en sujetos inmunocompetentes seropositivos para el HSV-2 sin antecedentes de infección sintomática por herpes genital. El estudio encontró que valaciclovir redujo significativamente la eliminación durante los días subclínicos en comparación con el placebo, mostrando una reducción del 71 %; El 84% de los sujetos no tuvo pérdida de secreción mientras recibían valaciclovir frente al 54% de los sujetos que recibían placebo. Alrededor del 88% de los pacientes tratados con valaciclovir no tuvieron signos o síntomas reconocidos frente al 77% de los que recibieron placebo.
En el caso del HSV-2, la diseminación subclínica puede representar la mayor parte de la transmisión. Los estudios sobre parejas discordantes (una infectada con HSV-2, otra no) muestran que la tasa de transmisión es aproximadamente de 5 a 8,9 por cada 10.000 contactos sexuales, y el uso de condones reduce en gran medida el riesgo de adquisición. Los síntomas atípicos a menudo se atribuyen a otras causas, como una candidiasis. El HSV-1 suele adquirirse por vía oral durante la infancia. También puede transmitirse sexualmente, incluido el contacto con la saliva, como los besos y el sexo oral. Históricamente, el VHS-2 era principalmente una infección de transmisión sexual, pero las tasas de infecciones genitales por VHS-1 han aumentado en las últimas décadas.
Ambos virus también pueden transmitirse verticalmente durante el parto. Sin embargo, el riesgo de transmisión es mínimo si la madre no presenta síntomas ni ampollas expuestas durante el parto. El riesgo es considerable cuando la madre se infecta con el virus por primera vez durante la última etapa del embarazo, lo que refleja una alta carga viral. Si bien la mayoría de las ETS virales no se pueden transmitir a través de objetos ya que el virus muere rápidamente fuera del cuerpo, el VHS puede sobrevivir hasta 4,5 horas en las superficies y puede transmitirse mediante el uso de toallas, cepillos de dientes, tazas, cubiertos, etc.
Los virus del herpes simple pueden afectar áreas de la piel expuestas al contacto con una persona infectada. Un ejemplo de esto es el panadizo herpético, que es una infección por herpes en los dedos; se encontraba comúnmente en las manos del cirujano dental antes del uso rutinario de guantes al tratar a los pacientes. Darle la mano a una persona infectada no transmite esta enfermedad. La infección genital por HSV-2 aumenta el riesgo de contraer VIH.
Virología
HSV ha sido un virus modelo para muchos estudios en biología molecular. Por ejemplo, uno de los primeros promotores funcionales en eucariotas se descubrió en el VHS (del gen de la timidina quinasa) y la proteína del virión VP16 es uno de los activadores transcripcionales más estudiados.
Estructura viral

Todos los virus del herpes animal comparten algunas propiedades comunes. La estructura de los virus del herpes consiste en un genoma de ADN lineal, bicatenario y relativamente grande encerrado dentro de una jaula de proteínas icosaédrica llamada cápside, que está envuelta en una bicapa lipídica llamada envoltura. La envoltura está unida a la cápside mediante un tegumento. Esta partícula completa se conoce como virión. HSV-1 y HSV-2 contienen cada uno al menos 74 genes (o marcos de lectura abiertos, ORF) dentro de sus genomas, aunque la especulación sobre el hacinamiento de genes permite hasta 84 genes codificantes de proteínas únicos mediante 94 ORF putativos. Estos genes codifican una variedad de proteínas involucradas en la formación de la cápside, el tegumento y la envoltura del virus, así como en el control de la replicación y la infectividad del virus. Estos genes y sus funciones se resumen en la siguiente tabla.
Los genomas de HSV-1 y HSV-2 son complejos y contienen dos regiones únicas llamadas región única larga (UL) y región única corta (US >). De los 74 ORF conocidos, UL contiene 56 genes virales, mientras que US contiene solo 12. La transcripción de genes HSV está catalizada por la ARN polimerasa II del huésped infectado. Los genes tempranos inmediatos, que codifican proteínas, por ejemplo ICP22, que regulan la expresión de genes virales tempranos y tardíos, son los primeros que se expresan después de la infección. Sigue la expresión genética temprana, para permitir la síntesis de enzimas involucradas en la replicación del ADN y la producción de ciertas glicoproteínas de la envoltura. La expresión de genes tardíos ocurre al final; Este grupo de genes codifica predominantemente proteínas que forman la partícula del virión.
Cinco proteínas de (UL) forman la cápside viral: UL6, UL18, UL35, UL38 y la proteína principal de la cápside UL19.
Entrada celular
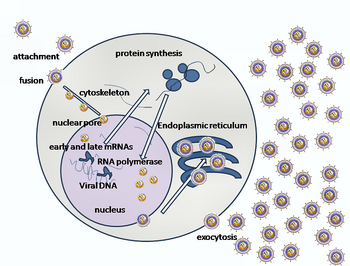
La entrada del HSV en una célula huésped implica que varias glicoproteínas en la superficie del virus envuelto se unen a sus receptores transmembrana en la superficie celular. Luego, la célula atrae muchos de estos receptores hacia adentro, lo que se cree que abre un anillo de tres heterodímeros gHgL que estabilizan una conformación compacta de la glicoproteína gB, de modo que salta y perfora la membrana celular. La envoltura que cubre la partícula viral luego se fusiona con la membrana celular, creando un poro a través del cual el contenido de la envoltura viral ingresa a la célula huésped.
Las etapas secuenciales de entrada del VHS son análogas a las de otros virus. Al principio, los receptores complementarios del virus y la superficie celular acercan las membranas viral y celular. Las interacciones de estas moléculas forman entonces un poro de entrada estable a través del cual el contenido de la envoltura viral se introduce en la célula huésped. El virus también puede ser endocitado después de unirse a los receptores, y la fusión podría ocurrir en el endosoma. En micrografías electrónicas, se han visto fusionadas las valvas externas de las bicapas lipídicas viral y celular; esta hemifusión puede estar en el camino habitual de entrada o puede ser generalmente un estado detenido con mayor probabilidad de ser capturado que un mecanismo de entrada transitorio.
En el caso de un virus del herpes, las interacciones iniciales ocurren cuando dos glicoproteínas de la envoltura viral llamadas glicoproteína C (gC) y glicoproteína B (gB) se unen a un polisacárido de la superficie celular llamado heparán sulfato. A continuación, la principal proteína de unión al receptor, la glicoproteína D (gD), se une específicamente a al menos uno de los tres receptores de entrada conocidos. Estos receptores celulares incluyen el mediador de entrada del herpesvirus (HVEM), la nectina-1 y el heparán sulfato sulfatado 3-O. Los receptores de nectina suelen producir adhesión célula-célula, para proporcionar un fuerte punto de unión del virus a la célula huésped. Estas interacciones acercan las superficies de las membranas y permiten que otras glicoproteínas incrustadas en la envoltura viral interactúen con otras moléculas de la superficie celular. Una vez unida a HVEM, gD cambia su conformación e interactúa con las glicoproteínas virales H (gH) y L (gL), que forman un complejo. La interacción de estas proteínas de membrana puede dar lugar a un estado de hemifusión. La interacción de gB con el complejo gH/gL crea un poro de entrada para la cápside viral. gB interactúa con los glucosaminoglicanos en la superficie de la célula huésped.
Inoculación genética
Después de que la cápside viral ingresa al citoplasma celular, comienza a expresar la proteína viral ICP27. ICP27 es una proteína reguladora que altera la síntesis de proteínas del huésped y la utiliza para la replicación viral. ICP27 se une a una enzima celular Serina-Arginina Proteína Quinasa 1, SRPK1. La formación de este complejo hace que SRPK1 se desplace del citoplasma al núcleo y el genoma viral se transporta al núcleo celular. Una vez unida al núcleo en un poro de entrada nuclear, la cápside expulsa su contenido de ADN a través del portal de la cápside. El portal de la cápsida está formado por 12 copias de la proteína portal, UL6, dispuestas en forma de anillo; Las proteínas contienen una secuencia de aminoácidos en cremallera de leucina, que les permite adherirse entre sí. Cada cápside icosaédrica contiene un único portal, ubicado en un vértice. El ADN sale de la cápside en un único segmento lineal.
Immune evasion
HSV evade el sistema inmunológico a través de la interferencia con la presentación del antígeno MHC clase I en la superficie celular, al bloquear el transportador asociado con el procesamiento de antígenos (TAP) inducido por la secreción de ICP-47 por HSV. En la célula huésped, TAP transporta péptidos de epítopos de antígenos virales digeridos desde el citosol al retículo endoplásmico, lo que permite que estos epítopos se combinen con moléculas de MHC de clase I y se presenten en la superficie de la célula. La presentación de epítopos virales con MHC clase I es un requisito para la activación de los linfocitos T citotóxicos (CTL), los principales efectores de la respuesta inmune mediada por células contra las células infectadas por virus. ICP-47 previene el inicio de una respuesta CTL contra HSV, lo que permite que el virus sobreviva durante un período prolongado en el huésped. El VHS suele producir un efecto citopático (CPE) entre 24 y 72 horas después de la infección en líneas celulares permisivas, que se observa mediante la formación de placas clásica. Sin embargo, también se han informado aislados clínicos de HSV-1 que no mostraron ningún CPE en cultivos de células Vero y A549 en varios pases con un nivel bajo de expresión de proteínas virales. Probablemente estos aislados de HSV-1 estén evolucionando hacia una forma más "críptica" forma para establecer una infección crónica, desentrañando así otra estrategia más para evadir el sistema inmunológico del huésped, además de la latencia neuronal.
Replicación

Después de la infección de una célula, se produce una cascada de proteínas del virus del herpes, denominadas inmediata-temprana, temprana y tardía. La investigación que utiliza citometría de flujo en otro miembro de la familia de los virus del herpes, el herpesvirus asociado al sarcoma de Kaposi, indica la posibilidad de una etapa lítica adicional, tardía. Estas etapas de la infección lítica, en particular la lítica tardía, son distintas de la etapa de latencia. En el caso del HSV-1, no se detectan productos proteicos durante la latencia, mientras que sí se detectan durante el ciclo lítico.
Las primeras proteínas transcritas se utilizan en la regulación de la replicación genética del virus. Al ingresar a la célula, una proteína α-TIF se une a la partícula viral y ayuda en la transcripción inmediata-temprana. La proteína de cierre del huésped del virión (VHS o UL41) es muy importante para la replicación viral. Esta enzima interrumpe la síntesis de proteínas en el huésped, degrada el ARNm del huésped, ayuda en la replicación viral y regula la expresión genética de las proteínas virales. El genoma viral viaja inmediatamente al núcleo, pero la proteína VHS permanece en el citoplasma.
Las proteínas tardías forman la cápside y los receptores en la superficie del virus. El empaquetado de las partículas virales (incluidos el genoma, el núcleo y la cápside) se produce en el núcleo de la célula. Aquí, los concatémeros del genoma viral se separan mediante escisión y se colocan en cápsides formadas. HSV-1 sufre un proceso de envolvimiento primario y secundario. La envoltura primaria se adquiere por gemación en la membrana nuclear interna de la célula. Luego se fusiona con la membrana nuclear externa. El virus adquiere su envoltura final mediante la gemación en vesículas citoplasmáticas.
Infección latente
Los VHS pueden persistir en una forma inactiva pero persistente conocida como infección latente, especialmente en los ganglios neurales. El ADN circular del genoma del HSV reside en el núcleo celular como un episoma. El VHS-1 tiende a residir en los ganglios del trigémino, mientras que el VHS-2 tiende a residir en los ganglios sacros, pero estas son tendencias históricas únicamente. Durante la infección latente de una célula, los HSV expresan ARN de transcripción asociada a latencia (LAT). LAT regula el genoma de la célula huésped e interfiere con los mecanismos naturales de muerte celular. Al mantener las células huésped, la expresión de LAT preserva un reservorio del virus, lo que permite recurrencias periódicas posteriores, generalmente sintomáticas, o "brotes" característica de la no latencia. Independientemente de que las recurrencias sean sintomáticas o no, la diseminación viral ocurre para infectar a un nuevo huésped.
Una proteína que se encuentra en las neuronas puede unirse al ADN del virus del herpes y regular la latencia. El ADN del virus del herpes contiene un gen para una proteína llamada ICP4, que es un importante transactivador de genes asociados con la infección lítica en HSV-1. Los elementos que rodean el gen de ICP4 se unen a una proteína conocida como factor de silenciamiento restrictivo neuronal de proteína neuronal humana (NRSF) o factor de transcripción silenciador de elemento represor humano (REST). Cuando se une a los elementos del ADN viral, se produce la desacetilación de histonas encima de la secuencia del gen ICP4 para impedir el inicio de la transcripción a partir de este gen, impidiendo así la transcripción de otros genes virales implicados en el ciclo lítico. Otra proteína del HSV revierte la inhibición de la síntesis de la proteína ICP4. ICP0 disocia NRSF del gen ICP4 y así evita el silenciamiento del ADN viral.
Genome
El genoma del HSV abarca aproximadamente 150.000 pb y consta de dos segmentos únicos, denominados único largo (UL) y único corto (US), así como repeticiones terminales invertidas que se encuentran en los dos extremos de ellos, denominadas repetición larga (RL) y repetir corto (RS). También existen problemas menores de "redundancia de terminal" (α) elementos que se encuentran en los extremos más alejados de RS. La disposición general es RL-UL-RL-α-RS-US-RS-α y cada par de repeticiones se invierten entre sí. Luego, toda la secuencia se encapsula en una repetición directa terminal. Cada una de las partes larga y corta tiene sus propios orígenes de replicación, con OriL ubicado entre UL28 y UL30 y OriS ubicado en un par cerca de RS. Como los segmentos L y S se pueden ensamblar en cualquier dirección, se pueden invertir libremente entre sí, formando varios isómeros lineales.
| ORF | Protein alias | HSV-1 | HSV-2 | Función/descripción |
|---|---|---|---|---|
| Repetir largo (R)L) | ||||
| ICP0/RL2 | ICP0; IE110; α0 | P08393 | P284 | El ligasón de ubiquitina E3 que activa la transcripción del gen viral al oponerse a la cromatinización del genoma viral y contrarresta respuestas antivirales intrínsecas e interferón. |
| RL1 | RL1; ICP34.5 | O12396 | P283 | Factor de neurovirulencia. Antagoniza PKR desfosforilando eIF4a. Acopla a BECN1 e inactiva la autofagia. |
| LAT | LRP1, LRP2 | P17588 P17589 | Latency-associated transcript abd protein products (latency-related protein) | |
| Único largo (UL) | ||||
| UL1 | Glycoproteína L | P10185 | P28278 | Superficie y membrana |
| UL2 | Uracil-DNA glycosylase | P10186 | P13158 P28275 | Uracil-DNA glycosylase |
| UL3 | UL3 | P10187 Q1XBW5 | P0C012 P28279 | desconocida |
| UL4 | UL4 | P10188 | P28280 | desconocida |
| UL5 | HELI | P10189 | P28277 | ADN helicase |
| UL6 | Proteína de portal UL-6 | P10190 | Doce de estas proteínas constituyen el anillo de portal capsid a través del cual el ADN entra y sale del capsid. | |
| UL7 | Proteína de envelopamiento citoplasmático 1 | P10191 | P89430 | maduración de Virion |
| UL8 | proteína helicasa/primas complejas asociadas | P10192 | P89431 | ADN virus helicase-primase proteína asociada |
| UL9 | Proteína de unión de origen replicación | P10193 | P89432 | Proteína de unión de origen replicación |
| UL10 | Glycoprotein M | P04288 | P89433 | Superficie y membrana |
| UL11 | proteína de envelopmento citoplasmático 3 | P04289 Q68980 | P13294 | virion exit and secondary envelopment |
| UL12 | nucleasa alcalina | P04294 | P06489 | Alkaline exonuclease |
| UL13 | UL13 | P04290 | P89436 | Kinase proteína serina-treonina |
| UL14 | UL14 | P04291 | P89437 | Proteína de tegumento |
| UL15 | TRM3 | P04295 | P89438 | Procesamiento y embalaje del ADN |
| UL16 | UL16 | P10200 | P89439 | Proteína de tegumento |
| UL17 | CVC1 | P10201 | Procesamiento y empaquetado de ADN | |
| UL18 | TRX2 | P10202 | P89441 | Proteína capsida |
| UL19 | VP5; ICP5 | P06491 | P89442 | proteína capsida mayor |
| UL20 | UL20 | P10204 | P89443 | Proteína de membrana |
| UL21 | UL21 | P10205 P09855 | P89444 | Proteína de tegumento |
| UL22 | Glycoproteína H | P06477 | P89445 | Superficie y membrana |
| UL23 | Thymidine kinase | O55259 | Reproducción de ADN periférica | |
| UL24 | UL24 | P10208 | desconocida | |
| UL25 | UL25 | P10209 | Procesamiento y empaquetado de ADN | |
| UL26 | P40; VP24; VP22A; UL26.5 (HV2 breve isoform) | P10210 | P89449 | Proteína capsida |
| UL27 | Glycoproteína B | A1Z0P5 | P08666 | Superficie y membrana |
| UL28 | ICP18.5 | P10212 | Procesamiento y empaquetado de ADN | |
| UL29 | UL29; ICP8 | Q2MGU6 | Principales proteínas de unión de ADN | |
| UL30 | polimerasa de ADN | Q4ACM2 | Replicación de ADN | |
| UL31 | UL31 | Q25BX0 | Proteína de matriz nuclear | |
| UL32 | UL32 | P10216 | Envelope glycoprotein | |
| UL33 | UL33 | P10217 | Procesamiento y empaquetado de ADN | |
| UL34 | UL34 | P10218 | Proteína interna de membrana nuclear | |
| UL35 | VP26 | P10219 | Proteína capsida | |
| UL36 | UL36 | P10220 | Proteína de tegumento grande | |
| UL37 | UL37 | P10216 | Montaje capsid | |
| UL38 | UL38; VP19C | P32888 | Montaje capsido y maduración de ADN | |
| UL39 | UL39; RR-1; ICP6 | P08543 | Ribonucleotide reductase (subunidad grande) | |
| UL40 | UL40; RR-2 | P06474 | Ribonucleotide reductase (pequeña subunidad) | |
| UL41 | UL41; VHS | P10225 | Tegument protein; virion host shutoff | |
| UL42 | UL42 | Q4H1G9 | Factor de procesador de polimerasa ADN | |
| UL43 | UL43 | P10227 | Proteína de membrana | |
| UL44 | Glycoproteína C | P10228 | Q89730 | Superficie y membrana |
| UL45 | UL45 | P10229 | Proteína de membrana; lectina de tipo C | |
| UL46 | VP11/12 | P08314 | Proteínas de tegumento | |
| UL47 | UL47; VP13/14 | P10231 | Proteína de tegumento | |
| UL48 | VP16 (Alpha-TIF) | P04486 | P68336 | maduración de Virion; activa los genes IE interactuando con los factores de transcripción celular Oct-1 y HCF. Ataques a la secuencia 5 'TAATGARAT3 '. |
| UL49 | UL49A | O09800 | Envelope protein | |
| UL50 | UL50 | P10234 | DUTP diphosphatase | |
| UL51 | UL51 | P10234 | Proteína de tegumento | |
| UL52 | UL52 | P10236 | proteína compleja helicasa/primas ADN | |
| UL53 | Glycoprotein K | P68333 | Superficie y membrana | |
| UL54 | IE63; ICP27 | P10238 | Regulación transcripcional e inhibición de la señalización STING | |
| UL55 | UL55 | P10239 | Desconocido | |
| UL56 | UL56 | P10240 | Desconocido | |
| Repetición invertida larga (IRL) | ||||
| Repetición invertida corta (IRS) | ||||
| Unique short (U)S) | ||||
| US1 | ICP22; IE68 | P04485 | Reproducción viral | |
| US2 | US2 | P06485 | Desconocido | |
| US3 | US3 | P04413 | Serine/threonine-protein kinase | |
| US4 | Glycoprotein G | P06484 | P13290 | Superficie y membrana |
| US5 | Glycoprotein J | P06480 | Superficie y membrana | |
| US6 | Glycoproteína D | A1Z0Q5 | Q69467 | Superficie y membrana |
| US7 | Glycoproteína I | P06487 | Superficie y membrana | |
| US8 | Glycoproteína E | Q703F0 | P89475 | Superficie y membrana |
| US9 | US9 | P06481 | Proteína de tegumento | |
| US10 | US10 | P06486 | Proteína capsid/tegument | |
| US11 | US11; Vmw21 | P56958 | ADN y ARN | |
| US12 | ICP47; IE12 | P03170 | Inhibiciones clase MHC Camino previniendo la unión del antígeno al TAP | |
| Terminal de repetición corta (TRS) | ||||
| RS1 | ICP4; IE175 | P08392 | Mayor activador transcripcional. Esencial para la progresión más allá de la fase inicial de infección. Represor de transcripción IEG. | |
Expresión genética
Los genes del HSV se expresan en 3 clases temporales: genes inmediatos tempranos (IE o α), tempranos (E o ß) y tardíos (γ). Sin embargo, la progresión de la expresión de genes virales es más bien gradual que en etapas claramente distintas. Los genes tempranos inmediatos se transcriben inmediatamente después de la infección y sus productos genéticos activan la transcripción de los genes tempranos. Los primeros productos genéticos ayudan a replicar el ADN viral. La replicación del ADN viral, a su vez, estimula la expresión de los genes tardíos que codifican las proteínas estructurales.
La transcripción de los genes tempranos inmediatos (IE) comienza justo después de que el ADN del virus ingresa al núcleo. Todos los genes del virus son transcritos por la ARN polimerasa II del huésped. Aunque las proteínas del huésped son suficientes para la transcripción del virus, las proteínas virales son necesarias para la transcripción de ciertos genes. Por ejemplo, VP16 desempeña un papel importante en la transcripción de IE y la partícula del virus aparentemente lo introduce en la célula huésped, por lo que no es necesario producirlo primero. De manera similar, las proteínas IE RS1 (ICP4), UL54 (ICP27) e ICP0 promueven la transcripción de los genes tempranos (E). Al igual que los genes IE, los promotores de genes tempranos contienen sitios de unión para factores de transcripción celular. Una proteína temprana, ICP8, es necesaria tanto para la transcripción de genes tardíos como para la replicación del ADN.
Más adelante en el ciclo de vida del VHS, se detiene la expresión de genes tempranos e inmediatos. Esto está mediado por proteínas virales específicas, p. ICP4, que se reprime a sí mismo uniéndose a elementos de su propio promotor. Como consecuencia, la regulación negativa de los niveles de ICP4 conduce a una reducción de la expresión genética temprana y tardía, ya que ICP4 es importante para ambos.
Es importante destacar que el VHS desactiva la síntesis de ARN, ADN y proteínas de la célula huésped para dirigir los recursos celulares a la producción de virus. En primer lugar, la proteína del virus vhs induce la degradación de los ARNm existentes en las primeras etapas de la infección. Otros genes virales impiden la transcripción y traducción celular. Por ejemplo, ICP27 inhibe el empalme del ARN, de modo que los ARNm del virus (que normalmente no están empalmados) obtienen una ventaja sobre los ARNm del huésped. Finalmente, las proteínas del virus desestabilizan ciertas proteínas celulares involucradas en el ciclo de la célula huésped, de modo que tanto la división celular como la replicación del ADN de la célula huésped se alteran a favor de la replicación del virus.
Evolución
Los genomas del herpes simple 1 se pueden clasificar en seis clados. Cuatro de ellos ocurren en África Oriental, uno en Asia Oriental y uno en Europa y América del Norte. Esto sugiere que el virus pudo haberse originado en África Oriental. El ancestro común más reciente de las cepas euroasiáticas parece haber evolucionado hace unos 60.000 años. Los aislamientos de HSV-1 del este de Asia tienen un patrón inusual que actualmente se explica mejor por las dos oleadas de migración responsables del poblamiento de Japón.
Los genomas del herpes simple 2 se pueden dividir en dos grupos: uno está distribuido globalmente y el otro se limita principalmente al África subsahariana. El genotipo distribuido globalmente ha experimentado cuatro recombinaciones antiguas con herpes simple 1. También se ha informado que HSV-1 y HSV-2 pueden tener eventos de recombinación contemporáneos y estables en huéspedes infectados simultáneamente con ambos patógenos. En todos los casos, el VHS-2 adquiere partes del genoma del VHS-1, y a veces cambia partes de su epítopo antigénico en el proceso.
Se ha estimado que la tasa de mutación es ~1,38×10−7 sustituciones/sitio/año. En el ámbito clínico, las mutaciones en el gen de la timidina quinasa o en el gen de la ADN polimerasa han causado resistencia al aciclovir. Sin embargo, la mayoría de las mutaciones ocurren en el gen de la timidina quinasa y no en el gen de la ADN polimerasa.
Otro análisis ha estimado que la tasa de mutación en el genoma del herpes simple 1 es de 1,82 × 10−8 sustitución de nucleótidos por sitio por año. Este análisis sitúa al ancestro común más reciente de este virus hace unos 710.000 años.
Los herpes simple 1 y 2 divergieron hace aproximadamente hace 6 millones de años.
Tratamiento
Al igual que otros herpesviridae, los virus del herpes simple establecen una infección latente de por vida y, por lo tanto, no pueden erradicarse del cuerpo con los tratamientos actuales.
El tratamiento generalmente incluye medicamentos antivirales de uso general que interfieren con la replicación viral, reducen la gravedad física de las lesiones asociadas al brote y reducen la posibilidad de transmisión a otras personas. Los estudios de poblaciones de pacientes vulnerables han indicado que el uso diario de antivirales como aciclovir y valaciclovir puede reducir las tasas de reactivación. El uso extensivo de fármacos antiherpéticos ha llevado al desarrollo de cierta resistencia a los medicamentos, lo que a su vez puede conducir al fracaso del tratamiento. Por lo tanto, se investigan ampliamente nuevas fuentes de medicamentos para abordar el problema. En enero de 2020, se publicó un artículo de revisión exhaustivo que demostró la eficacia de los productos naturales como fármacos prometedores contra el VHS. La piritiona, un ionóforo de zinc, ha demostrado actividad antiviral contra el herpes simple.
Enfermedad de Alzheimer
En 1979, se informó que existe un posible vínculo entre el HSV-1 y la enfermedad de Alzheimer, en personas con el alelo épsilon4 del gen APOE. HSV-1 parece ser particularmente dañino para el sistema nervioso y aumenta el riesgo de desarrollar la enfermedad de Alzheimer. El virus interactúa con los componentes y receptores de las lipoproteínas, lo que puede provocar el desarrollo de la enfermedad de Alzheimer. Esta investigación identifica a los HSV como el patógeno más claramente relacionado con el establecimiento del Alzheimer. Según un estudio realizado en 1997, sin la presencia del alelo genético, HSV-1 no parece causar ningún daño neurológico ni aumentar el riesgo de Alzheimer. Sin embargo, un estudio prospectivo más reciente publicado en 2008 con una cohorte de 591 personas mostró una diferencia estadísticamente significativa entre los pacientes con anticuerpos que indicaban una reactivación reciente del VHS y aquellos sin estos anticuerpos en la incidencia de la enfermedad de Alzheimer, sin correlación directa con el alelo APOE-épsilon4.
El ensayo contó con una pequeña muestra de pacientes que no tenían el anticuerpo al inicio del estudio, por lo que los resultados deben considerarse muy inciertos. En 2011, científicos de la Universidad de Manchester demostraron que el tratamiento de células infectadas por HSV1 con agentes antivirales disminuía la acumulación de proteína β-amiloide y tau y también disminuía la replicación de HSV-1.
Un estudio retrospectivo de 2018 realizado en Taiwán sobre 33.000 pacientes encontró que estar infectado con el virus del herpes simple aumentaba el riesgo de demencia 2,56 veces (IC 95%: 2,3-2,8) en pacientes que no recibían medicamentos antiherpéticos (2,6 veces para HSV- 1 infecciones y 2,0 veces para infecciones por HSV-2). Sin embargo, los pacientes infectados por HSV que estaban recibiendo medicamentos antiherpéticos (p. ej., aciclovir, famciclovir, ganciclovir, idoxuridina, penciclovir, tromantadina, valaciclovir o valganciclovir) no mostraron un riesgo elevado de demencia en comparación con los pacientes no infectados por HSV.
reactivación multiplicidad
La reactivación de multiplicidad (MR) es el proceso mediante el cual los genomas virales que contienen daño inactivante interactúan dentro de una célula infectada para formar un genoma viral viable. La RM se descubrió originalmente con el bacteriófago T4 del virus bacteriano, pero posteriormente también se encontró con virus patógenos, incluidos el virus de la influenza, el VIH-1, el virus del adenovirus simio 40, el virus vaccinia, el reovirus, el poliovirus y el virus del herpes simple.
Cuando las partículas de HSV se exponen a dosis de un agente que daña el ADN que sería letal en infecciones únicas, pero luego se les permite sufrir infecciones múltiples (es decir, dos o más virus por célula huésped), se observa RM. La supervivencia mejorada del HSV-1 debido a la RM se produce tras la exposición a diferentes agentes que dañan el ADN, incluido el metanosulfonato de metilo, el trimetilpsoraleno (que provoca enlaces cruzados entre cadenas de ADN) y la luz ultravioleta. Después del tratamiento del HSV genéticamente marcado con trimetilpsoraleno, aumenta la recombinación entre los virus marcados, lo que sugiere que el daño del trimetilpsoraleno estimula la recombinación. La RM del VHS parece depender parcialmente de la maquinaria de reparación recombinante de la célula huésped, ya que las células de fibroblastos de la piel defectuosas en un componente de esta maquinaria (es decir, las células de pacientes con síndrome de Bloom) son deficientes en la RM.
Estas observaciones sugieren que la RM en las infecciones por HSV implica una recombinación genética entre genomas virales dañados que da como resultado la producción de una progenie de virus viables. HSV-1, al infectar las células huésped, induce inflamación y estrés oxidativo. Por lo tanto, parece que el genoma del HSV puede estar sujeto a daño oxidativo del ADN durante la infección y que la RM puede mejorar la supervivencia y la virulencia viral en estas condiciones.
Uso como agente anti-cáncer
El virus del herpes simple modificado se considera una terapia potencial para el cáncer y ha sido ampliamente probado clínicamente para evaluar su capacidad oncolítica (eliminación del cáncer). Los datos provisionales de supervivencia general del ensayo de fase 3 de Amgen de un virus del herpes genéticamente atenuado sugieren eficacia contra el melanoma.
Uso en el rastreo de conexión neuronal
El virus del herpes simple también se utiliza como marcador transneuronal que define conexiones entre neuronas en virtud de atravesar sinapsis.
Otros resultados conexos
HSV-2 es la causa más común de meningitis de Mollaret. El HSV-1 puede provocar casos potencialmente mortales de encefalitis por herpes simple. Los virus del herpes simple también se han estudiado en trastornos del sistema nervioso central como la esclerosis múltiple, pero las investigaciones han sido contradictorias y no concluyentes.
Tras un diagnóstico de infección por herpes simple genital, los pacientes pueden desarrollar un episodio de depresión profunda. Además de ofrecer medicamentos antivirales para aliviar los síntomas y acortar su duración, los médicos también deben abordar el impacto de un nuevo diagnóstico en la salud mental. Proporcionar información sobre la altísima prevalencia de estas infecciones, sus tratamientos efectivos y futuras terapias en desarrollo puede brindar esperanza a pacientes que de otro modo estarían desmoralizados.
Research
Existen vacunas de uso común para algunos herpesvirus, como la vacuna veterinaria HVT/LT (vacuna contra la laringotraqueítis vectorial del herpesvirus de Turquía). Sin embargo, previene la aterosclerosis (que histológicamente refleja la aterosclerosis en humanos) en los animales vacunados. Las únicas vacunas humanas disponibles contra los herpesvirus son las del virus varicela zoster, que se administran a niños alrededor de su primer cumpleaños para prevenir la varicela (varicela), o a adultos para prevenir un brote de culebrilla (herpes zoster). Sin embargo, no existe ninguna vacuna humana contra los virus del herpes simple. A partir de 2022, se están realizando estudios clínicos y preclínicos activos sobre el herpes simple en humanos; Se están desarrollando vacunas tanto para el tratamiento como para la prevención.
Referencias
- ^ a b c Ryan KJ, Ray CG, Eds. (2004). Sherris Medical Microbiology (4a edición). McGraw Hill. pp. 555–62. ISBN 978-0-8385-8529-0.
- ^ Chayavichitsilp P, Buckwalter JV, Krakowski AC, Friedlander SF (abril de 2009). "Herpes simplex". Pediatr Rev. 30 (4): 119–29, quiz 130. doi:10.1542/pir.30-4-119. PMID 19339385. S2CID 34735917.
- ^ "Herpes simplex virus". World Health Organization31 de enero de 2017.
- ^ "Prevalencia de Herpes Simplex Virus Tipo 1 y 2" (PDF). CDC NCHS Reunión de datos16 de febrero de 2020.
- ^ Straface G, Selmin A, Zanardo V, De Santis M, Ercoli A, Scambia G (2012). "Herpes simplex infección de virus en el embarazo". Enfermedades Infecciosas en Obstetricia y Ginecología. 2012: 385697. doi:10.1155/2012/385697. PMC 3332182. PMID 22566740.
- ^ "Herpes simplex virus". World Health Organization31 de enero de 2017. Retrieved 22 de septiembre, 2018.
- ^ Stephenson M (2020-09-09). "Cómo manejar herpes oculares". Examen de la Oftalmología. Retrieved 2021-06-07.
- ^ "Herpes simplex". Derm Net NZ — New Zealand Dermatological Society. 2006-09-16. Retrieved 2006-10-15.
- ^ Johnston C, Magaret A, Son H, Stern M, Rathbun M, Renner D, et al. (Noviembre 2022). "Sembrada Viral 1 año después de la Infección HSV-1 de Primera Episode". JAMA. 328 (17): 1730–1739. doi:10.1001/jama.2022.19061. PMC 9588168. PMID 36272098.
- ^ Schiffer JT, Mayer BT, Fong Y, Swan DA, Wald A (2014). "Herpes simplex virus-2 estimaciones de probabilidad de transmisión basado en la cantidad de cobertura viral". J R Soc Interface. 11 (95): 20140160. doi:10.1098/rsif.2014.0160. PMC 4006256. PMID 24671939.
- ^ a b Johnston C, Koelle DM, Wald A (Dec 2011). "HSV-2: en busca de una vacuna". J Clin Invest. 121 (12): 4600–9. doi:10.1172/JCI57148. 3223069. PMID 22133885.
- ^ Sperling RS, Fife KH, Warren TJ, Dix LP, Brennan CA (marzo de 2008). "El efecto de la supresión diaria de valaciclovir en herpes simple virus tipo 2 derramamiento viral en sujetos seropositivos HSV-2 sin antecedentes de herpes genital". Sex Transm Dis. 35 (3): 286-90. doi:10.1097/OCQ.0b013e31815b0132PMID 18157071. S2CID 20687438.
- ^ Wald A, Langenberg AG, Link K, Izu AE, Ashley R, Warren T, et al. (junio de 2001). "Efecto de los condones en la reducción de la transmisión de herpes simples virus tipo 2 de hombres a mujeres". JAMA. 285 (24): 3100–3106. doi:10.1001/jama.285.24.3100. PMID 11427138.
- ^ a b Gupta R, Warren T, Wald A (diciembre de 2007). "Herpes genitales". Lancet. 370 (9605): 2127–2137. doi:10.1016/S0140-6736(07)61908-4. PMID 18156035. S2CID 40916450.
- ^ a b Koelle DM, Corey L (2008). "Herpes simplex: información sobre patogenesis y posibles vacunas". Examen anual de la medicina. 59: 381–95. doi:10.1146/annurev.med.59.061606.095540. PMID 18186706.
- ^ "Todo lo que necesitas saber sobre los herpes". 2017-12-11.
- ^ Corey L, Wald A (octubre de 2009). "Infecciones de virus del herpes materno y neonatal". El New England Journal of Medicine. 361 (14): 1376–1385. doi:10.1056/NEJMra0807633. PMC 2780322. PMID 19797284.
- ^ Usatine RP, Tinitigan R (noviembre de 2010). "El herpes nongenital simplex virus". American Family Fisician. 82 (9): 1075–1082. PMID 21121552.
- ^ Kimberlin DW (febrero de 2007). "Herpes simplex virus infecciones del recién nacido". Seminarios en Perinatología. 31 (1): 19–25. doi:10.1053/j.semperi.2007.01.003. PMID 17317423.
- ^ "Mijn kind heeft blaasjes in de mond door herpes durable Thuisarts.nl". www.thuisarts.nl 21 de septiembre de 2022. Retrieved 2022-12-18.
- ^ "¿Puedes atrapar a las ETS de un asiento de inodoro?". mylabbox.com. 2019-02-12. Retrieved 16 de julio 2019.
- ^ García-García B, Galache-Osuna C, Coto-Segura P, Suárez-Casado H, Mallo-García S, Jiménez JS (febrero de 2013). "Presentación inusual de la infección por el virus herpes simples en un boxeador: 'Boxing glove herpes'". The Australasian Journal of Dermatology. 54 (1): e22–e24. doi:10.1111/j.1440-0960.2011.00815.x. PMID 23373892. S2CID 11353611.
- ^ Suissa CA, Upadhyay R, Dabney MD, Mack RJ, Masica D, Margulies BJ (Marzo 2023). "Investigando la supervivencia del virus herpes simplex en cepillos de dientes y dispositivos de fálico surrogados". International Journal of STD " AIDS. 34 (3): 152–158. doi:10.1177/09564624221142380. PMID 36448203. S2CID 254095088.
- ^ Regezi JA, Sciubba JJ, Jordan RC, eds. (2012-01-01). "Capítulo 1 - Enfermedades Vesiculobulares". Patología Oral (Sexta edición). St. Louis: W.B. Saunders. pp. 1–21. doi:10.1016/B978-1-4557-0262-6.00001-X. hdl:20.500.12613/9321. ISBN 978-1-4557-0262-6.
- ^ Looker KJ, Elmes JA, Gottlieb SL, Schiffer JT, Vickerman P, Turner KM, et al. (diciembre de 2017). "Efecto de la infección HSV-2 en la adquisición posterior del VIH: revisión sistemática actualizada y metaanálisis". El Lancet. Enfermedades Infecciosas. 17 (12): 1303–1316. doi:10.1016/S1473-3099(17)30405-X. PMC 5700807. PMID 28843576.
- ^ a b c d e f Taylor TJ, Brockman MA, McNamee EE, Knipe DM (marzo de 2002). "Herpes simplex virus". Frontiers in Bioscience. 7 (1–3): d752–d764. doi:10.2741/taylor. PMID 11861220.
- ^ a b Mettenleiter TC, Klupp BG, Granzow H (2006). "Ensamblamiento del virus del herpes: un relato de dos membranas". Curr. Opin. Microbiol. 9 (4): 423-9. doi:10.1016/j.mib.2006.06.013. PMID 16814597.
- ^ a b c d e McGeoch DJ, Rixon FJ, Davison AJ (2006). "Temas en la genómica y evolución del herpesvirus". Virus Res. 117 (1): 90–104. doi:10.1016/j.virusres.2006.01.002. PMID 16490275.
- ^ Rajcáni J, Andrea V, Ingeborg R (2004). "Peculiarities of herpes simplex virus (HSV) transcripción: un resumen". Virus Genes. 28 (3): 293–310. doi:10.1023/B:VIRU.0000025777.62826.92 PMID 15266111. S2CID 19737920.
- ^ Isa NF, Bensaude O, Aziz NC, Murphy S (septiembre 2021). "HSV-1 ICP22 Es un Represor Viral Selectivo de la Elongación de la Transcripción Polimerasa Polimerasa II Mediada". Vacunas. 9 (10): 1054. doi:10.3390/vaccines9101054. PMC 8539892. PMID 34696162.
- ^ a b Clarke RW (2015). "Forces and Structures of the Herpes Simplex Virus (HSV) Entry Mechanism". ACS Enfermedades Infecciosas. 1 (9): 403-415. doi:10.1021/acsinfecdis.5b00059. PMID 27617923.
- ^ a b Subramanian RP, Geraghty RJ (2007). "Herpes simplex virus tipo 1 media la fusión a través de una hemifusión intermedia por actividad secuencial de glicoproteínas D, H, L y B". Proc. Natl. Acad. Sci. USA. 104 (8): 2903-8. Código:2007PNAS..104.2903S. doi:10.1073/pnas.0608374104. PMC 1815279. PMID 17299053.
- ^ Akhtar J, Shukla D (2009). "Mecanismos de entrada viales: Mediadores celulares y virales de la entrada de virus herpes simples". FEBS Journal. 276 (24): 7228–7236. doi:10.1111/j.1742-4658.2009.07402.x. PMC 2801626. PMID 19878306.
- ^ Tunnicliffe RB, Hu WK, Wu MY, Levy C, Mould AP, McKenzie EA, et al. (Octubre 2019). Damania B (ed.). "Molecular Mechanism of SR Protein Kinase 1 Inhibition by the Herpes Virus Protein ICP27". m Bio. 10 (5): e02551–19. doi:10.1128/mBio.02551-19. PMC 6805999. PMID 31641093.
- ^ a b Cardone G, Winkler DC, Trus BL, Cheng N, Heuser JE, Newcomb WW, et al. (Mayo 2007). "Visualization of the Herpes Simplex Virus Portal in situ by Cryo-electron Tomography". Virología. 361 (2): 426–34. doi:10.1016/j.virol.2006.10.047. PMC 1930166. PMID 17188319.
- ^ a b Trus BL, Cheng N, Newcomb WW, Homa FL, Brown JC, Steven AC (noviembre de 2004). "Strutura y polimorfismo del Portal UL6 Proteína de Herpes Simplex Virus Tipo 1". Journal of Virology. 78 (22): 12668–71. doi:10.1128/JVI.78.22.12668-12671.2004. PMC 525097. PMID 15507654.
- ^ a b Nellissery JK, Szczepaniak R, Lamberti C, Weller SK (2007-06-20). "A Putative Leucine Zipper in the Herpes Simplex Virus Tipo 1 UL6 Protein es obligatorio para la formación de anillos de portal". Journal of Virology. 81 (17): 8868–77. doi:10.1128/JVI.00739-07. PMC 1951442. PMID 17581990.
- ^ Newcomb WW, Booy FP, Brown JC (2007). "Uncoating the Herpes Simplex Virus Genome". J. Mol. Biol. 370 (4): 633–42. doi:10.1016/j.jmb.2007.05.023. PMC 1975772. PMID 17540405.
- ^ Berger C, Xuereb S, Johnson DC, Watanabe KS, Kiem HP, Greenberg PD, et al. (mayo de 2000). "Expresión del virus herpes simples ICP47 y el citomegalovirus humano US11 evita el reconocimiento de los productos transgénicos por los linfocitos T citotóxicos CD8(+). Journal of Virology. 74 (10): 4465-73. doi:10.1128/jvi.74.10.4465-4473.2000. PMC 111967. PMID 10775582.
- ^ Roy S, Sukla S, De A, Biswas S (enero 2022). "Hepes no citopáticos virus simplex tipo-1 aislados de pacientes tratados con aciclovir con infecciones recurrentes". Scientific Reports. 12 (1): 1345. Bibcode:2022NatSR..12.1345R. doi:10.1038/s41598-022-05188-w. PMC 8789845. PMID 35079057.
- ^ Adang LA, Parsons CH, Kedes DH (2006). "Progreso sincrónico a través de la Cascada Lítico y Variaciones en Cargas Virales Intracelulares Reveladas por el Análisis de Célula Única de Alto Cresculo de la Infección de Herpesvirus asociado de Kaposi". J. Virol. 80 (20): 10073–82. doi:10.1128/JVI.01156-06. PMC 1617294. PMID 17005685.
- ^ a b Matis J, Kúdelová M (2001). "Desactivación total de la síntesis de proteínas anfitrionas en células infectadas con virus herpes simples". Acta Virol. 45 (5–6): 269–77. doi:10.2217/fvl.11.24. hdl:1808/23396. PMID 12083325.
- ^ Taddeo B, Roizman B (2006). "The Virion Host Shutoff Protein (UL41) of Herpes Simplex Virus 1 Es una Endoribonucleasa con una especificación de sustrato similar a la de RNase A". J. Virol. 80 (18): 9341–5. doi:10.1128/JVI.01008-06. PMC 1563938. PMID 16940547.
- ^ Skepper JN, Whiteley A, Browne H, Minson A (junio de 2001). "Herpes Simplex Virus Nucleocapsids Mature to Progeny Virions by an Envelopment → Deenvelopment → Reenvelopment Pathway". J. Virol. 75 (12): 5697–702. doi:10.1128/JVI.75.12.5697-5702.2001. PMC 114284. PMID 11356979.
- ^ Granzow H, Klupp BG, Fuchs W, Veits J, Osterrieder N, Mettenleiter TC (abril de 2001). "Egress of Alphaherpesviruses: Comparative Ultrastructural Study". J. Virol. 75 (8): 3675–84. doi:10.1128/JVI.75.8.3675-3684.2001. PMC 114859. PMID 11264357.
- ^ Jeffrey I Cohen (4 de mayo de 2020). "Herpesvirus latencia". Journal of Clinical Investigation. doi:10.1172/JCI136225. ISSN 0021-9738. PMC 7324166. PMID 32364538. Wikidata Q94509178.
- ^ a b Pinnoji RC, Bedadala GR, George B, Holland TC, Hill JM, Hsia SC (2007). "El elemento de presión-1 silenciador factor de transcripción/factor de silenciador neuronal restrictivo (REST/NRSF) puede regular la transcripción inmediata de HSV-1 mediante modificación de la piedra angular". Virol. J. 4: 56. doi:10.1186/1743-422X-4-56. PMC 1906746. PMID 17555596.
- ^ Bedadala GR, Pinnoji RC, Hsia SC (2007). "La respuesta al crecimiento temprano gene 1 (Egr-1) regula la expresión de genes HSV-1 ICP4 e ICP22". Cell Res. 17 (6): 546-55. doi:10.1038/cr.2007.44. PMC 7092374. PMID 17502875.
- ^ Roizman B, Gu H, Mandel G (2005). "Los primeros 30 minutos en la vida de un virus: unREST en el núcleo". Ciclo de células. 4 (8): 1019–21. doi:10.4161/cc.4.8.1902. PMID 16082207.
- ^ Davidson AJ (2007-08-16). "Análisis comparativo de los genomas". Herpesvirus humanos. Cambridge University Press. ISBN 978-0-521-82714-0. PMID 21348122.
- ^ Slobedman B, Zhang X, Simmons A (enero de 1999). "Herpes simplex virus genome isomerization: origins of adjacent long segments in concatemeric viral DNA". Journal of Virology. 73 (1): 810–3. doi:10.1128/JVI.73.1.810-813.1999. PMC 103895. PMID 9847394.
- ^ "Buscar en UniProt Knowledgebase (Swiss-Prot y TrEMBL) para: HHV1". exasy.org.
- ^ Smith MC, Boutell C, Davido DJ (2011). "HSV-1 ICP0: allanando el camino para la replicación viral". Future Virology. 6 (4): 421-429. doi:10.2217/fvl.11.24. PMC 3133933. PMID 21765858.
- ^ Vittone V, Diefenbach E, Triffett D, Douglas MW, Cunningham AL, Diefenbach RJ (2005). "Determination of Interactions between Tegument Proteins of Herpes Simplex Virus Tipo 1". J. Virol. 79 (15): 9566–71. doi:10.1128/JVI.79.15.9566-9571.2005. PMC 1181608. PMID 16014918.
- ^ Wyrwicz LS, Ginalski K, Rychlewski L (2007). "HSV-1 UL45 codifica una proteína de lectina de tipo C de carbohidratos". Ciclo de células. 7 (2): 269-71. doi:10.4161/cc.7.2.5324. PMID 18256535.
- ^ Christensen MH, Jensen SB, Miettinen JJ, Luecke S, Prabakaran T, Reinert LS, et al. (Julio 2016). "HSV-1 ICP27 apunta al STING activado por TBK1 para inhibir la expresión IFN del tipo inducida por virus". El Diario EMBO. 35 (13): 1385–99. doi:10.15252/embj.201593458. PMC 4931188. PMID 27234299.
- ^ a b Kolb AW, Ané C, Brandt CR (2013). "Usando la filogenética del genoma HSV-1 para rastrear las migraciones humanas pasadas". PLOS ONE. 8 (10): e76267. Bibcode:2013PLoSO...876267K. doi:10.1371/journal.pone.0076267. PMC 3797750. PMID 24146849.
- ^ a b Bowden R, Sakaoka H, Ward R, Donnelly P (2006). "Patterns of Eurasian HSV-1 molecular diversity and inferences of human migrations". Infect Genet Evol. 6 (1): 63–74. Código:2006InfGE...6...63B. doi:10.1016/j.meegid.2005.01.004. PMID 16376841.
- ^ Burrel S, Boutolleau D, Ryu D, Agut H, Merkel K, Leendertz FH, et al. (Julio de 2017). "Ancient Recombination Events between Human Herpes Simplex Viruses". Biología molecular y evolución. 34 (7): 1713-1721. doi:10.1093/molbev/msx113. PMC 5455963. PMID 28369565.
- ^ a b Casto AM, Roychoudhury P, Xie H, Selke S, Perchetti GA, Wofford H, et al. (Marzo 2020). "Large, Stable, Contemporary Interspecies Recombination Events in Circulating Human Herpes Simplex Viruses". El Diario de las Enfermedades Infecciosas. 221 (8): 1271–1279. biorxiv 10.1101/472639. doi:10.1093/infdis/jiz199. PMC 7325804. PMID 31016321.
- ^ Hussin A, Md Nor NS, Ibrahim N (noviembre de 2013). "Caracterización penotípica y genotípica de aislados clínicos inducidos acyclovir resistentes a herpes simples virus tipo 1". Antiviral Research. 100 (2): 306–13. doi:10.1016/j.antiviral.2013.09.008. PMID 24055837.
- ^ Norberg P, Tyler S, Severini A, Whitley R, Liljeqvist JÅ, Bergström T (2011). "Un análisis evolutivo comparativo a nivel genoma del virus herpes simplex tipo 1 y varicella zoster virus". PLOS ONE. 6 (7): e22527. Bibcode:2011PLoSO...622527N. doi:10.1371/journal.pone.0022527. PMC 3143153. PMID 21799886.
- ^ "Datos STD - Herpes Genital". 2017-12-11. Retrieved 30 de octubre 2018.
- ^ Kimberlin DW, Whitley RJ, Wan W, Powell DA, Storch G, Ahmed A, et al. (2011). "Oral acyclovir suppression and neurodevelopment after neonatal herpes". N. Engl. J. Med. 365 (14): 1284–92. doi:10.1056/NEJMoa1003509. PMC 3250992. PMID 21991950.
- ^ Treml J, Gazdová M, Šmejkal K, Šudomová M, Kubatka P, Hassan ST (enero 2020). "Productos Naturales Productos Químicos Derivados: Barreras de Breaking para Novel Anti-HSV Drug Development". Viruses. 12 (2): 154. doi:10.3390/v12020154. PMC 7077281. PMID 32013134.
- ^ Qiu M, Chen Y, Chu Y, Song S, Yang N, Gao J, et al. (octubre de 2013). "Zinc ionophores pyrithione inhibe la replicación del virus herpes simplex interfiriendo con la función proteasome y la activación NF-κB". Antiviral Research. 100 (1): 44–53. doi:10.1016/j.antiviral.2013.07.001. PMID 23867132.
- ^ Middleton PJ, Petric M, Kozak M, Rewcastle NB, McLachlan DR (mayo de 1980). "Herpes-simplex genoma viral y demencias seniles y preseniles de Alzheimer y Pick". Lancet. 315 (8176): 1038. doi:10.1016/S0140-6736(80)91490-7. PMID 6103379. S2CID 11603071.
- ^ Dobson CB, Itzhaki RF (1999). "Herpes simplex virus tipo 1 y enfermedad de Alzheimer". Neurobiol. El envejecimiento. 20 (4): 457–65. doi:10.1016/S0197-4580(99)00055-X. PMID 10604441. S2CID 23633290.
- ^ Pyles RB (noviembre de 2001). "La asociación del virus herpes simple y la enfermedad de Alzheimer: una síntesis potencial de factores genéticos y ambientales" (PDF). Herpes. 8 (3): 64–8. PMID 11867022.
- ^ Itzhaki RF, Lin WR, Shang D, Wilcock GK, Faragher B, Jamieson GA (enero de 1997). "Herpes simplex virus tipo 1 en cerebro y riesgo de enfermedad de Alzheimer". Lancet. 349 (9047): 241–4. doi:10.1016/S0140-6736(96)10149-5. PMID 9014911. S2CID 23380460.
- ^ Letenneur L, Pérès K, Fleury H, Garrigue I, Barberger-Gateau P, Helmer C, et al. (2008). "Seropositividad a los herpes anticuerpos de virus simples y riesgo de enfermedad de Alzheimer: un estudio de cohortes basado en la población". PLOS ONE. 3 (11): e3637. Bibcode:2008PLoSO...3.3637L. doi:10.1371/journal.pone.0003637. PMC 2572852. PMID 18982063.
- ^ Wozniak MA, Frost AL, Preston CM, Itzhaki RF (2011). "Los antivirales reducen la formación de moléculas clave de enfermedad de Alzheimer en las culturas celulares agudamente infectadas con herpes Simplex Virus Tipo 1". PLOS ONE. 6 (10): e25152. Bibcode:2011PLoSO...625152W. doi:10.1371/journal.pone.0025152. PMC 3189195. PMID 22003387.
- ^ Tzeng NS, Chung CH, Lin FH, Chiang CP, Yeh CB, Huang SY, et al. (Abril 2018). " Medicamentos antihépticos y riesgo reducido de demencia en pacientes con herpes Infecciones de virus simples-un estudio de cohortes nacional, basado en la población en Taiwán". Neuroterapia. 15 (2): 417-429. doi:10.1007/s13311-018-0611-x. PMC 5935641. PMID 29488144.
- ^ Michod RE, Bernstein H, Nedelcu AM (2008). "Valoración positiva del sexo en patógenos microbianos". Infección, genética y evolución. 8 (3): 267–285. Código:2008 InfGE...8..267M. doi:10.1016/j.meegid.2008.01.002. PMID 18295550.
- ^ Das SK (agosto de 1982). "Multiplicity reactivation of alkylating agent damaged herpes simplex virus (tipo I) en células humanas". Mutation Research. 105 (1–2): 15–8. doi:10.1016/0165-7992(82)90201-9. PMID 6289091.
- ^ a b Hall JD, Scherer K (diciembre de 1981). "Reparar el ADN tratado con psoralen por recombinación genética en células humanas infectadas con el virus herpes simple". Cancer Research. 41 (12 Pt 1): 5033-8. PMID 6272987.
- ^ Coppey J, Sala-Trepat M, López B (enero de 1989). "Multiplicity reactivation and mutagenesis of trimethylpsoralen-damaged herpes virus in normal and Fanconi's anaemia cells". Mutagenesis. 4 (1): 67–71. doi:10.1093/mutage/4.1.67. PMID 2541311.
- ^ a b Selsky CA, Henson P, Weichselbaum RR, Little JB (septiembre de 1979). "Reactivación defectuosa del herpesvirus de luz ultravioleta por una cepa fibroblast del síndrome de Bloom". Cancer Research. 39 (9): 3392–6. PMID 225021.
- ^ Valyi-Nagy T, Olson SJ, Valyi-Nagy K, Montine TJ, Dermody TS (diciembre de 2000). "Herpes simplex virus tipo 1 latencia en el sistema nervioso murino se asocia con daño oxidativo a las neuronas". Virología. 278 (2): 309–21. doi:10.1006/viro.2000.0678. PMID 11118355.
- ^ Varghese S, Rabkin SD (1 de diciembre de 2002). "Herpes oncolíticos simples vectores de virus para la viroterapia del cáncer". Cancer Gene Terapia. 9 (12): 967-978. doi:10.1038/sj.cgt.7700537. PMID 12522436.
- ^ "Amgen presenta datos de supervivencia general provisionales del estudio de fase 3 de Talimogene Laherparepvec en pacientes con melanoma metastásico" (liberación de prensa). 18 de noviembre de 2013. Retrieved 30 de octubre 2015.
- ^ Norgren RB, Lehman MN (octubre de 1998). "Herpes simplex virus como trazador transneuronal". Neurociencia y Biobehavioral Reviews. 22 (6): 695–708. doi:10.1016/s0149-7634(98)00008-6. PMID 9809305. S2CID 40884240.
- ^ Harrisons Principles of Internal Medicine, 19th edition. p. 1179. ISBN 9780071802154.
- ^ "Meningitis - Enfermedad Infecciosa y Agentes Antimicrobianos". www.antimicrobe.org. Retrieved 2016-03-14.
- ^ Boukhvalova MS, Mortensen E, Mbaye A, Lopez D, Kastrukoff L, Blanco JC (12 de diciembre de 2019). "Herpes Simplex Virus 1 induce la inflamación cerebral y la desmitación multifocal en el Cotton Rat Sigmodon hispidus". J Virol. 94 (1): e01161-19. doi:10.1128/JVI.01161-19. PMC 6912097. PMID 31597775.
- ^ Esaki M, Noland L, Eddins T, Godoy A, Saeki S, Saitoh S, et al. (junio 2013). "Safety and effectiveness of a turkey herpesvirus vector laryngotracheitis vacuna para las gallinas". Enfermedades aviares. 57 (2): 192-8. doi:10.1637/10383-092412-reg.1. PMID 24689173. S2CID 23804575.
- ^ Shih JC (22 de febrero de 1999). "Estudios animales de aterosclerosis inducida por virus". Role of Herpesvirus in Artherogenesis. CRC Press. p. 25. ISBN 978-90-5702-321-7.
Enlaces externos
- "Herpes genitales". Agencia de Salud Pública del Canadá. 2006-05-29.
- Herpes simple: Host viral protein interactions: Una base de datos de HSV-1 que interactúa las proteínas anfitrionas Archived 2010-08-12 en la máquina Wayback
- Estructuras macromoleculares 3D del virus Herpes simplex archivadas en el EM Data Bank(EMDB)