Simbiogénesis
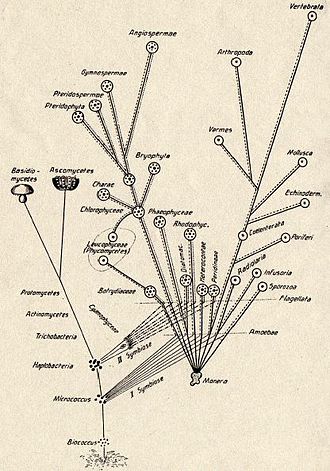
Simbiogénesis (teoría endosimbiótica, o teoría endosimbiótica en serie) es la principal teoría evolutiva del origen de las células eucariotas a partir de organismos procariotas. La teoría sostiene que las mitocondrias, los plástidos como los cloroplastos y posiblemente otros orgánulos de las células eucariotas descienden de procariotas que antes vivían libremente (más estrechamente relacionados con las bacterias que con las arqueas) tomados uno dentro del otro en endosimbiosis. Las mitocondrias parecen estar relacionadas filogenéticamente con las bacterias Rickettsiales, mientras que se cree que los cloroplastos están relacionados con las cianobacterias.
La idea de que los cloroplastos eran originalmente organismos independientes que se fusionaron en una relación simbiótica con otros organismos unicelulares se remonta al siglo XIX, cuando fue defendida por investigadores como Andreas Schimper. La teoría endosimbiótica fue articulada en 1905 y 1910 por el botánico ruso Konstantin Mereschkowski, y desarrollada y fundamentada con evidencia microbiológica por Lynn Margulis en 1967.
Entre las muchas líneas de evidencia que respaldan la simbiogénesis se encuentran que las nuevas mitocondrias y plástidos se forman solo al dividirse en dos, y que las células no pueden crear otras nuevas de otra manera; que las proteínas de transporte denominadas porinas se encuentran en las membranas externas de las mitocondrias, los cloroplastos y las membranas celulares bacterianas; que la cardiolipina se encuentra solo en la membrana mitocondrial interna y en las membranas de las células bacterianas; y que algunas mitocondrias y plástidos contienen moléculas de ADN circulares simples similares a los cromosomas circulares de las bacterias.
Historia
El botánico ruso Konstantin Mereschkowski esbozó por primera vez la teoría de la simbiogénesis (del griego: σύν syn "juntos", βίος bios "vida& #34; y γένεσις génesis "origen, nacimiento") en su obra de 1905, La naturaleza y los orígenes de los cromatóforos en el reino vegetal, y luego lo elaboró en su obra de 1910 La teoría de dos plasmas como base de la simbiogénesis, un nuevo estudio de los orígenes de los organismos. Mereschkowski conocía el trabajo del botánico Andreas Schimper. En 1883, Schimper había observado que la división de los cloroplastos en las plantas verdes se parecía mucho a la de las cianobacterias de vida libre. Había propuesto tentativamente (en una nota a pie de página) que las plantas verdes habían surgido de una unión simbiótica de dos organismos. En 1918, el científico francés Paul Jules Portier publicó Les Symbiotes, en el que afirmaba que las mitocondrias se originaron a partir de un proceso de simbiosis. Ivan Wallin abogó por la idea de un origen endosimbiótico de las mitocondrias en la década de 1920. El botánico ruso Boris Kozo-Polyansky fue el primero en explicar la teoría en términos de evolución darwiniana. En su libro de 1924 Un nuevo principio de biología. Ensayo sobre la teoría de la simbiogénesis, escribió: "La teoría de la simbiogénesis es una teoría de la selección que se basa en el fenómeno de la simbiosis".
Estas teorías no cobraron fuerza hasta que se realizaron comparaciones microscópicas electrónicas más detalladas entre las cianobacterias y los cloroplastos, como las que realizó Hans Ris en 1961 y 1962. Estas, combinadas con el descubrimiento de que los plástidos y las mitocondrias contienen su propio ADN, llevaron a una resurrección de la idea de simbiogénesis en la década de 1960. Lynn Margulis avanzó y justificó la teoría con evidencia microbiológica en un artículo de 1967, Sobre el origen de las células mitosantes. En su trabajo de 1981 Simbiosis en la evolución celular, argumentó que las células eucariotas se originaron como comunidades de entidades que interactúan, incluidas las espiroquetas endosimbióticas que se desarrollaron en flagelos y cilios eucarióticos. Esta última idea no ha recibido mucha aceptación, porque los flagelos carecen de ADN y no muestran similitudes ultraestructurales con bacterias o arqueas (ver también: Evolución de flagelos y citoesqueleto procariótico). Según Margulis y Dorion Sagan, "La vida no se apoderó del mundo mediante el combate, sino mediante la creación de redes" (es decir, por cooperación). Christian de Duve propuso que los peroxisomas pueden haber sido los primeros endosimbiontes, lo que permitió que las células resistieran cantidades crecientes de oxígeno molecular libre en la atmósfera terrestre. Sin embargo, ahora parece que los peroxisomas pueden formarse de novo, contradiciendo la idea de que tienen un origen simbiótico. La teoría fundamental de la simbiogénesis como origen de las mitocondrias y los cloroplastos es ahora ampliamente aceptada.
De endosimbiontes a orgánulos
Los biólogos suelen distinguir los orgánulos de los endosimbiontes (organismos completos que viven dentro de otros organismos) por el tamaño reducido de su genoma. A medida que un endosimbionte se convierte en un orgánulo, la mayoría de sus genes se transfieren al genoma de la célula huésped. Por lo tanto, la célula huésped y el orgánulo necesitan desarrollar un mecanismo de transporte que permita el retorno de los productos proteicos que necesita el orgánulo pero que ahora fabrica la célula.
Antepasados de vida libre
Anteriormente se pensaba que las alfaproteobacterias eran los organismos de vida libre más estrechamente relacionados con las mitocondrias. Investigaciones posteriores indican que las mitocondrias están más estrechamente relacionadas con las bacterias Pelagibacterales, en particular, las del clado SAR11.
Las cianobacterias filamentosas fijadoras de nitrógeno son los organismos de vida libre más estrechamente relacionados con los plástidos.
Tanto las cianobacterias como las alfaproteobacterias mantienen un genoma grande (>6 Mb) que codifica miles de proteínas. Los plástidos y las mitocondrias exhiben una reducción dramática en el tamaño del genoma en comparación con sus parientes bacterianos. Los genomas de los cloroplastos en los organismos fotosintéticos normalmente tienen entre 120 y 200 kb que codifican entre 20 y 200 proteínas y los genomas mitocondriales en los seres humanos tienen aproximadamente 16 kb y codifican 37 genes, 13 de los cuales son proteínas. Sin embargo, utilizando el ejemplo del ameboides de agua dulce, Paulinella chromatophora, que contiene cromatóforos que se descubrió que evolucionaron a partir de cianobacterias, Keeling y Archibald argumentan que este no es el único criterio posible; otra es que la célula huésped ha asumido el control de la regulación de la división del endosimbionte anterior, sincronizándola así con la propia división de la célula. Nowack y sus colegas secuenciaron el gen del cromatóforo (1,02 Mb) y descubrieron que estas células fotosintéticas solo codificaban 867 proteínas. Las comparaciones con sus cianobacterias de vida libre más cercanas del género Synechococcus (con un tamaño de genoma de 3 Mb, con 3300 genes) revelaron que los cromatóforos habían sufrido un cambio drástico contracción. Los cromatóforos contenían genes que eran responsables de la fotosíntesis pero carecían de genes que pudieran llevar a cabo otras funciones biosintéticas; esta observación sugiere que estas células endosimbióticas dependen en gran medida de sus huéspedes para sus mecanismos de supervivencia y crecimiento. Por lo tanto, se encontró que estos cromatóforos no eran funcionales para propósitos específicos de orgánulos en comparación con las mitocondrias y los plástidos. Esta distinción podría haber promovido la evolución temprana de los orgánulos fotosintéticos.
La pérdida de autonomía genética, es decir, la pérdida de muchos genes de los endosimbiontes, ocurrió muy temprano en el tiempo evolutivo. Teniendo en cuenta todo el genoma endosimbionte original, existen tres posibles destinos principales para los genes a lo largo del tiempo evolutivo. El primero es la pérdida de genes funcionalmente redundantes, en los que los genes que ya están representados en el núcleo finalmente se pierden. El segundo es la transferencia de genes al núcleo, mientras que el tercero es que los genes permanecen en el orgánulo que alguna vez fue un organismo. La pérdida de autonomía e integración del endosimbionte con su huésped puede atribuirse principalmente a la transferencia de genes nucleares. Como los genomas de los orgánulos se han reducido considerablemente a lo largo del tiempo evolutivo, los genes nucleares se han expandido y se han vuelto más complejos. Como resultado, muchos procesos de plástidos y mitocondriales están impulsados por productos genéticos codificados en el núcleo. Además, muchos genes nucleares que se originan en endosimbiontes han adquirido funciones novedosas no relacionadas con sus orgánulos.
Mecanismos de transferencia de genes
Los mecanismos de transferencia de genes no se conocen por completo; sin embargo, existen múltiples hipótesis para explicar este fenómeno. Los posibles mecanismos incluyen la hipótesis del ADN complementario (ADNc) y la hipótesis del flujo masivo.
La hipótesis del ADNc implica el uso de ARN mensajero (ARNm) para transportar genes desde los orgánulos hasta el núcleo, donde se convierten en ADNc y se incorporan al genoma. La hipótesis del ADNc se basa en estudios de los genomas de las plantas con flores. Los ARN que codifican proteínas en las mitocondrias se empalman y editan utilizando sitios de edición y empalme específicos de orgánulos. Sin embargo, las copias nucleares de algunos genes mitocondriales no contienen sitios de corte y empalme específicos de orgánulos, lo que sugiere un intermediario de ARNm procesado. Desde entonces, la hipótesis del ADNc se ha revisado, ya que es poco probable que los ADNc mitocondriales editados se recombinen con el genoma nuclear y es más probable que se recombinen con su genoma mitocondrial nativo. Si la secuencia mitocondrial editada se recombina con el genoma mitocondrial, los sitios de empalme mitocondrial ya no existirían en el genoma mitocondrial. Por lo tanto, cualquier transferencia de genes nucleares posterior también carecería de sitios de empalme mitocondrial.
La hipótesis del flujo masivo es la alternativa a la hipótesis del ADNc, y establece que el ADN escapado, en lugar del ARNm, es el mecanismo de transferencia de genes. Según esta hipótesis, las alteraciones de los orgánulos, incluida la autofagia (destrucción celular normal), la gametogénesis (formación de gametos) y el estrés celular, liberan ADN que se importa al núcleo y se incorpora al ADN nuclear mediante la unión de extremos no homólogos (reparación). de roturas de doble cadena). Por ejemplo, en las etapas iniciales de la endosimbiosis, debido a la falta de transferencia de genes importantes, la célula huésped tenía poco o ningún control sobre el endosimbionte. El endosimbionte experimentó división celular independientemente de la célula huésped, lo que resultó en muchas 'copias'. del endosimbionte dentro de la célula huésped. Algunos de los endosimbiontes se lisaron (explotaron) y se incorporaron altos niveles de ADN en el núcleo. Se cree que ocurre un mecanismo similar en las plantas de tabaco, que muestran una alta tasa de transferencia de genes y cuyas células contienen múltiples cloroplastos. Además, la hipótesis del flujo masivo también está respaldada por la presencia de grupos no aleatorios de genes de orgánulos, lo que sugiere el movimiento simultáneo de múltiples genes.
Ford Doolittle propuso que (cualquiera que sea el mecanismo) la transferencia de genes se comporta como un trinquete, lo que resulta en una transferencia unidireccional de genes desde el orgánulo al genoma nuclear. Cuando el material genético de un orgánulo se incorpora al genoma nuclear, la población puede perder el orgánulo o la copia nuclear del gen. Si la copia del orgánulo se pierde y se arregla, o se pierde por deriva genética, un gen se transfiere con éxito al núcleo. Si se pierde la copia nuclear, la transferencia horizontal de genes puede ocurrir nuevamente y la célula puede "intentar nuevamente" para tener una transferencia exitosa de genes al núcleo. De esta manera similar a un trinquete, se esperaría que los genes de un orgánulo se acumularan en el genoma nuclear a lo largo del tiempo evolutivo.
Endosimbiosis de protomitocondrias
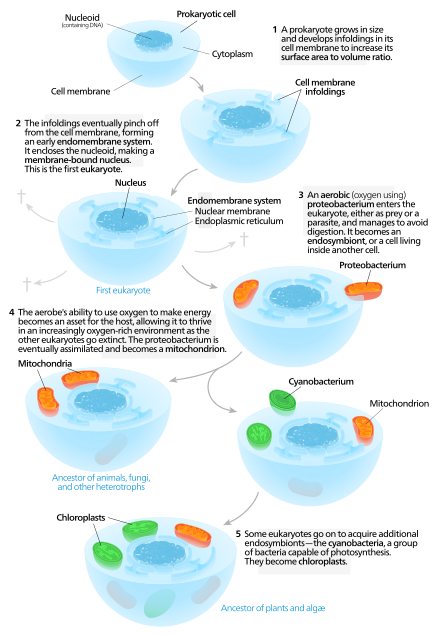
La teoría endosimbiótica para el origen de las mitocondrias sugiere que el protoeucariota engulló una protomitocondria, y este endosimbionte se convirtió en un orgánulo.
Mitocondrias
Las mitocondrias son orgánulos que sintetizan la molécula ATP que transporta energía para la célula al metabolizar macromoléculas a base de carbono. La presencia de ADN en las mitocondrias y proteínas derivadas del mtDNA sugiere que este orgánulo pudo haber sido un procariota antes de su integración en el protoeucariota. Las mitocondrias se consideran orgánulos en lugar de endosimbiontes porque las mitocondrias y las células huésped comparten algunas partes de su genoma, experimentan mitosis simultáneamente y se proporcionan mutuamente los medios para producir energía. Se planteó la hipótesis de que el sistema de endomembranas y la membrana nuclear se derivaron de las protomitocondrias.
Membrana nuclear
La presencia de un núcleo es una de las principales diferencias entre eucariotas y procariotas. Algunas proteínas nucleares conservadas entre eucariotas y procariotas sugieren que estos dos tipos tenían un ancestro común. Otra teoría detrás de la nucleación es que las primeras proteínas de la membrana nuclear hicieron que la membrana celular se doblara y formara una esfera con poros como la envoltura nuclear. Como una forma de formar una membrana nuclear, se podría esperar que la endosimbiosis use menos energía que si la célula desarrollara un proceso metabólico para plegar la membrana celular con ese propósito. La digestión de células engullidas sin mitocondrias productoras de energía habría sido un desafío para la célula huésped. En esta vista, las burbujas o vesículas unidas a la membrana que salen de las protomitocondrias pueden haber formado la envoltura nuclear.
El proceso de simbiogénesis mediante el cual la célula eucariota primitiva integró la protomitocondria probablemente incluía la protección del genoma huésped de la arquea frente a la liberación de especies reactivas de oxígeno. Estos se habrían formado durante la fosforilación oxidativa y la producción de ATP por parte de la proto-mitocondria. La membrana nuclear puede haber evolucionado como una innovación adaptativa para proteger contra el daño del ADN del genoma nuclear causado por especies reactivas de oxígeno. La transferencia sustancial de genes del genoma proto-mitocondrial ancestral al genoma nuclear probablemente ocurrió durante la evolución eucariótica temprana. La mayor protección del genoma nuclear contra las especies reactivas de oxígeno proporcionada por la membrana nuclear puede explicar el beneficio adaptativo de esta transferencia de genes.
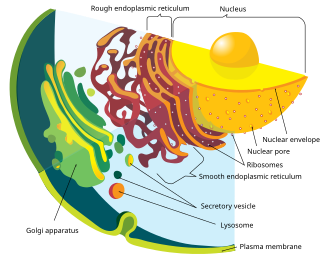
Sistema de endomembranas
Las células eucariotas modernas utilizan el sistema de endomembranas para transportar productos y desechos dentro, dentro y fuera de las células. La membrana de la envoltura nuclear y las vesículas endomembranosas están compuestas por proteínas de membrana similares. Estas vesículas también comparten proteínas de membrana similares con el orgánulo del que se originaron o hacia el que viajan. Esto sugiere que lo que formó la membrana nuclear también formó el sistema de endomembranas. Los procariotas no tienen una red de membrana interna compleja como los eucariotas, pero podrían producir vesículas extracelulares a partir de su membrana externa. Después de que el procariota primitivo fuera consumido por un protoeucariota, el procariota habría continuado produciendo vesículas que se acumularon dentro de la célula. La interacción de los componentes internos de las vesículas puede haber dado lugar al retículo endoplásmico y al aparato de Golgi, siendo ambos partes del sistema de endomembranas.
Genomas organulares
Plastomas y mitogenomas
Algunos genes endosimbiontes permanecen en los orgánulos. Los plástidos y las mitocondrias retienen genes que codifican rRNA, tRNA, proteínas involucradas en reacciones redox y proteínas requeridas para la transcripción, traducción y replicación. Hay muchas hipótesis para explicar por qué los orgánulos conservan una pequeña porción de su genoma; sin embargo, ninguna hipótesis se aplicará a todos los organismos y el tema sigue siendo bastante controvertido. La hipótesis de la hidrofobicidad establece que las proteínas altamente hidrofóbicas (que odian el agua) (como las proteínas unidas a la membrana involucradas en las reacciones redox) no se transportan fácilmente a través del citosol y, por lo tanto, estas proteínas deben codificarse en sus respectivos orgánulos. La hipótesis de la disparidad del código establece que el límite de transferencia se debe a los diferentes códigos genéticos y la edición del ARN entre el orgánulo y el núcleo. La hipótesis del control redox establece que los genes que codifican las proteínas de reacción redox se conservan para acoplar eficazmente la necesidad de reparación y la síntesis de estas proteínas. Por ejemplo, si uno de los fotosistemas se pierde del plástido, los portadores de electrones intermedios pueden perder o ganar demasiados electrones, lo que indica la necesidad de reparar un fotosistema. El retraso de tiempo involucrado en la señalización del núcleo y el transporte de una proteína citosólica al orgánulo da como resultado la producción de especies reactivas de oxígeno dañinas. La hipótesis final establece que el ensamblaje de las proteínas de membrana, en particular las involucradas en las reacciones redox, requiere la síntesis coordinada y el ensamblaje de subunidades; sin embargo, la coordinación de la traducción y el transporte de proteínas es más difícil de controlar en el citoplasma.
Genomas de plástidos no fotosintéticos
La mayoría de los genes en las mitocondrias y los plástidos están relacionados con la expresión (transcripción, traducción y replicación) de genes que codifican proteínas involucradas en la fotosíntesis (en los plástidos) o en la respiración celular (en las mitocondrias). Se podría predecir que la pérdida de la fotosíntesis o de la respiración celular permitiría la pérdida completa del genoma plástido o del genoma mitocondrial, respectivamente. Si bien existen numerosos ejemplos de descendientes mitocondriales (mitosomas e hidrogenosomas) que han perdido todo su genoma organelar, los plástidos no fotosintéticos tienden a retener un genoma pequeño. Hay dos hipótesis principales para explicar este hecho:
La hipótesis del ARNt esencial señala que no se han documentado transferencias funcionales de genes de plástidos a núcleos de genes que codifican productos de ARN (ARNt y ARNr). Como resultado, los plástidos deben producir sus propios ARN funcionales o importar contrapartes nucleares. Sin embargo, los genes que codifican tRNA-Glu y tRNA-fmet parecen ser indispensables. El plástido es responsable de la biosíntesis del hemo, que requiere tRNA-Glu codificado por plástido (del gen trnE) como molécula precursora. Al igual que otros genes que codifican ARN, trnE no se puede transferir al núcleo. Además, es poco probable que trnE pueda ser reemplazado por un tRNA-Glu citosólico ya que trnE está altamente conservado; los cambios de una sola base en trnE han resultado en la pérdida de la síntesis de hemo. El gen de la tRNA-formilmetionina (tRNA-fmet) también está codificado en el genoma del plástido y es necesario para el inicio de la traducción tanto en los plástidos como en las mitocondrias. Se requiere un plástido para continuar expresando el gen para tRNA-fmet mientras la mitocondria esté traduciendo proteínas.
La hipótesis de la ventana limitada ofrece una explicación más general de la retención de genes en plástidos no fotosintéticos. Según esta hipótesis, los genes se transfieren al núcleo tras la alteración de los orgánulos. La perturbación era común en las primeras etapas de la endosimbiosis, sin embargo, una vez que la célula huésped obtuvo el control de la división de los orgánulos, los eucariotas pudieron evolucionar para tener solo un plástido por célula. Tener solo un plástido limita severamente la transferencia de genes ya que la lisis del único plástido probablemente daría como resultado la muerte celular. De acuerdo con esta hipótesis, los organismos con plástidos múltiples muestran un aumento de 80 veces en la transferencia de genes de plástido a núcleo en comparación con organismos con plástidos individuales.
Evidencia
Hay muchas líneas de evidencia de que las mitocondrias y los plástidos, incluidos los cloroplastos, surgieron de bacterias.
- Nueva mitocondria y plastoides se forman sólo a través de la fisión binaria, la forma de división celular utilizada por bacterias y arqueas.
- Si se eliminan las mitocondrias o los cloroplastos de una célula, la célula no tiene los medios para crear nuevos. En algunas algas, como Euglena, los plastoides pueden ser destruidos por ciertos químicos o ausencia prolongada de luz sin afectar de otra manera a la célula: los plastoides no regeneran.
- Las proteínas de transporte llamadas porinas se encuentran en las membranas exteriores de mitocondria y cloroplastos y también se encuentran en las membranas celulares bacterianas.
- Una cardiolipina lipídica de membrana se encuentra exclusivamente en la membrana mitocondrial interna y las membranas celulares bacterianas.
- Algunas mitocondrias y algunos plastoides contienen moléculas de ADN circular únicas que son similares al ADN de bacterias tanto en tamaño como estructura.
- Comparaciones de genomas sugieren una estrecha relación entre la mitocondria y las bacterias Rickettsiales.
- Comparaciones de genomas sugieren una estrecha relación entre plastoides y cianobacteria.
- Muchos genes en los genomas de mitocondria y cloroplastos se han perdido o transferido al núcleo de la célula anfitriona. En consecuencia, los cromosomas de muchos eucariotas contienen genes originados por los genomas de mitocondria y plastoides.
- La mitocondria y los plastoides contienen sus propios ribosomas; estos son más similares a los de las bacterias (70S) que los de los eucariotas.
- Las proteínas creadas por mitocondria y cloroplastos utilizan N-formilmetionina como el aminoácido iniciador, como proteínas creadas por bacterias pero no proteínas creadas por genes nucleares eucariotas o arqueas.
Endosimbiosis secundaria
La endosimbiosis primaria implica la absorción de una célula por otro organismo de vida libre. La endosimbiosis secundaria ocurre cuando el producto de la endosimbiosis primaria es engullido y retenido por otro eucariota de vida libre. La endosimbiosis secundaria ha ocurrido varias veces y ha dado lugar a grupos extremadamente diversos de algas y otros eucariotas. Algunos organismos pueden aprovechar oportunistamente un proceso similar, en el que engullen un alga y utilizan los productos de su fotosíntesis, pero una vez que la presa muere (o se pierde), el huésped vuelve a un estado de vida libre. Los endosimbiontes secundarios obligados se vuelven dependientes de sus orgánulos y no pueden sobrevivir en su ausencia. Un evento de endosimbiosis secundaria que involucró un alga roja ancestral y un eucariota heterótrofo resultó en la evolución y diversificación de varios otros linajes fotosintéticos, incluidos Cryptophyta, Haptophyta, Stramenopiles (o Heterokontophyta) y Alveolata.
Se ha observado una posible endosimbiosis secundaria en proceso en el protista heterótrofo Hatena. Este organismo se comporta como un depredador hasta que ingiere un alga verde, que pierde sus flagelos y citoesqueleto pero sigue viviendo como simbionte. Mientras tanto, Hatena, ahora un anfitrión, cambia a la nutrición fotosintética, adquiere la capacidad de moverse hacia la luz y pierde su aparato de alimentación.
A pesar de la diversidad de organismos que contienen plástidos, la morfología, la bioquímica, la organización genómica y la filogenia molecular de los ARN y las proteínas de los plástidos sugieren un origen único de todos los plástidos existentes, aunque esta teoría aún se debate.
Algunas especies, incluido Pediculus humanus (piojos), tienen múltiples cromosomas en la mitocondria. Esto y la filogenética de los genes codificados dentro de la mitocondria sugieren que las mitocondrias tienen múltiples ancestros, que estos fueron adquiridos por endosimbiosis en varias ocasiones en lugar de solo una, y que ha habido amplias fusiones y reordenamientos de genes en los varios cromosomas mitocondriales originales.
Fecha
La cuestión de cuándo se produjo la transición de la forma procariota a la eucariota y cuándo aparecieron en la Tierra los primeros eucariotas del grupo de la corona sigue sin resolverse. Los fósiles corporales más antiguos conocidos que pueden asignarse positivamente a los eucariotas son los acritarcos acantomórficos de la Formación Deonar de 1.631 Gya de la India. Estos fósiles aún pueden identificarse como eucariotas posnucleares derivados con un citoesqueleto sofisticado generador de morfología sostenido por mitocondrias. Esta evidencia fósil indica que la adquisición endosimbiótica de alfaproteobacteria debe haber ocurrido antes de 1,6 Gya. Los relojes moleculares también se han utilizado para estimar el último ancestro común eucariótico, sin embargo, estos métodos tienen una gran incertidumbre inherente y dan una amplia gama de fechas. Los resultados razonables incluyen la estimación de c. 1,8 Gya. Una estimación de 2,3 Gya también parece razonable, y tiene el atractivo adicional de coincidir con una de las perturbaciones biogeoquímicas más pronunciadas en la historia de la Tierra, el Gran Evento de Oxigenación Paleoproterozoico temprano. Se ha sugerido que el marcado aumento en las concentraciones de oxígeno atmosférico en ese momento es una causa que contribuye a la eucariogénesis, lo que induce la evolución de mitocondrias desintoxicantes de oxígeno. Alternativamente, el Gran Evento de Oxidación podría ser una consecuencia de la eucariogénesis y su impacto en la exportación y entierro de carbono orgánico.
Contenido relacionado
Lista de áreas protegidas de Sudáfrica
Christiaan Hendrik Persoon
Pudú