Secuencia conservada

Las secuencias son los aminoácidos para residuos 120-180 de las proteínas. Residuos que se conservan en todas las secuencias se destacan en gris. Debajo de cada sitio (es decir, posición) de la secuencia de proteínas alineación es una clave que denota sitios conservados (*), sitios con reemplazos conservadores (:), sitios con reemplazos semiconservadores (.), y sitios con reemplazos no conservativos (.).
En biología evolutiva, las secuencias conservadas son secuencias idénticas o similares en ácidos nucleicos (ADN y ARN) o proteínas entre especies (secuencias ortólogas), o dentro de un genoma (secuencias parálogas), o entre donantes. y taxones de receptores (secuencias xenólogas). La conservación indica que una secuencia se ha mantenido por selección natural.
Una secuencia altamente conservada es aquella que se ha mantenido relativamente sin cambios en etapas muy anteriores del árbol filogenético y, por lo tanto, en tiempos geológicos muy lejanos. Ejemplos de secuencias altamente conservadas incluyen los componentes de ARN de los ribosomas presentes en todos los ámbitos de la vida, las secuencias homeobox muy extendidas entre los eucariotas y el ARNtm en las bacterias. El estudio de la conservación de secuencias se superpone con los campos de la genómica, la proteómica, la biología evolutiva, la filogenética, la bioinformática y las matemáticas.
Historia
El descubrimiento del papel del ADN en la herencia y las observaciones realizadas por Frederick Sanger sobre la variación entre las insulinas animales en 1949 impulsaron a los primeros biólogos moleculares a estudiar la taxonomía desde una perspectiva molecular. Los estudios de la década de 1960 utilizaron técnicas de hibridación de ADN y reactividad cruzada de proteínas para medir la similitud entre proteínas ortólogas conocidas, como la hemoglobina y el citocromo c. En 1965, Émile Zuckerkandl y Linus Pauling introdujeron el concepto de reloj molecular y propusieron que se podrían utilizar tasas constantes de reemplazo de aminoácidos para estimar el tiempo transcurrido desde que dos organismos divergieron. Si bien las filogenias iniciales coincidían estrechamente con el registro fósil, las observaciones de que algunos genes parecían evolucionar a ritmos diferentes llevaron al desarrollo de teorías de la evolución molecular. La comparación de secuencias de ferredoxina realizada por Margaret Dayhoff en 1966 demostró que la selección natural actuaría para conservar y optimizar secuencias de proteínas esenciales para la vida.
Mecanismos
A lo largo de muchas generaciones, las secuencias de ácido nucleico en el genoma de un linaje evolutivo pueden cambiar gradualmente con el tiempo debido a mutaciones y deleciones aleatorias. Las secuencias también pueden recombinarse o eliminarse debido a reordenamientos cromosómicos. Las secuencias conservadas son secuencias que persisten en el genoma a pesar de tales fuerzas y tienen tasas de mutación más lentas que la tasa de mutación de fondo.
La conservación puede ocurrir en secuencias de ácidos nucleicos codificantes y no codificantes. Se cree que las secuencias de ADN altamente conservadas tienen valor funcional, aunque no se comprende bien el papel de muchas secuencias de ADN no codificantes altamente conservadas. El grado de conservación de una secuencia puede verse afectado por distintas presiones de selección, su resistencia a la mutación, el tamaño de la población y la deriva genética. Muchas secuencias funcionales también son modulares y contienen regiones que pueden estar sujetas a presiones de selección independientes, como los dominios proteicos.
Secuencia de codificación
En las secuencias codificantes, la secuencia de ácidos nucleicos y aminoácidos puede conservarse en diferentes grados, ya que la degeneración del código genético significa que las mutaciones sinónimas en una secuencia codificante no afectan la secuencia de aminoácidos de su producto proteico.
Las secuencias de aminoácidos se pueden conservar para mantener la estructura o función de una proteína o dominio. Las proteínas conservadas sufren menos reemplazos de aminoácidos o es más probable que sustituyan aminoácidos con propiedades bioquímicas similares. Dentro de una secuencia, los aminoácidos que son importantes para el plegamiento, la estabilidad estructural o que forman un sitio de unión pueden estar más conservados.
La secuencia de ácido nucleico de un gen codificante de proteína también puede conservarse mediante otras presiones selectivas. El sesgo en el uso de codones en algunos organismos puede restringir los tipos de mutaciones sinónimas en una secuencia. Las secuencias de ácido nucleico que causan una estructura secundaria en el ARNm de un gen codificante pueden seleccionarse, ya que algunas estructuras pueden afectar negativamente a la traducción, o conservarse cuando el ARNm también actúa como un ARN no codificante funcional.
Sin codificación
Las secuencias no codificantes importantes para la regulación genética, como los sitios de unión o reconocimiento de los ribosomas y los factores de transcripción, pueden conservarse dentro de un genoma. Por ejemplo, también puede conservarse el promotor de un gen u operón conservado. Al igual que ocurre con las proteínas, también se pueden conservar los ácidos nucleicos que son importantes para la estructura y función del ARN no codificante (ARNnc). Sin embargo, la conservación de la secuencia en los ncRNA es generalmente deficiente en comparación con las secuencias que codifican proteínas, y en su lugar a menudo se conservan los pares de bases que contribuyen a la estructura o función.
Identificación
Las secuencias conservadas normalmente se identifican mediante enfoques bioinformáticos basados en la alineación de secuencias. Los avances en la secuenciación de ADN de alto rendimiento y la espectrometría de masas de proteínas han aumentado sustancialmente la disponibilidad de secuencias de proteínas y genomas completos para comparar desde principios de la década de 2000.
Búsqueda de homología
Las secuencias conservadas se pueden identificar mediante búsqueda de homología, utilizando herramientas como BLAST, HMMER, OrthologR e Infernal. Las herramientas de búsqueda de homología pueden tomar una secuencia de proteína o ácido nucleico individual como entrada, o utilizar modelos estadísticos generados a partir de múltiples alineamientos de secuencias relacionadas conocidas. Los modelos estadísticos, como los HMM de perfil y los modelos de covarianza de ARN que también incorporan información estructural, pueden resultar útiles a la hora de buscar secuencias relacionadas más lejanamente. Luego, las secuencias de entrada se alinean con una base de datos de secuencias de individuos u otras especies relacionadas. Luego, los alineamientos resultantes se califican en función del número de aminoácidos o bases coincidentes y del número de espacios o deleciones generados por el alineamiento. Se pueden identificar sustituciones conservadoras aceptables utilizando matrices de sustitución como PAM y BLOSUM. Se supone que las alineaciones con puntuaciones altas provienen de secuencias homólogas. La conservación de una secuencia puede entonces inferirse mediante la detección de homólogos muy similares en un amplio rango filogenético.
Alineación de secuencias múltiples
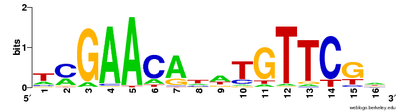
Se pueden utilizar múltiples alineamientos de secuencias para visualizar secuencias conservadas. El formato CLUSTAL incluye una clave de texto sin formato para anotar columnas conservadas de la alineación, que denota secuencia conservada (*), mutaciones conservadoras (:), mutaciones semiconservadoras (.) y mutaciones no conservativas (). Los logotipos de secuencia también pueden mostrar secuencia conservada representando las proporciones de caracteres en cada punto de la alineación por altura.
Alineación del genoma

Las alineaciones del genoma completo (WGA) también se pueden utilizar para identificar regiones altamente conservadas entre especies. Actualmente, la precisión y escalabilidad de las herramientas WGA siguen siendo limitadas debido a la complejidad computacional de lidiar con reordenamientos, regiones repetidas y el gran tamaño de muchos genomas eucariotas. Sin embargo, los WGA de 30 o más bacterias estrechamente relacionadas (procariotas) son cada vez más factibles.
Sistemas de puntuación
Otros enfoques utilizan mediciones de conservación basadas en pruebas estadísticas que intentan identificar secuencias que mutan de manera diferente a una tasa de mutación de fondo (neutral) esperada.
El marco GERP (Genomic Evolutionary Rate Profiling) puntúa la conservación de secuencias genéticas entre especies. Este enfoque estima la tasa de mutación neutra en un conjunto de especies a partir de un alineamiento de secuencias múltiples y luego identifica regiones de la secuencia que exhiben menos mutaciones de las esperadas. Luego, a estas regiones se les asignan puntuaciones basadas en la diferencia entre la tasa de mutación observada y la tasa de mutación de fondo esperada. Una puntuación GERP alta indica una secuencia altamente conservada.
LISTA (Identidad local y taxones compartidos) se basa en el supuesto de que las variaciones observadas en especies estrechamente relacionadas con los humanos son más significativas al evaluar la conservación en comparación con las de especies lejanamente relacionadas. Por lo tanto, LIST utiliza la identidad de alineación local alrededor de cada posición para identificar secuencias relevantes en la alineación de secuencias múltiples (MSA) y luego estima la conservación en función de las distancias taxonómicas de estas secuencias a los humanos. A diferencia de otras herramientas, LIST ignora el recuento/frecuencia de variaciones en el MSA.
Aminode combina múltiples alineamientos con análisis filogenético para analizar cambios en proteínas homólogas y producir un gráfico que indique las tasas locales de cambios evolutivos. Este enfoque identifica las regiones evolutivamente restringidas en una proteína, que son segmentos que están sujetos a una selección purificadora y que suelen ser críticos para la función normal de la proteína.
Otros enfoques, como PhyloP y PhyloHMM, incorporan métodos filogenéticos estadísticos para comparar distribuciones de probabilidad de tasas de sustitución, lo que permite la detección tanto de conservación como de mutación acelerada. Primero, se genera una distribución de probabilidad de fondo del número de sustituciones que se espera que ocurran para una columna en una alineación de secuencias múltiples, basada en un árbol filogenético. Las relaciones evolutivas estimadas entre las especies de interés se utilizan para calcular la importancia de cualquier sustitución (es decir, una sustitución entre dos especies estrechamente relacionadas puede ser menos probable que ocurra que entre especies lejanamente relacionadas y, por lo tanto, más significativa). Para detectar la conservación, se calcula una distribución de probabilidad para un subconjunto del alineamiento de secuencias múltiples y se compara con la distribución de fondo mediante una prueba estadística como una prueba de índice de verosimilitud o una prueba de puntuación. Los valores P generados al comparar las dos distribuciones se utilizan luego para identificar regiones conservadas. PhyloHMM utiliza modelos ocultos de Markov para generar distribuciones de probabilidad. El paquete de software PhyloP compara distribuciones de probabilidad utilizando una prueba de índice de verosimilitud o una prueba de puntuación, además de utilizar un sistema de puntuación similar a GERP.
Conservación extrema
Elementos ultraconservados
Los elementos ultraconservados o UCE son secuencias que son muy similares o idénticas en múltiples grupos taxonómicos. Estos se descubrieron por primera vez en vertebrados y posteriormente se identificaron dentro de taxones muy diferentes. Si bien el origen y la función de las UCE no se conocen bien, se han utilizado para investigar divergencias profundas en amniotas, insectos y entre animales y plantas.
Genes universalmente conservados
Los genes mejor conservados son aquellos que se pueden encontrar en todos los organismos. Estos consisten principalmente en los ncRNA y las proteínas necesarias para la transcripción y la traducción, que se supone que se han conservado del último ancestro común universal de toda la vida.
Los genes o familias de genes que se han encontrado universalmente conservados incluyen factores de elongación de unión a GTP, metionina aminopeptidasa 2, serina hidroximetiltransferasa y transportadores de ATP. Los componentes de la maquinaria de transcripción, como la ARN polimerasa y las helicasas, y de la maquinaria de traducción, como los ARN ribosómicos, los ARNt y las proteínas ribosómicas, también se conservan universalmente.
Aplicaciones
Filogenética y taxonomía
Con frecuencia se utilizan conjuntos de secuencias conservadas para generar árboles filogenéticos, ya que se puede suponer que los organismos con secuencias similares están estrechamente relacionados. La elección de secuencias puede variar según el alcance taxonómico del estudio. Por ejemplo, los genes más conservados, como el ARN 16S y otras secuencias ribosómicas, son útiles para reconstruir relaciones filogenéticas profundas e identificar filos bacterianos en estudios de metagenómica. Las secuencias que se conservan dentro de un clado pero que sufren algunas mutaciones, como los genes constitutivos, se pueden utilizar para estudiar las relaciones entre especies. La región espaciadora transcrita interna (ITS), que es necesaria para espaciar los genes de ARNr conservados pero que experimenta una rápida evolución, se usa comúnmente para clasificar hongos y cepas de bacterias que evolucionan rápidamente.
Investigación médica
Dado que las secuencias altamente conservadas a menudo tienen funciones biológicas importantes, pueden ser un punto de partida útil para identificar la causa de enfermedades genéticas. Muchos trastornos metabólicos congénitos y enfermedades por almacenamiento lisosomal son el resultado de cambios en genes conservados individuales, lo que da como resultado enzimas faltantes o defectuosas que son la causa subyacente de los síntomas de la enfermedad. Las enfermedades genéticas se pueden predecir identificando secuencias conservadas entre humanos y organismos de laboratorio, como ratones o moscas de la fruta, y estudiando los efectos de la desactivación de estos genes. Los estudios de asociación de todo el genoma también se pueden utilizar para identificar variaciones en secuencias conservadas asociadas con enfermedades o resultados de salud. Se han descubierto más de dos docenas de nuevos loci potenciales de susceptibilidad a la enfermedad de Alzheimer.
Anotación funcional
La identificación de secuencias conservadas se puede utilizar para descubrir y predecir secuencias funcionales como los genes. Las secuencias conservadas con una función conocida, como los dominios proteicos, también se pueden utilizar para predecir la función de una secuencia. Las bases de datos de dominios de proteínas conservados, como Pfam y la base de datos de dominios conservados, se pueden utilizar para anotar dominios funcionales en genes codificantes de proteínas predichos.