Ribonucleasa H
Ribonucleasa H (abreviada RNasa H o RNH) es una familia de enzimas endonucleasas no específicas de secuencia que catalizan la escisión del ARN. en un sustrato de ARN/ADN mediante un mecanismo hidrolítico. Los miembros de la familia RNase H se pueden encontrar en casi todos los organismos, desde bacterias hasta arqueas y eucariotas.
La familia se divide en grupos evolutivamente relacionados con preferencias de sustrato ligeramente diferentes, denominados en términos generales ribonucleasa H1 y H2. El genoma humano codifica tanto H1 como H2. La ribonucleasa H2 humana es un complejo heterotrimérico compuesto por tres subunidades, cuyas mutaciones se encuentran entre las causas genéticas de una enfermedad rara conocida como síndrome de Aicardi-Goutières. Un tercer tipo, estrechamente relacionado con el H2, se encuentra sólo en unos pocos procariotas, mientras que el H1 y el H2 se encuentran en todos los ámbitos de la vida. Además, los dominios de ribonucleasa retroviral H de tipo RNasa H1 se encuentran en proteínas transcriptasa inversa multidominio, que están codificadas por retrovirus como el VIH y son necesarias para la replicación viral.
En eucariotas, la ribonucleasa H1 participa en la replicación del ADN del genoma mitocondrial. Tanto H1 como H2 participan en tareas de mantenimiento del genoma, como el procesamiento de estructuras de bucle R.
Clasificación y nomenclatura
La ribonucleasa H es una familia de enzimas endonucleasas con una especificidad de sustrato compartida por la cadena de ARN de los dúplex de ARN-ADN. Por definición, las RNasas H escinden los enlaces fosfodiéster del esqueleto del ARN para dejar una estructura de 3' hidroxilo y un 5' grupo fosfato. Las RNasas H se han propuesto como miembros de una superfamilia evolutivamente relacionada que abarca otras nucleasas y enzimas procesadoras de ácidos nucleicos, como integrasas retrovirales, transposasas de ADN, resolvasas de unión Holliday, proteínas Piwi y Argonaute, varias exonucleasas y la proteína empalmeosomal Prp8.
Las RNasas H se pueden dividir en términos generales en dos subtipos, H1 y H2, que, por razones históricas, reciben designaciones con números arábigos en los eucariotas y designaciones con números romanos en los procariotas. Así, la RNasa HI de Escherichia coli es un homólogo de la RNasa H1 del Homo sapiens. En E. coli y muchos otros procariotas, el gen rnhA codifica HI y el gen rnhB codifica HII. Una tercera clase relacionada, denominada HIII, se presenta en unas pocas bacterias y arqueas; está estrechamente relacionado con las enzimas HII procarióticas.
Estructura
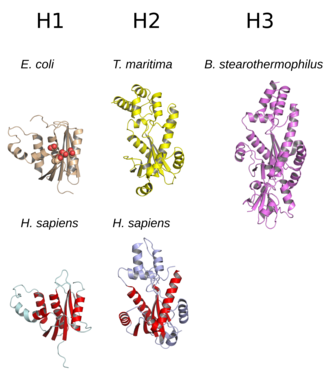
La estructura de la RNasa H comúnmente consiste en una hoja β de 5 cadenas rodeada por una distribución de hélices α. Todas las RNasas H tienen un sitio activo centrado en un motivo de secuencia conservada compuesto de residuos de aspartato y glutamato, a menudo denominado motivo DEDD. Estos residuos interactúan con iones de magnesio necesarios catalíticamente.
Las RNasas H2 son más grandes que las H1 y normalmente tienen hélices adicionales. La organización de dominios de las enzimas varía; Algunos miembros procarióticos y la mayoría de los eucariotas del grupo H1 tienen un pequeño dominio adicional en el extremo N conocido como "dominio de unión híbrido", que facilita la unión a dúplex híbridos de ARN:ADN y, a veces, confiere una mayor procesividad. Mientras que todos los miembros del grupo H1 y los miembros procarióticos del grupo H2 funcionan como monómeros, las enzimas H2 eucarióticas son heterotrímeros obligados. Las enzimas procarióticas HIII son miembros del grupo H2 más amplio y comparten la mayoría de las características estructurales con H2, con la adición de un dominio de unión a la caja TATA N-terminal. Los dominios retrovirales de RNasa H que se encuentran en proteínas transcriptasa inversa multidominio tienen estructuras que se parecen mucho al grupo H1.
Las RNasas H1 se han estudiado exhaustivamente para explorar las relaciones entre la estructura y la actividad enzimática. También se utilizan, especialmente la E. coli homólogo, como sistemas modelo para estudiar el plegamiento de proteínas. Dentro del grupo H1, se ha identificado una relación entre una mayor afinidad de unión al sustrato y la presencia de elementos estructurales que consisten en una hélice y un bucle flexible que proporcionan una superficie de unión al sustrato más grande y más básica. La hélice C tiene una distribución taxonómica dispersa; está presente en el E. coli y homólogos de la RNasa H1 humana y están ausentes en el dominio de la RNasa H del VIH, pero existen ejemplos de dominios retrovirales con hélices C.
Función
Las enzimas ribonucleasa H escinden los enlaces fosfodiéster del ARN en un híbrido de ARN:ADN de doble cadena, dejando un núcleo de 3' hidroxilo y un 5' grupo fosfato en cada extremo del sitio de corte con un mecanismo de catálisis de dos iones metálicos, en el que dos cationes divalentes, como Mg2+ y Mn2+, participan directamente en la función catalítica. Dependiendo de las diferencias en sus secuencias de aminoácidos, estas RNasas H se clasifican en RNasas H tipo 1 y tipo 2. Las RNasas H tipo 1 tienen RNasas H1 procarióticas y eucariotas y RNasas H retrovirales. Las RNasas H tipo 2 tienen RNasas H2 procarióticas y eucariotas y ARNasa bacteriana H3. Estas RNasas H existen en forma monomérica, excepto las RNasas H2 eucarióticas, que existen en forma heterotrimérica. Las ARNasa H1 y H2 tienen distintas preferencias de sustrato y funciones distintas pero superpuestas en la célula. En procariotas y eucariotas inferiores, ninguna enzima es esencial, mientras que se cree que ambas son esenciales en eucariotas superiores. La actividad combinada de las enzimas H1 y H2 está asociada con el mantenimiento de la estabilidad del genoma debido a la acción de las enzimas. degradación del componente de ARN de los bucles R.
Ribonucleasa H1
Las enzimas ribonucleasa H1 requieren al menos cuatro pares de bases que contienen ribonucleótidos en un sustrato y no pueden eliminar un solo ribonucleótido de una cadena que de otro modo estaría compuesta de desoxirribonucleótidos. Por esta razón, se considera poco probable que las enzimas RNasa H1 estén involucradas en el procesamiento de cebadores de ARN de fragmentos de Okazaki durante la replicación del ADN. La RNasa H1 no es esencial en los organismos unicelulares donde se ha investigado; en E. coli, los knockouts de RNasa H1 confieren un fenotipo sensible a la temperatura, y en S. cerevisiae, producen defectos en la respuesta al estrés.
En muchos eucariotas, incluidos los mamíferos, los genes de la RNasa H1 incluyen una secuencia de direccionamiento mitocondrial, lo que conduce a la expresión de isoformas con y sin el MTS presente. Como resultado, la RNasa H1 se localiza tanto en las mitocondrias como en el núcleo. En modelos de ratones knockout, los mutantes nulos de RNasa H1 son letales durante la embriogénesis debido a defectos en la replicación del ADN mitocondrial. Los defectos en la replicación del ADN mitocondrial inducidos por la pérdida de RNasa H1 probablemente se deban a defectos en el procesamiento del bucle R.
Ribonucleasa H2
En procariotas, la RNasa H2 es enzimáticamente activa como proteína monomérica. En eucariotas, es un heterotrímero obligado compuesto por una subunidad catalítica A y subunidades estructurales B y C. Mientras que la subunidad A es estrechamente homóloga a la RNasa H2 procariótica, las subunidades B y C no tienen homólogos aparentes en procariotas y están mal conservadas en el nivel de secuencia incluso entre eucariotas. La subunidad B media las interacciones proteína-proteína entre el complejo H2 y PCNA, que localiza H2 en focos de replicación.
Las enzimas H2 tanto procarióticas como eucariotas pueden escindir ribonucleótidos individuales en una hebra. sin embargo, tienen patrones de escisión y preferencias de sustrato ligeramente diferentes: las enzimas procarióticas tienen una procesividad menor e hidrolizan ribonucleótidos sucesivos de manera más eficiente que los ribonucleótidos con un extremo de 5' desoxirribonucleótido, mientras que las enzimas eucariotas son más procesivas e hidrolizan ambos tipos de sustrato con similar eficiencia. La especificidad de sustrato de la RNasa H2 le otorga un papel en la reparación por escisión de ribonucleótidos, eliminando los ribonucleótidos mal incorporados del ADN, además del procesamiento del bucle R. Aunque tanto H1 como H2 están presentes en el núcleo de las células de mamíferos, H2 es la fuente dominante de actividad de la RNasa H allí y es importante para mantener la estabilidad del genoma.
Algunos procariotas poseen un gen adicional de tipo H2 denominado RNasa HIII en la nomenclatura de números romanos utilizada para los genes procarióticos. Las proteínas HIII están más estrechamente relacionadas con el grupo H2 por identidad de secuencia y similitud estructural, pero tienen preferencias de sustrato que se parecen más a H1. A diferencia de HI y HII, que están ampliamente distribuidos entre procariotas, HIII se encuentra sólo en unos pocos organismos con una distribución taxonómica dispersa; es algo más común en arqueas y rara vez o nunca se encuentra en el mismo genoma procariótico que HI.
Mecanismo

El sitio activo de casi todas las RNasas H contiene cuatro residuos de aminoácidos cargados negativamente, conocidos como motivo DEDD; a menudo una histidina, p. en el VIH-1, humano o E. coli también está presente.
Los residuos cargados unen dos iones metálicos que son necesarios para la catálisis; En condiciones fisiológicas se trata de iones de magnesio, pero el manganeso también suele favorecer la actividad enzimática, mientras que el calcio o una alta concentración de Mg2+ inhibe la actividad.
Basado en evidencia experimental y simulaciones por computadora, la enzima activa una molécula de agua unida a uno de los iones metálicos con la histidina conservada. El estado de transición es de naturaleza asociativa y forma un intermedio con el grupo saliente fosfato protonado y alcóxido desprotonado. El grupo saliente se protona a través del glutamato, que tiene un pKa elevado y es probable que se protone. El mecanismo es similar al de la RNasa T y la subunidad RuvC de la enzima Cas9, que también utilizan un mecanismo de histidina y de iones de dos metales.
El mecanismo de liberación del producto escindido aún no está resuelto. La evidencia experimental de cristalografía de resolución temporal y nucleasas similares apunta a la participación de un tercer ion en la reacción reclutado en el sitio activo.
En biología humana
El genoma humano contiene cuatro genes que codifican la RNasa H:
- RNASEH1, un ejemplo del subtipo H1 (monomeric)
- RNASEH2A, la subunidad catalítica del complejo H2 trimérico
- RNASEH2B, una subunidad estructural del complejo H2 trimérico
- RNASEH2C, una subunidad estructural del complejo H2 trimérico
Además, el material genético de origen retroviral aparece con frecuencia en el genoma, lo que refleja la integración de los genomas de los retrovirus endógenos humanos. Dichos eventos de integración dan como resultado la presencia de genes que codifican la transcriptasa inversa retroviral, que incluye un dominio RNasa H. Un ejemplo es ERVK6. Los retrotransposones de repetición terminal larga (LTR) y de repetición terminal no larga (no LTR) también son comunes en el genoma y a menudo incluyen sus propios dominios de RNasa H, con una historia evolutiva compleja.
Papel en la enfermedad

En estudios pequeños, las mutaciones en la RNasa H1 humana se han asociado con la oftalmoplejía externa progresiva crónica, una característica común de la enfermedad mitocondrial.
Las mutaciones en cualquiera de las tres subunidades de la RNasa H2 están bien establecidas como causas de un raro trastorno genético conocido como síndrome de Aicardi-Goutières (AGS), que se manifiesta como síntomas neurológicos y dermatológicos a una edad temprana. Los síntomas de AGS se parecen mucho a los de una infección viral congénita y se asocian con una regulación positiva inadecuada del interferón tipo I. La AGS también puede ser causada por mutaciones en otros genes: TREX1, SAMHD1, ADAR y MDA5/IFIH1, todos los cuales participan en el procesamiento de ácidos nucleicos. La caracterización de la distribución mutacional en una población de pacientes con AGS encontró el 5 % de todas las mutaciones de AGS en RNASEH2A, el 36 % en 2B y el 12 % en 2C. Las mutaciones en 2B se han asociado con un deterioro neurológico algo más leve y con una ausencia de regulación positiva del gen inducida por interferón que puede detectarse en pacientes con otros genotipos asociados a AGS.
En virus

Dos grupos de virus utilizan la transcripción inversa como parte de sus ciclos de vida: los retrovirus, que codifican sus genomas en ARN monocatenario y se replican a través de un intermediario de ADN bicatenario; y virus dsDNA-RT, que replican sus genomas de ADN bicatenario a través de un "pregenoma" intermedio. Los ejemplos patógenos incluyen el virus de la inmunodeficiencia humana y el virus de la hepatitis B, respectivamente. Ambos codifican grandes proteínas transcriptasa inversa (RT) multifuncionales que contienen dominios RNasa H.
Las proteínas retrovirales RT del VIH-1 y el virus de la leucemia murina son los miembros de la familia mejor estudiados. La RT retroviral es responsable de convertir el virus & # 39; genoma de ARN monocatenario en ADN bicatenario. Este proceso requiere tres pasos: primero, la actividad de la ADN polimerasa dependiente de ARN produce ADN de cadena negativa a partir de la plantilla de ARN de cadena positiva, generando un intermedio híbrido de ARN:ADN; en segundo lugar, se destruye la cadena de ARN; y tercero, la actividad de la ADN polimerasa dependiente de ADN sintetiza ADN de cadena positiva, generando ADN de doble cadena como producto final. El segundo paso de este proceso lo lleva a cabo un dominio RNasa H ubicado en el extremo C de la proteína RT.
La RNasa H realiza tres tipos de acciones de escisión: degradación no específica del genoma de ARN de cadena positiva, eliminación específica del cebador de ARNt de cadena negativa y eliminación del cebador del tracto de polipurina rico en purinas (PPT) de cadena positiva.. La RNasa H desempeña un papel en el cebado de la cadena plus, pero no en el método convencional de sintetizar una nueva secuencia de cebador. Más bien, la RNasa H crea un "cebador" del PPT que es resistente a la escisión de la RNasa H. Al eliminar todas las bases excepto el PPT, el PPT se utiliza como marcador para el final de la región U3 de su repetición terminal larga.
Dado que la actividad de la RNasa H es necesaria para la proliferación viral, este dominio se ha considerado un objetivo farmacológico para el desarrollo de fármacos antirretrovirales utilizados en el tratamiento del VIH/SIDA y otras afecciones causadas por retrovirus. Se han identificado inhibidores de la RNasa H retroviral de varios quimiotipos diferentes, muchos de los cuales tienen un mecanismo de acción basado en la quelación de los cationes del sitio activo. Los inhibidores de la transcriptasa inversa que inhiben específicamente la función polimerasa de RT tienen un uso clínico generalizado, pero no los inhibidores de la función RNasa H; es la única función enzimática codificada por el VIH que aún no está dirigida a los fármacos de uso clínico.
Evolución
Las RNasas H están ampliamente distribuidas y ocurren en todos los ámbitos de la vida. La familia pertenece a una superfamilia más grande de enzimas nucleasas y se considera evolutivamente antigua. En los genomas procarióticos, a menudo están presentes múltiples genes de RNasa H, pero hay poca correlación entre la aparición de genes HI, HII y HIII y las relaciones filogenéticas generales, lo que sugiere que la transferencia horizontal de genes puede haber desempeñado un papel en el establecimiento de la distribución de estas enzimas. Las RNasa HI y HIII rara vez o nunca aparecen en el mismo genoma procariótico. Cuando el genoma de un organismo contiene más de un gen de RNasa H, a veces tienen diferencias significativas en el nivel de actividad. Se ha sugerido que estas observaciones reflejan un patrón evolutivo que minimiza la redundancia funcional entre los genes de la RNasa H. La ARNasa HIII, que es exclusiva de los procariotas, tiene una distribución taxonómica dispersa y se encuentra tanto en bacterias como en arqueas; se cree que se separó del HII bastante pronto.
La trayectoria evolutiva de la RNasa H2 en eucariotas, especialmente el mecanismo por el cual los homólogos eucariotas se convirtieron en heterotrímeros obligados, no está clara; las subunidades B y C no tienen homólogos aparentes en los procariotas.
Aplicaciones
Debido a que la RNasa H degrada específicamente solo el ARN en híbridos de ARN:ADN de doble cadena, se usa comúnmente como reactivo de laboratorio en biología molecular. Preparaciones purificadas de E. coli RNase HI y HII están disponibles comercialmente. La RNasa HI se utiliza a menudo para destruir el molde de ARN después de la síntesis de ADN complementario de primera cadena (ADNc) mediante transcripción inversa. También se puede utilizar para escindir secuencias de ARN específicas en presencia de segmentos cortos de ADN complementarios. Para la detección se pueden utilizar técnicas altamente sensibles, como la resonancia de plasmones superficiales. La RNasa HII se puede utilizar para degradar el componente cebador de ARN de un fragmento de Okazaki o para introducir mellas monocatenarias en posiciones que contienen un ribonucleótido. Se ha descrito una variante de la PCR de inicio en caliente, conocida como PCR dependiente de RNasa H o rhPCR, que utiliza una RNasa HII termoestable de la arqueona hipertermófila Pyrococcus abyssi. Es de destacar que la proteína inhibidora de ribonucleasa comúnmente utilizada como reactivo no es eficaz para inhibir la actividad de HI o HII.
Historia
Las ribonucleasas H se descubrieron por primera vez en el laboratorio de Peter Hausen cuando los investigadores encontraron actividad de endonucleasa híbrida ARN:ADN en el timo de la pantorrilla en 1969 y le dieron el nombre de "ribonucleasa H" para designar su especificidad híbrida. Posteriormente se descubrió la actividad de la RNasa H en E. coli y en una muestra de oncovirus con genomas de ARN durante los primeros estudios de transcripción inversa viral. Posteriormente quedó claro que el extracto de timo de ternera contenía más de una proteína con actividad RNasa H y que E. coli contenía dos genes de RNasa H. Originalmente, la enzima ahora conocida como RNasa H2 en eucariotas se designó H1 y viceversa, pero los nombres de las enzimas eucariotas se cambiaron para que coincidieran con los de E. coli para facilitar el análisis comparativo, dando lugar a la nomenclatura moderna en la que las enzimas procarióticas se designan con números romanos y las enzimas eucariotas con números arábigos. La RNasa HIII procariótica, reportada en 1999, fue el último subtipo de RNasa H identificado.
Caracterizar la ARNasa H2 eucariota fue históricamente un desafío, en parte debido a su baja abundancia. Los cuidadosos esfuerzos de purificación de la enzima sugirieron que, a diferencia de la E. coli RNasa H2, la enzima eucariota tenía múltiples subunidades. El S. cerevisiae homólogo de E. coli (es decir, la subunidad H2A) fue fácilmente identificable mediante bioinformática cuando se secuenció el genoma de la levadura, pero se descubrió que la proteína correspondiente no tenía actividad enzimática de forma aislada. Finalmente, las subunidades B y C de la levadura se aislaron mediante copurificación y se descubrió que eran necesarias para la actividad enzimática. Sin embargo, las subunidades B y C de la levadura tienen una identidad de secuencia muy baja con sus homólogos en otros organismos, y las proteínas humanas correspondientes se identificaron de manera concluyente sólo después de que se descubrió que las mutaciones en las tres causaban el síndrome de Aicardi-Goutières.