Regulación de la expresión genética.
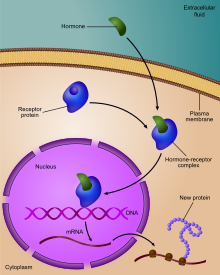

Regulación de la expresión genética, o regulación genética, incluye una amplia gama de mecanismos que utilizan las células para aumentar o disminuir la producción de productos genéticos específicos (proteínas o ARN). En biología se observan ampliamente programas sofisticados de expresión genética, por ejemplo para desencadenar vías de desarrollo, responder a estímulos ambientales o adaptarse a nuevas fuentes de alimentos. Prácticamente cualquier paso de la expresión génica se puede modular, desde el inicio de la transcripción hasta el procesamiento del ARN y la modificación postraduccional de una proteína. A menudo, un regulador genético controla a otro, y así sucesivamente, en una red reguladora de genes.
La regulación genética es esencial para virus, procariotas y eucariotas, ya que aumenta la versatilidad y adaptabilidad de un organismo al permitir que la célula exprese proteínas cuando sea necesario. Aunque ya en 1951, Barbara McClintock demostró la interacción entre dos loci genéticos, el Activador (Ac) y el Disociador (Ds), en la formación del color de las semillas de maíz, el primer descubrimiento Se considera ampliamente que un sistema de regulación genética es la identificación en 1961 del operón lac, descubierto por François Jacob y Jacques Monod, en el que algunas enzimas implicadas en el metabolismo de la lactosa se expresan mediante E. coli sólo en presencia de lactosa y ausencia de glucosa.
En los organismos multicelulares, la regulación genética impulsa la diferenciación celular y la morfogénesis en el embrión, lo que lleva a la creación de diferentes tipos de células que poseen diferentes perfiles de expresión genética de la misma secuencia del genoma. Aunque esto no explica cómo se originó la regulación genética, los biólogos evolutivos la incluyen como una explicación parcial de cómo funciona la evolución a nivel molecular, y es central para la ciencia de la biología evolutiva del desarrollo ("evo-devo").
Etapas reguladas de la expresión genética
Cualquier paso de la expresión genética puede modularse, desde la señalización hasta la transcripción y la modificación postraduccional de una proteína. La siguiente es una lista de etapas en las que se regula la expresión genética, donde el punto más utilizado es el inicio de la transcripción, la primera etapa de la transcripción:
- Transducción de señales
- Cromatina, remodelación de cromatina, dominios de cromatina
- Transcripción
- Modificación post-transcripción
- Transporte RNA
- Traducción
- mRNA degradation
Modificación del ADN

En eucariotas, la accesibilidad de grandes regiones de ADN puede depender de su estructura de cromatina, que puede alterarse como resultado de modificaciones de histonas dirigidas por la metilación del ADN, ncRNA o proteína de unión al ADN. Por lo tanto, estas modificaciones pueden regular hacia arriba o hacia abajo la expresión de un gen. Algunas de estas modificaciones que regulan la expresión genética son hereditarias y se denominan regulación epigenética.
Estructural
La transcripción del ADN está dictada por su estructura. En general, la densidad de su empaquetamiento es indicativa de la frecuencia de transcripción. Los complejos de proteínas octaméricas llamados histonas, junto con un segmento de ADN enrollado alrededor de las ocho proteínas histonas (denominadas en conjunto nucleosoma), son responsables de la cantidad de superenrollamiento del ADN, y estos complejos pueden modificarse temporalmente mediante procesos como la fosforilación o de forma más permanente. modificado por procesos como la metilación. Se considera que tales modificaciones son responsables de cambios más o menos permanentes en los niveles de expresión genética.
Química
(feminine)La metilación del ADN es un método común de silenciamiento de genes. El ADN suele ser metilado por enzimas metiltransferasas en nucleótidos de citosina en una secuencia de dinucleótidos CpG (también denominadas "islas CpG" cuando están densamente agrupadas). El análisis del patrón de metilación en una región determinada del ADN (que puede ser un promotor) se puede lograr mediante un método llamado mapeo con bisulfito. Los residuos de citosina metilados no se modifican con el tratamiento, mientras que los no metilados se transforman en uracilo. Las diferencias se analizan mediante secuenciación de ADN o mediante métodos desarrollados para cuantificar SNP, como Pyrosequencing (Biotage) o MassArray (Sequenom), midiendo las cantidades relativas de C/T en el dinucleótido CG. Se cree que los patrones anormales de metilación están implicados en la oncogénesis.
La acetilación de histonas también es un proceso importante en la transcripción. Las enzimas histona acetiltransferasa (HAT), como la proteína de unión a CREB, también disocian el ADN del complejo de histonas, lo que permite que continúe la transcripción. A menudo, la metilación del ADN y la desacetilación de histonas funcionan juntas en el silenciamiento de genes. La combinación de los dos parece ser una señal para que el ADN se empaquete más densamente, lo que reduce la expresión genética.
Regulación de la transcripción

La regulación de la transcripción controla así cuándo se produce la transcripción y cuánto ARN se crea. La transcripción de un gen por la ARN polimerasa puede regularse mediante varios mecanismos. Los factores de especificidad alteran la especificidad de la ARN polimerasa por un promotor o conjunto de promotores determinado, lo que hace que sea más o menos probable que se una a ellos (es decir, factores sigma utilizados en la transcripción procariótica). Los represores se unen al operador, codificando secuencias en la cadena de ADN que están cerca o superpuestas a la región promotora, impidiendo el progreso de la ARN polimerasa a lo largo de la cadena, impidiendo así la expresión del gen. La imagen de la derecha muestra la regulación por parte de un represor en el operón lac. Los factores de transcripción generales colocan la ARN polimerasa al comienzo de una secuencia codificante de proteínas y luego liberan la polimerasa para transcribir el ARNm. Los activadores mejoran la interacción entre la ARN polimerasa y un promotor particular, fomentando la expresión del gen. Los activadores hacen esto aumentando la atracción de la ARN polimerasa por el promotor, a través de interacciones con subunidades de la ARN polimerasa o indirectamente cambiando la estructura del ADN. Los potenciadores son sitios en la hélice del ADN que están unidos por activadores para formar un bucle en el ADN y llevar un promotor específico al complejo de iniciación. Los potenciadores son mucho más comunes en eucariotas que en procariotas, de los que sólo existen unos pocos ejemplos (hasta la fecha). Los silenciadores son regiones de secuencias de ADN que, cuando se unen a factores de transcripción particulares, pueden silenciar la expresión del gen.
Regulación por ARN
El ARN puede ser un importante regulador de la actividad genética, p. por microARN (miARN), ARN antisentido o ARN largo no codificante (lncRNA). Los LncRNA se diferencian de los mRNA en el sentido de que tienen ubicaciones y funciones subcelulares específicas. Se descubrió por primera vez que estaban ubicados en el núcleo y la cromatina, y en la actualidad sus localizaciones y funciones son muy diversas. Algunos todavía residen en la cromatina donde interactúan con las proteínas. Si bien este lncRNA afecta en última instancia a la expresión genética en trastornos neuronales como la enfermedad de Parkinson, Huntington y Alzheimer, otros, como PNCTR (transcriptores no codificantes ricos en pirimidina), desempeñan un papel en el cáncer de pulmón. Dada su función en las enfermedades, los lncRNA son biomarcadores potenciales y pueden ser objetivos útiles para medicamentos o terapia génica, aunque todavía no existen medicamentos aprobados que se dirijan a los lncRNA. El número de lncRNA en el genoma humano sigue estando mal definido, pero algunas estimaciones oscilan entre 16.000 y 100.000 genes lnc.
Regulación de genes epigenéticos

La epigenética se refiere a la modificación de genes sin cambiar la secuencia de ADN o ARN. Las modificaciones epigenéticas también son un factor clave que influye en la expresión genética. Ocurren en el ADN genómico y las histonas y sus modificaciones químicas regulan la expresión genética de una manera más eficiente. Hay varias modificaciones del ADN (generalmente metilación) y más de 100 modificaciones del ARN en células de mamíferos”. Esas modificaciones dan como resultado una unión alterada de la proteína al ADN y un cambio en la estabilidad del ARN y la eficiencia de la traducción.
Casos especiales en biología y enfermedades humanas
Regulación de la transcripción en cáncer
En los vertebrados, la mayoría de los promotores de genes contienen una isla CpG con numerosos sitios CpG. Cuando muchos de los sitios CpG promotores de un gen están metilados, el gen queda silenciado. Los cánceres colorrectales suelen tener de 3 a 6 mutaciones de conductor y de 33 a 66 mutaciones de autoestopista o pasajero. Sin embargo, el silenciamiento transcripcional puede ser más importante que la mutación a la hora de provocar la progresión al cáncer. Por ejemplo, en los cánceres colorrectales, la metilación de la isla CpG silencia transcripcionalmente entre 600 y 800 genes (ver regulación de la transcripción en el cáncer). La represión transcripcional en el cáncer también puede ocurrir por otros mecanismos epigenéticos, como la expresión alterada de microARN. En el cáncer de mama, la represión transcripcional de BRCA1 puede ocurrir con más frecuencia por microARN-182 sobreexpresado que por hipermetilación del promotor BRCA1 (consulte Baja expresión de BRCA1 en cánceres de mama y ovario).
Regulación de la transcripción en adicciones
Una de las características cardinales de la adicción es su persistencia. Los cambios de comportamiento persistentes parecen deberse a cambios duraderos, resultantes de alteraciones epigenéticas que afectan la expresión genética, dentro de regiones particulares del cerebro. Las drogas de abuso provocan tres tipos de alteración epigenética en el cerebro. Estos son (1) acetilaciones y metilaciones de histonas, (2) metilación del ADN en sitios CpG y (3) regulación negativa o positiva epigenética de microARN. (Consulte Epigenética de la adicción a la cocaína para obtener algunos detalles).
La ingesta crónica de nicotina en ratones altera el control epigenético de la expresión genética de las células cerebrales mediante la acetilación de histonas. Esto aumenta la expresión en el cerebro de la proteína FosB, importante en la adicción. La adicción al cigarrillo también se estudió en unos 16.000 seres humanos, incluidos los que nunca habían fumado, los fumadores actuales y los que habían dejado de fumar durante hasta 30 años. En las células sanguíneas, más de 18.000 sitios CpG (de los aproximadamente 450.000 sitios CpG analizados en el genoma) tenían con frecuencia metilación alterada entre los fumadores actuales. Estos sitios CpG se produjeron en más de 7.000 genes, o aproximadamente un tercio de los genes humanos conocidos. La mayoría de los sitios CpG con metilación diferencial regresaron al nivel de los no fumadores dentro de los cinco años posteriores a dejar de fumar. Sin embargo, 2.568 CpG entre 942 genes permanecieron metilados de manera diferencial en ex fumadores y en quienes nunca fumaron. Estos cambios epigenéticos restantes pueden verse como “cicatrices moleculares” que pueden afectar la expresión genética.
En modelos de roedores, las drogas de abuso, incluidas la cocaína, la metanfetamina, el alcohol y los productos del humo del tabaco, causan daños en el ADN del cerebro. Durante la reparación de daños en el ADN, algunos eventos de reparación individuales pueden alterar la metilación del ADN y/o las acetilaciones o metilaciones de histonas en los sitios de daño y, por lo tanto, pueden contribuir a dejar una cicatriz epigenética en la cromatina.
Es probable que estas cicatrices epigenéticas contribuyan a los cambios epigenéticos persistentes que se encuentran en la adicción.
Regulación de la transcripción en el aprendizaje y la memoria

En los mamíferos, la metilación de la citosina (ver figura) en el ADN es un mediador regulador importante. Las citosinas metiladas se producen principalmente en secuencias de dinucleótidos donde la citosina va seguida de una guanina, un sitio CpG. El número total de sitios CpG en el genoma humano es de aproximadamente 28 millones. y generalmente alrededor del 70% de todos los sitios CpG tienen una citosina metilada.
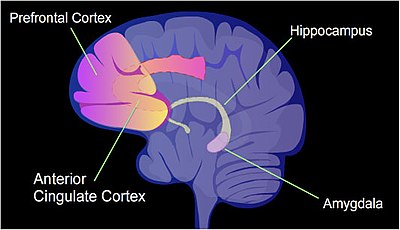
En una rata, una experiencia de aprendizaje dolorosa, un condicionamiento contextual del miedo, puede resultar en un recuerdo aterrador para toda la vida después de un único evento de entrenamiento. La metilación de la citosina está alterada en las regiones promotoras de aproximadamente el 9,17% de todos los genes del ADN de las neuronas del hipocampo de una rata que ha sido sometida a una breve experiencia de condicionamiento del miedo. El hipocampo es donde inicialmente se almacenan los nuevos recuerdos.
La metilación de CpG en una región promotora de un gen reprime la transcripción, mientras que la metilación de CpG en el cuerpo de un gen aumenta la expresión. Las enzimas TET desempeñan un papel central en la desmetilación de citosinas metiladas. La desmetilación de CpG en un promotor de gen mediante la actividad de la enzima TET aumenta la transcripción del gen.
Cuando se aplica el condicionamiento del miedo contextual a una rata, se producen más de 5.000 regiones metiladas diferencialmente (DMR) (de 500 nucleótidos cada una) en el genoma neural del hipocampo de la rata, tanto una hora como 24 horas después del condicionamiento en el hipocampo. Esto hace que alrededor de 500 genes estén regulados positivamente (a menudo debido a la desmetilación de los sitios CpG en una región promotora) y alrededor de 1000 genes estén regulados negativamente (a menudo debido a la 5-metilcitosina recién formada en los sitios CpG de una región promotora). El patrón de genes inducidos y reprimidos dentro de las neuronas parece proporcionar una base molecular para formar el primer recuerdo transitorio de este evento de entrenamiento en el hipocampo del cerebro de la rata.
Regulación postranscripcional
Después de que se transcribe el ADN y se forma el ARNm, debe haber algún tipo de regulación sobre cuánto se traduce el ARNm en proteínas. Las células hacen esto modulando la protección, el empalme, la adición de una cola Poly(A), las tasas de exportación nuclear específicas de la secuencia y, en varios contextos, el secuestro de la transcripción de ARN. Estos procesos ocurren en eucariotas pero no en procariotas. Esta modulación es el resultado de una proteína o transcrito que, a su vez, está regulado y puede tener afinidad por determinadas secuencias.
Tres regiones principales no traducidas y microARN
Tres regiones principales no traducidas (3'-UTR) de los ARN mensajeros (ARNm) a menudo contienen secuencias reguladoras que influyen postranscripcionalmente en la expresión génica. Estas 3'-UTR suelen contener tanto sitios de unión para microARN (miARN) como para proteínas reguladoras. Al unirse a sitios específicos dentro de la 3'-UTR, los miARN pueden disminuir la expresión genética de varios ARNm inhibiendo la traducción o provocando directamente la degradación de la transcripción. La 3'-UTR también puede tener regiones silenciadoras que se unen a proteínas represoras que inhiben la expresión de un ARNm.
La 3'-UTR a menudo contiene elementos de respuesta de miARN (MRE). Los MRE son secuencias a las que se unen los miARN. Estos son motivos prevalentes dentro de las 3'-UTR. Entre todos los motivos regulatorios dentro de las 3'-UTR (por ejemplo, incluidas las regiones silenciadoras), los MRE constituyen aproximadamente la mitad de los motivos.
En 2014, el sitio web miRBase, un archivo de secuencias y anotaciones de miARN, enumeraba 28.645 entradas en 233 especies biológicas. De estos, 1.881 miARN estaban en loci de miARN humanos anotados. Se predijo que los miARN tendrían un promedio de aproximadamente cuatrocientos ARNm diana (que afectan la expresión de varios cientos de genes). Freidman et al. Se estima que >45 000 sitios objetivo de miARN dentro de las 3'-UTR de ARNm humano se conservan por encima de los niveles de fondo, y >60% de los genes codificadores de proteínas humanas han estado bajo presión selectiva para mantener el emparejamiento con los miARN.
Los experimentos directos muestran que un solo miARN puede reducir la estabilidad de cientos de ARNm únicos. Otros experimentos muestran que un solo miARN puede reprimir la producción de cientos de proteínas, pero que esta represión suele ser relativamente leve (menos del doble).
Los efectos de la desregulación de la expresión genética de los miARN parecen ser importantes en el cáncer. Por ejemplo, en el caso de los cánceres gastrointestinales, un artículo de 2015 identificó nueve miARN como alterados epigenéticamente y eficaces para regular negativamente las enzimas reparadoras del ADN.
Los efectos de la desregulación de la expresión genética de los miARN también parecen ser importantes en los trastornos neuropsiquiátricos, como la esquizofrenia, el trastorno bipolar, el trastorno depresivo mayor, la enfermedad de Parkinson, la enfermedad de Alzheimer y los trastornos del espectro autista.
Reglamento de la traducción
La traducción del ARNm también puede controlarse mediante varios mecanismos, principalmente en el nivel de iniciación. De hecho, el reclutamiento de la subunidad ribosomal pequeña puede modularse mediante la estructura secundaria del ARNm, la unión de ARN antisentido o la unión a proteínas. Tanto en procariotas como en eucariotas, existe una gran cantidad de proteínas de unión a ARN, que a menudo son dirigidas a su secuencia objetivo por la estructura secundaria del transcrito, que puede cambiar dependiendo de ciertas condiciones, como la temperatura o la presencia de un ligando (aptámero).. Algunas transcripciones actúan como ribozimas y autorregulan su expresión.
Ejemplos de regulación genética
- La inducción enzimática es un proceso en el que una molécula (por ejemplo, un medicamento) induce (es decir, inicia o mejora) la expresión de una enzima.
- La inducción de proteínas de choque térmico en la mosca de la fruta Drosophila melanogaster.
- El Operon Lac es un ejemplo interesante de cómo se puede regular la expresión génica.
- Los virus, a pesar de tener sólo unos pocos genes, poseen mecanismos para regular su expresión génica, típicamente en una fase temprana y tardía, utilizando sistemas collinear regulados por anti-terminadores (fago de lambda) o moduladores de espionaje (VIH).
- Gal4 es un activador transcripcional que controla la expresión de GAL1, GAL7, y GAL10 (todos los cuales código para el metabólico de la galactosa en levadura). El sistema GAL4/UAS se ha utilizado en una variedad de organismos a través de diversos phyla para estudiar la expresión génica.
Biología del desarrollo
Un gran número de sistemas regulatorios estudiados provienen de la biología del desarrollo. Ejemplos incluyen:
- La colinearidad del grupo de genes Hox con su patrón antero-posterior anidado
- Generación paterna de la mano (digits - interdigits): el gradiente de hedgehog sónico (factor de inducción secreto) de la zona de actividad polarizante en la extremidad, que crea un gradiente de Gli activo, que activa Gremlin, que inhibe los BMPs también secretados en la extremidad, resulta en la formación de un patrón de actividad alternado como resultado de esta reacción.
- La somitogénesis es la creación de segmentos (somites) de un tejido uniforme (Mesoderm presomítico). Se forman secuencialmente de anteriores a posteriores. Esto se logra en amniotes posiblemente por medio de dos gradientes opuestos, ácido retinoico en el anterior (frontera de onda) y Wnt y Fgf en el posterior, junto a un patrón oscilante (reloj de segmento) compuesto por FGF + Notch y Wnt en antifase.
- La determinación sexual en la soma de una Drosophila requiere la detección de la relación de genes autosómicos con genes codificados por cromosomas sexuales, lo que resulta en la producción de factor de espolvoramiento sin sexo en las mujeres, dando lugar a la isoforma femenina de doble sexo.
Circuitos
Regulación al alza y regulación a la baja
La regulación positiva es un proceso que ocurre dentro de una célula desencadenado por una señal (que se origina interna o externa a la célula), lo que resulta en una mayor expresión de uno o más genes y, como resultado, de las proteínas codificadas por esos genes. Por el contrario, la regulación negativa es un proceso que resulta en una disminución de la expresión de genes y proteínas correspondientes.
- La regulación se produce, por ejemplo, cuando una célula es deficiente en algún tipo de receptor. En este caso, más proteína receptora se sintetiza y se transporta a la membrana de la célula y, por lo tanto, la sensibilidad de la célula se vuelve a la homeostasis normal, restableciendo.
- La regulación descendente ocurre, por ejemplo, cuando una célula es sobreestimulada por un neurotransmisor, hormona o fármaco durante un período prolongado de tiempo, y la expresión de la proteína receptora se disminuye para proteger la célula (véase también la taquifilaxis).
Sistemas inducibles versus reprimibles

La regulación genética se puede resumir en la respuesta del sistema respectivo:
- Sistemas inducibles - Un sistema inducible está apagado a menos que haya presencia de alguna molécula (llamado inductor) que permita la expresión génica. Se dice que la molécula "induce expresión". La forma en que esto sucede depende de los mecanismos de control, así como de las diferencias entre las células procariotas y eucariotas.
- Sistemas represibles - Un sistema represible está encendido excepto en la presencia de una molécula (llamado un corepressor) que suprime la expresión génica. Se dice que la molécula "reprime la expresión". La forma en que esto sucede depende de los mecanismos de control, así como de las diferencias entre las células procariotas y eucariotas.
El sistema GAL4/UAS es un ejemplo de sistema tanto inducible como reprimible. Gal4 se une a una secuencia de activación ascendente (UAS) para activar la transcripción del casete GAL1/GAL7/GAL10. Por otro lado, una respuesta MIG1 a la presencia de glucosa puede inhibir GAL4 y por tanto detener la expresión del casete GAL1/GAL7/GAL10.
Circuitos teóricos
- Repressor/Inducer: una activación de un sensor resulta en el cambio de expresión de un gen
- retroalimentación negativa: el producto gene desregula su propia producción directa o indirectamente, lo que puede resultar en
- mantener los niveles de transcripción constante/proporcional a un factor
- inhibición de reacciones fugas cuando se une con un bucle de retroalimentación positiva
- crear un oscilador aprovechando el retraso del tiempo de transcripción y traducción, dado que el mRNA y la media vida de proteína es más corta
- retroalimentación positiva: el producto gen aumenta su propia producción directa o indirectamente, lo que puede resultar en
- amplificación de señal
- interruptores bistables cuando dos genes se inhiben y ambos tienen una retroalimentación positiva
- generación de patrones
Métodos de estudio

En general, la mayoría de los experimentos que investigan la expresión diferencial utilizaron extractos de ARN de células completas, llamados niveles de estado estacionario, para determinar qué genes cambiaron y en qué medida. Sin embargo, estos no son informativos sobre dónde se ha producido la regulación y pueden enmascarar procesos regulatorios conflictivos (ver regulación postranscripcional), pero sigue siendo el más comúnmente analizado (PCR cuantitativa y microarrays de ADN).
Al estudiar la expresión genética, existen varios métodos para observar las distintas etapas. En eucariotas estos incluyen:
- El entorno local de la cromatina de la región puede ser determinado por el análisis ChIP-chip mediante la eliminación de la ARN Polymerase II, Histone 3 modificaciones, proteína Trithorax-group, proteína Polycomb-group, o cualquier otro elemento de unión de ADN al que esté disponible un buen anticuerpo.
- Las interacciones epistáticas pueden ser investigadas por análisis de matriz genética sintética
- Debido a la regulación post-transcripción, las tasas de transcripción y los niveles totales de ARN difieren significativamente. Para medir las tasas de transcripción se pueden hacer ensayos nucleares y se están desarrollando nuevos métodos de alto rendimiento, utilizando el etiquetado tiol en lugar de la radioactividad.
- Sólo el 5% del ARN polimerizado en las salidas del núcleo, y no sólo los intrones, los productos abortivos y las transcripciones sin sentido se degradan. Por lo tanto, las diferencias en los niveles nuclear y citoplasmático pueden verse separando las dos fracciones por lisis suave.
- Se puede analizar el aguijón alternativo con una matriz de afilado o con una matriz de afilado (ver la microarraya de ADN).
- All in vivo RNA is complexed as RNPs. La cantidad de transcripciones vinculadas a proteínas específicas también puede ser analizada por RIP-Chip. Por ejemplo, DCP2 dará una indicación de proteína secuestrada; da ribososome-bound e indicación de las transcripciones activas en la transcripción (aunque un método más fechado, llamado fraccionamiento del polígono, sigue siendo popular en algunos laboratorios)
- Los niveles de proteína pueden ser analizados por la espectrometría de masas, que sólo pueden compararse con datos cuantitativos de PCR, ya que los datos de microarray son relativos y no absolutos.
- El ARN y las tasas de degradación de proteínas se miden mediante inhibidores de la transcripción (actinomycin D o α-Amanitina) o inhibidores de la traducción (Cycloheximida), respectivamente.
Notas y referencias
- ^ "¿Pueden encenderse los genes y apagarse en las células?". Referencia de origen genético.
- ^ Bell JT, Pai AA, Pickrell JK, Gaffney DJ, Pique-Regi R, Degner JF, et al. (2011). "Los patrones de metilación de ADN se asocian con la variación de la expresión genética y genética en las líneas celulares de HapMap". Génova Biología. 12 (1): R10. doi:10.1186/gb-2011-12-1-r10. PMC3091299. PMID 21251332.
- ^ Vertino PM, Spillare EA, Harris CC, Baylin SB (abril de 1993). "Los patrones de metilación cromosómica alterados acompañan la transformación inducida por el oncogeno de las células epiteliales bronquiales humanas" (PDF). Cancer Research. 53 (7): 1684–9. PMID 8453642.
- ^ Austin S, Dixon R (junio de 1992). "La proteína potenciadora procariota NTRC tiene una actividad ATPase que es fosforilación y dependiente del ADN". El Diario EMBO. 11 (6): 2219–28. doi:10.1002/j.1460-2075.1992.tb05281.x. PMC556689. PMID 1534752.
- ^ Statello L, Guo CJ, Chen LL, Huarte M (Febrero 2021). "Regulación de genes por ARN largos no codificación y sus funciones biológicas". Reseñas de la naturaleza. Biología celular molecular. 22 (2): 96–118. doi:10.1038/s41580-020-00315-9. ISSN 1471-0072. PMC7754182. PMID 33353982.
- ^ Kan RL, Chen J, Sallam T (julio 2021). "Crosstalk entre mecanismos epitranscriptomicos y epigenéticos en regulación de genes". Tendencias en la genética. 38 (2): 182–193. doi:10.1016/j.tig.2021.06.014. PMC9093201. PMID 34294427. S2CID 236200223.
- ^ Saxonov S, Berg P, Brutlag DL (enero de 2006). "Un análisis genoma de dinucleótidos CpG en el genoma humano distingue dos clases distintas de promotores". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 103 (5): 1412-7. Código:2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC1345710. PMID 16432200.
- ^ Ave A (enero de 2002). "DNA patrones de metilación y memoria epigenética". Genes " Development. 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (Marzo 2013). "Paisajes genomas del cáncer". Ciencia. 339 (6127): 1546–58. Código:2013...339.1546V. doi:10.1126/ciencia.1235122. PMC3749880. PMID 23539594.
- ^ Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, et al. (2014). "MicroRNAs in the DNA Damage/Repair Network and Cancer". International Journal of Genomics. 2014: 820248. doi:10.1155/2014/820248. PMC3926391. PMID 24616890.
- ^ a b Nestler EJ (enero 2014). "Mecanismos electrónicos de adicción a las drogas". Neurofarmacología76 Pt B: 259–68. doi:10.1016/j.neuropharm.2013.04.004. PMC3766384. PMID 23643695.
- ^ a b Robison AJ, Nestler EJ (octubre de 2011). "Mecanismos tradicionales y epigenéticos de la adicción". Reseñas de la naturaleza. Neurociencia. 12 11): 623–37. doi:10.1038/nrn3111. PMC3272277. PMID 21989194.
- ^ Levine A, Huang Y, Drisaldi B, Griffin EA, Pollak DD, Xu S, et al. (noviembre de 2011). "Mecanismo molecular para una droga de gateway: cambios epigenéticos iniciados por la expresión de genes primarios de nicotina por la cocaína". Science Translational Medicina. 3 (107): 107ra109. doi:10.1126/scitranslmed.3003062. PMC4042673. PMID 22049069.
- ^ Joehanes R, Just AC, Marioni RE, Pilling LC, Reynolds LM, Mandaviya PR, et al. (Octubre 2016). "Señalidades Epigenéticas de Fumar Cigarrillo". Circulación: Genética cardiovascular. 9 (5): 436-447. doi:10.1161/CIRCGENETICS.116.001506. PMC5267325. PMID 27651444.
- ^ de Souza MF, Gonçales TA, Steinmetz A, Moura DJ, Saffi J, Gomez R, Barros HM (abril de 2014). "La cocaína induce daño al ADN en distintas áreas cerebrales de ratas hembras bajo diferentes condiciones hormonales". Farmacología Clínica y Experimental. 41 (4): 265-9. doi:10.1111/1440-1681.12218. PMID 24552452. S2CID 20849951.
- ^ Johnson Z, Venters J, Guarraci FA, Zewail-Foote M (junio 2015). "La metanfetamina induce daño al ADN en regiones específicas del cerebro de la rata femenina". Farmacología Clínica y Experimental. 42 (6): 570-5. doi:10.1111/1440-1681.12404. PMID 25867833. S2CID 24182756.
- ^ Tokunaga I, Ishigami A, Kubo S, Gotohda T, Kitamura O (agosto de 2008). "El daño al ADN peroxidativo y la apoptosis en el cerebro de rata tratados con metanfetamina". The Journal of Medical Investigation. 55 (3–4): 241–5. doi:10.2152/jmi.55.241. PMID 18797138.
- ^ Rulten SL, Hodder E, Ripley TL, Stephens DN, Mayne LV (Julio 2008). "Alcohol induce el daño al ADN y la proteína D2 de la anemia Fanconi implicando FANCD2 en las vías de respuesta del daño al ADN en el cerebro". Alcoholismo: Investigación clínica y experimental. 32 (7): 1186–96. doi:10.1111/j.1530-0277.2008.00673.x. PMID 18482162.
- ^ Adhami N, Chen Y, Martins-Green M (octubre de 2017). "Los marcadores de enfermedad se pueden detectar en ratones tan pronto como 4 semanas después de la iniciación de la exposición a niveles de humo de tercera mano equivalentes a los encontrados en hogares de fumadores". Clinical Science. 131 (19): 2409–2426. doi:10.1042/CS20171053. PMID 28912356.
- ^ Dabin J, Fortuny A, Polo SE (junio 2016). "Epigenome Maintenance in Response to DNA Damage". Celda molecular. 62 (5): 712–27. doi:10.1016/j.molcel.2016.04.006. PMC5476208. PMID 27259203.
- ^ Lövkvist C, Dodd IB, Sneppen K, Haerter JO (junio de 2016). "La metilación de ADN en epigenomios humanos depende de la topología local de los sitios de CpG". Nucleic Acids Research. 44 11): 5123–32. doi:10.1093/nar/gkw124. PMC4914085. PMID 26932361.
- ^ Jabbari K, Bernardi G (mayo de 2004). "Metilación Cytosine y frecuencias CpG, TpG (CpA) y TpA". Gene. 333: 143–9. doi:10.1016/j.gene.2004.02.043. PMID 15177689.
- ^ Kim JJ, Jung MW (2006). "Los circuitos y mecanismos neuronales involucrados en el condicionamiento del miedo pavloviano: una revisión crítica". Neurociencia y Biobehavioral Reviews. 30 (2): 188–202. doi:10.1016/j.neubiorev.2005.06.005. PMC4342048. PMID 16120461.
- ^ a b c Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (Julio 2017). "Reorganización epigómica dependiente de la experiencia en el hipocampo". Aprender " Memoria ". 24 (7): 278–288. doi:10.1101/lm.045112.117. PMC5473107. PMID 28620075.
- ^ Weber M, Hellmann I, Stadler MB, Ramos L, Pääbo S, Rebhan M, Schübeler D (abril de 2007). "Distribución, silenciar el potencial y el impacto evolutivo de la metilación del ADN promotor en el genoma humano". Nat.. 39 (4): 457–66. doi:10.1038/ng1990. PMID 17334365. S2CID 22446734.
- ^ Yang X, Han H, De Carvalho DD, Lay FD, Jones PA, Liang G (octubre de 2014). "La metilación del cuerpo de genes puede alterar la expresión del gen y es un objetivo terapéutico en el cáncer". Célula de cáncer. 26 (4): 577-90. doi:10.1016/j.ccr.2014.07.028. PMC4224113. PMID 25263941.
- ^ Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Bernstein BE, Costello JF, Wilkinson MF, Joung JK (diciembre de 2013). "Demetilación de ADN y activación de genes endógenos mediante proteínas de fusión TALE-TET1 programables". Nat. Biotechnol. 31 (12): 1137–42. doi:10.1038/nbt.2726. PMC3858462. PMID 24108092.
- ^ Ogorodnikov A, Kargapolova Y, Danckwardt S (junio de 2016). "Procesamiento y expansión de transcripciones en el extremo mRNA 3 en salud y enfermedad: encontrar el final correcto". Pflügers Archiv. 468 (6): 993–1012. doi:10.1007/s00424-016-1828-3.4893057. PMID 27220521.
- ^ miRBase.org
- ^ a b Friedman RC, Farh KK, Burge CB, Bartel DP (enero de 2009). "La mayoría de los MRNAs mamíferos se conservan objetivos de microRNAs". Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC2612969. PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, et al. (febrero de 2005). "Microarray analysis shows that some micro Los ARN desregularon un gran número de ARN blancos". Naturaleza. 433 (7027): 769–73. código:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (septiembre de 2008). "Cambios de pan en síntesis de proteínas inducidos por microRNAs". Naturaleza. 455 (7209): 58–63. código:2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (septiembre de 2008). "El impacto de microRNAs en la producción de proteínas". Naturaleza. 455 (7209): 64–71. código:2008Natur.455...64B. doi:10.1038/nature07242. PMC2745094. PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, Marques MM (Julio 2011). "Mecanismos y papel de la desregulación del microRNA en el inicio y la progresión del cáncer". Genética y Biología Molecular. 34 (3): 363–70. doi:10.1590/S1415-47572011000300001. PMC3168173. PMID 21931505.
- ^ Bernstein C, Bernstein H (mayo de 2015). "Reducción epígenética de la reparación de ADN en progresión al cáncer gastrointestinal". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC4434036. PMID 25987950.
- ^ Maffioletti E, Tardito D, Gennarelli M, Bocchio-Chiavetto L (2014). "Espia micro del cerebro a la periferia: nuevas pistas de estudios sobre microRNAs en trastornos neuropsiquiátricos". Fronteras en Neurociencia Celular. 8: 75. doi:10.3389/fncel.2014.00075. PMC3949217. PMID 24653674.
- ^ Mellios N, Sur M (2012). "El papel emergente de los microRNAs en la esquizofrenia y los trastornos del espectro autismo". Fronteras en Psiquiatría. 3: 39. doi:10.3389/fpsyt.2012.00039. PMC3336189. PMID 22539927.
- ^ Geaghan M, Cairns MJ (agosto de 2015). "MicroRNA y Dysregulación Posttranscripción en Psiquiatría". Psiquiatría Biológica. 78 (4): 231-9. doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ Barnett JA (julio de 2004). "Una historia de investigación sobre levaduras 7: adaptación enzimática y regulación". Levadura. 21 (9): 703–46. doi:10.1002/yea.1113. PMID 15282797. S2CID 36606279.
- ^ Dequéant ML, Pourquié O (mayo de 2008). "Modificación sensible del eje embrionario vertebrado". Reseñas de la naturaleza. Genética. 9 (5): 370–82. doi:10.1038/nrg2320. PMID 18414404. S2CID 2526914.
- ^ Gilbert SF (2003). Biología del desarrollo, séptima edición, Sunderland, Misa: Sinauer Associates, 65–6. ISBN 0-87893-258-5.
- ^ Nehlin JO, Carlberg M, Ronne H (noviembre de 1991). "Control de genes GAL de levadura por represor MIG1: una cascada transcripcional en la respuesta a la glucosa". El Diario EMBO. 10 (11): 3373–7. doi:10.1002/j.1460-2075.1991.tb04901.x. PMC453065. PMID 1915298.
- ^ Cheadle C, Fan J, Cho-Chung YS, Werner T, Ray J, Do L, et al. (Mayo 2005). "Control de expresión génica durante la activación celular T: regulación alternativa de la transcripción mRNA y estabilidad mRNA". BMC Genomics. 6: 75. doi:10.1186/1471-2164-6-75. PMC1156890. PMID 15907206.
- ^ Jackson DA, Pombo A, Iborra F (Febrero 2000). "El balance para la transcripción: un análisis del metabolismo del ARN nuclear en las células mamíferas". FASEB Journal. 14 (2): 242-54. doi:10.1096/fasebj.14.2.242. PMID 10657981. S2CID 23518786.
- ^ Schwanekamp JA, Sartor MA, Karyala S, Halbleib D, Medvedovic M, Tomlinson CR (2006). "Los análisis de genomas muestran que los niveles de ARN nuclear y citoplasmático están diferentesmente afectados por la dioxina". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1759 (8–9): 388–402. doi:10.1016/j.bbaexp.2006.07.005. PMID 16962184.