Receptor nuclear

En el campo de la biología molecular, los receptores nucleares son una clase de proteínas responsables de detectar esteroides, hormonas tiroideas, vitaminas y otras moléculas determinadas. Estos receptores intracelulares trabajan con otras proteínas para regular la expresión de genes específicos, controlando así el desarrollo, la homeostasis y el metabolismo del organismo.
Los receptores nucleares se unen directamente al ADN y regulan la expresión de los genes adyacentes; por lo tanto, estos receptores se clasifican como factores de transcripción. La regulación de la expresión génica por parte de los receptores nucleares a menudo ocurre en presencia de un ligando, una molécula que afecta el comportamiento del receptor. La unión del ligando a un receptor nuclear da como resultado un cambio conformacional que activa el receptor. El resultado es una regulación positiva o negativa de la expresión génica.
Una propiedad única de los receptores nucleares que los diferencia de otras clases de receptores es su control directo del ADN genómico. Los receptores nucleares desempeñan papeles clave tanto en el desarrollo embrionario como en la homeostasis adulta. Como se analiza a continuación, los receptores nucleares se clasifican según su mecanismo u homología.
Distribución de especies
Los receptores nucleares son específicos de los metazoos (animales) y no se encuentran en protistas, algas, hongos o plantas. Entre los linajes animales de ramificación temprana con genomas secuenciados, se han reportado dos de la esponja Amphimedon queenslandica, dos de la medusa peine Mnemiopsis leidyi, cuatro del placozoo Trichoplax adhaerens y 17 del cnidario Nematostella vectensis. Hay 270 receptores nucleares solo en el gusano redondo Caenorhabditis elegans, 21 en la mosca de la fruta y otros insectos, 73 en el pez cebra. Los humanos, los ratones y las ratas tienen respectivamente 48, 49 y 47 receptores nucleares cada uno.
Ligands

Los ligandos que se unen a los receptores nucleares y los activan incluyen sustancias lipofílicas como las hormonas endógenas, las vitaminas A y D y las hormonas xenobióticas. Debido a que la expresión de una gran cantidad de genes está regulada por los receptores nucleares, los ligandos que activan estos receptores pueden tener efectos profundos en el organismo. Muchos de estos genes regulados están asociados con diversas enfermedades, lo que explica por qué los objetivos moleculares de aproximadamente el 13% de los medicamentos aprobados por la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) se dirigen a los receptores nucleares.
Varios receptores nucleares, denominados receptores huérfanos, no tienen ligandos endógenos conocidos (o al menos generalmente aceptados). Algunos de estos receptores, como FXR, LXR y PPAR, se unen a varios intermediarios metabólicos, como ácidos grasos, ácidos biliares y/o esteroles, con una afinidad relativamente baja. Por lo tanto, estos receptores pueden funcionar como sensores metabólicos. Otros receptores nucleares, como CAR y PXR, parecen funcionar como sensores xenobióticos que regulan positivamente la expresión de las enzimas del citocromo P450 que metabolizan estos xenobióticos.
Estructura
La mayoría de los receptores nucleares tienen masas moleculares de entre 50.000 y 100.000 daltons.
Los receptores nucleares tienen una estructura modular y contienen los siguientes dominios:
- (A-B) Dominio reglamentario N-terminal: Contiene la función de activación 1 (AF-1) cuya acción es independiente de la presencia de ligand. La activación transcripcional de AF-1 es normalmente muy débil, pero sí sinergia con AF-2 en el dominio E (ver más abajo) para producir una regulación más robusta de la expresión génica. El dominio A-B es muy variable en secuencia entre varios receptores nucleares.
- (C) dominio de unión de ADN (DBD): Dominio altamente conservado que contiene dos dedos de zinc que se unen a secuencias específicas de ADN llamados elementos de respuesta hormonal (HRE). Recientemente, un nuevo motivo de dedo de zinc (CHC2) se identifica en NRs parasitarios de alambrado.
- (D) Región de la cuenca: Pensamos ser un dominio flexible que conecta el DBD con el LBD. Influences intracellular trafficking and subcellular distribution with a target peptide sequence.
- (E) Dominio vinculante de ligand (LBD): Moderadamente conservado en secuencia y altamente conservado en estructura entre los diversos receptores nucleares. La estructura de la LBD se conoce como un emparedado alfa helical en el que tres helices anti alfa paralelas (el relleno sandwich) están flanqueados por dos helices alfa de un lado y tres de otro (el "pan"). La cavidad de unión ligando está dentro del interior del LBD y justo debajo de tres bocadillos antialfa helicoidal "filling". Junto con el DBD, el LBD contribuye a la interfaz de dimerización del receptor y, además, une coactivador y proteínas Corepressor. El LBD también contiene la función de activación 2 (AF-2) cuya acción depende de la presencia de ligando atado, controlada por la conformación de helix 12 (H12).
- (F) Dominio C-terminal: Altamente variable en secuencia entre varios receptores nucleares.
Los dominios de unión al ADN (C) y de unión al ligando (E) están bien plegados de forma independiente y son estructuralmente estables, mientras que los dominios N-terminal (A/B), de la región bisagra (D) y opcionalmente C-terminal (F) pueden ser conformacionalmente flexibles y desordenados. Las orientaciones relativas de los dominios son muy diferentes si se comparan tres estructuras cristalinas multidominio conocidas, dos de ellas unidas a DR1 (DBD separados por 1 pb), una unida a DR4 (por 4 pb).
Mecanismo de acción
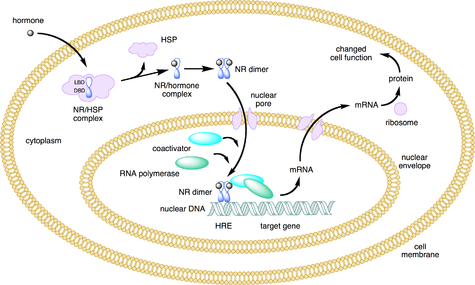

Los receptores nucleares son proteínas multifuncionales que transducen señales de sus ligandos afines. Los receptores nucleares (NR) pueden clasificarse en dos grandes clases según su mecanismo de acción y distribución subcelular en ausencia de ligando.
Las sustancias lipofílicas pequeñas, como las hormonas naturales, se difunden a través de la membrana celular y se unen a los receptores nucleares ubicados en el citosol (NR de tipo I) o el núcleo (NR de tipo II) de la célula. La unión provoca un cambio conformacional en el receptor que, dependiendo de la clase de receptor, desencadena una cascada de eventos posteriores que dirigen a los NR a sitios de regulación de la transcripción del ADN que dan como resultado una regulación positiva o negativa de la expresión génica. Por lo general, funcionan como homo/heterodímeros. Además, también se han identificado dos clases adicionales, el tipo III, que es una variante del tipo I, y el tipo IV, que se unen al ADN como monómeros.
En consecuencia, los receptores nucleares pueden subdividirse en las siguientes cuatro clases mecanísticas:
Tipo I
La unión del ligando a los receptores nucleares de tipo I en el citosol da como resultado la disociación de las proteínas de choque térmico, la homodimerización, la translocación (es decir, el transporte activo) desde el citoplasma hacia el núcleo celular y la unión a secuencias específicas de ADN conocidas como elementos de respuesta hormonal (ERH). Los receptores nucleares de tipo I se unen a los ERH que consisten en dos semisitios separados por una longitud variable de ADN, y el segundo semisitio tiene una secuencia invertida con respecto al primero (repetición invertida). Los receptores nucleares de tipo I incluyen miembros de la subfamilia 3, como el receptor de andrógenos, los receptores de estrógenos, el receptor de glucocorticoides y el receptor de progesterona.
Se ha observado que algunos de los receptores nucleares de la subfamilia 2 de NR pueden unirse a HRE de repetición directa en lugar de a HRE de repetición invertida. Además, algunos receptores nucleares se unen como monómeros o dímeros, con un solo dominio de unión al ADN del receptor unido a un solo HRE de medio sitio. Estos receptores nucleares se consideran receptores huérfanos, ya que sus ligandos endógenos aún se desconocen.
El complejo receptor nuclear/ADN recluta entonces otras proteínas que transcriben el ADN aguas abajo del HRE en ARN mensajero y, finalmente, en proteína, lo que provoca un cambio en la función celular.
Tipo II
Los receptores de tipo II, a diferencia de los de tipo I, se mantienen en el núcleo independientemente del estado de unión del ligando y, además, se unen como heterodímeros (normalmente con RXR) al ADN. En ausencia de ligando, los receptores nucleares de tipo II suelen formar complejos con proteínas correpresoras. La unión del ligando al receptor nuclear provoca la disociación del correpresor y el reclutamiento de proteínas coactivadoras. A continuación, se reclutan proteínas adicionales, incluida la ARN polimerasa, al complejo NR/ADN que transcribe el ADN en ARN mensajero.
Los receptores nucleares de tipo II incluyen principalmente la subfamilia 1, por ejemplo, el receptor de ácido retinoico, el receptor X de retinoide y el receptor de la hormona tiroidea.
Tipo III
Los receptores nucleares de tipo III (principalmente la subfamilia 2 de NR) son similares a los receptores de tipo I en que ambas clases se unen al ADN como homodímeros. Sin embargo, los receptores nucleares de tipo III, a diferencia del tipo I, se unen a los receptores de repetición directa en lugar de a los de repetición invertida.
Tipo IV
Los receptores nucleares de tipo IV se unen como monómeros o dímeros, pero sólo un único dominio de unión al ADN del receptor se une a un único medio sitio HRE. Se encuentran ejemplos de receptores de tipo IV en la mayoría de las subfamilias NR.
Dimerization
Los receptores nucleares humanos son capaces de dimerizarse con muchos otros receptores nucleares (dimerización homotípica), como se ha demostrado a partir de experimentos a gran escala con Y2H y de esfuerzos de minería de textos de la literatura que se centraron en interacciones específicas. Sin embargo, existe especificidad, ya que los miembros de la misma subfamilia tienen socios de dimerización de NR muy similares y la red de dimerización subyacente tiene ciertas características topológicas, como la presencia de centros altamente conectados (RXR y SHP).
Proteínas coregulatorias
Los receptores nucleares unidos a elementos de respuesta hormonal reclutan una cantidad significativa de otras proteínas (denominadas correguladores de la transcripción) que facilitan o inhiben la transcripción del gen diana asociado en ARNm. La función de estos correguladores es variada e incluye la remodelación de la cromatina (haciendo que el gen diana sea más o menos accesible a la transcripción) o una función de puente para estabilizar la unión de otras proteínas correguladoras. Los receptores nucleares pueden unirse específicamente a una cantidad de proteínas correguladoras y, por lo tanto, influir en los mecanismos celulares de transducción de señales tanto de manera directa como indirecta.
Coactivadores
La unión de ligandos agonistas (ver sección a continuación) a receptores nucleares induce una conformación del receptor que se une preferentemente a proteínas coactivadoras. Estas proteínas a menudo tienen una actividad intrínseca de histona acetiltransferasa (HAT), que debilita la asociación de las histonas al ADN y, por lo tanto, promueve la transcripción génica.
Corepressors
En cambio, la unión de ligandos antagonistas a receptores nucleares induce una conformación del receptor que se une preferentemente a proteínas correpresoras. Estas proteínas, a su vez, reclutan histonas desacetilasas (HDAC), que fortalecen la asociación de las histonas al ADN y, por lo tanto, reprimen la transcripción génica.
Agonismo vs antagonismo
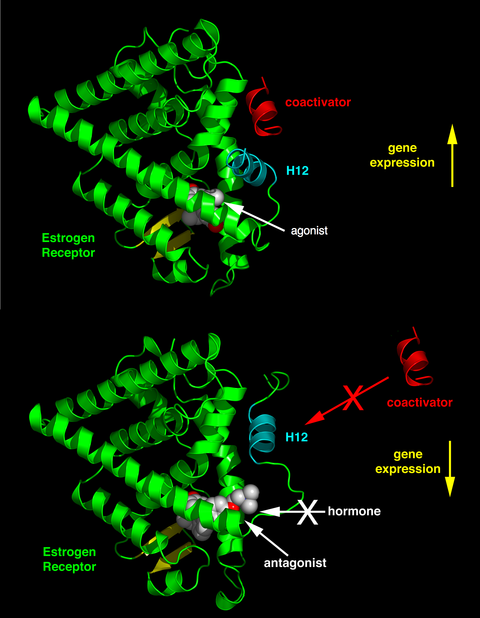
Dependiendo del receptor involucrado, la estructura química del ligando y el tejido afectado, los ligandos de los receptores nucleares pueden mostrar efectos muy diversos que van desde el agonismo al antagonismo y al agonismo inverso.
Agonistas
La actividad de los ligandos endógenos (como las hormonas estradiol y testosterona) cuando se unen a sus receptores nucleares afines es normalmente la de aumentar la expresión génica. Esta estimulación de la expresión génica por el ligando se conoce como respuesta agonista. Los efectos agonistas de las hormonas endógenas también pueden ser imitados por ciertos ligandos sintéticos, por ejemplo, el fármaco antiinflamatorio del receptor de glucocorticoides dexametasona. Los ligandos agonistas funcionan induciendo una conformación del receptor que favorece la unión del coactivador (véase la mitad superior de la figura a la derecha).
Antagonistas
Otros ligandos sintéticos de receptores nucleares no tienen un efecto aparente sobre la transcripción génica en ausencia de ligando endógeno. Sin embargo, bloquean el efecto del agonista mediante la unión competitiva al mismo sitio de unión en el receptor nuclear. Estos ligandos se denominan antagonistas. Un ejemplo de fármaco antagonista de receptores nucleares es la mifepristona, que se une a los receptores de glucocorticoides y progesterona y, por lo tanto, bloquea la actividad de las hormonas endógenas cortisol y progesterona respectivamente. Los ligandos antagonistas funcionan induciendo una conformación del receptor que evita la unión del coactivador y promueve la unión del correpresor (véase la mitad inferior de la figura a la derecha).
Agonistas inversos
Por último, algunos receptores nucleares promueven un nivel bajo de transcripción génica en ausencia de agonistas (también denominada actividad basal o constitutiva). Los ligandos sintéticos que reducen este nivel basal de actividad en los receptores nucleares se conocen como agonistas inversos.
Moduladores selectivos de receptores
Varios fármacos que actúan a través de receptores nucleares muestran una respuesta agonista en algunos tejidos y una respuesta antagonista en otros. Este comportamiento puede tener beneficios sustanciales, ya que puede permitir retener los efectos terapéuticos beneficiosos deseados de un fármaco mientras se minimizan los efectos secundarios indeseables. Los fármacos con este perfil de acción agonista/antagonista mixto se denominan moduladores selectivos de receptores (SRM). Algunos ejemplos incluyen moduladores selectivos de receptores de andrógenos (SARM), moduladores selectivos de receptores de estrógenos (SERM) y moduladores selectivos de receptores de progesterona (SPRM). El mecanismo de acción de los SRM puede variar según la estructura química del ligando y el receptor involucrado, sin embargo, se cree que muchos SRM funcionan promoviendo una conformación del receptor que está estrechamente equilibrada entre agonismo y antagonismo. En los tejidos donde la concentración de proteínas coactivadoras es mayor que la de los correpresores, el equilibrio se desplaza en la dirección del agonista. Por el contrario, en los tejidos donde predominan los correpresores, el ligando se comporta como un antagonista.
Mecanismos alternativos

Transrepresión
El mecanismo más común de acción de los receptores nucleares implica la unión directa del receptor nuclear a un elemento de respuesta hormonal del ADN. Este mecanismo se denomina transactivación. Sin embargo, algunos receptores nucleares no sólo tienen la capacidad de unirse directamente al ADN, sino también a otros factores de transcripción. Esta unión a menudo da como resultado la desactivación del segundo factor de transcripción en un proceso conocido como transrepresión. Un ejemplo de un receptor nuclear que puede transreprimir es el receptor de glucocorticoides (GR). Además, ciertos ligandos de GR conocidos como agonistas selectivos del receptor de glucocorticoides (SEGRA) pueden activar GR de tal manera que GR transreprime más fuertemente que transactiva. Esta selectividad aumenta la separación entre los efectos antiinflamatorios deseados y los efectos secundarios metabólicos no deseados de estos glucocorticoides selectivos.
No genómica
Los efectos directos clásicos de los receptores nucleares sobre la regulación génica normalmente tardan horas en observarse en las células debido a la gran cantidad de pasos intermedios entre la activación del receptor nuclear y los cambios en los niveles de expresión de proteínas. Sin embargo, se ha observado que muchos efectos de la aplicación de hormonas nucleares, como los cambios en la actividad de los canales iónicos, se producen en cuestión de minutos, lo que es incompatible con el mecanismo clásico de acción de los receptores nucleares. Si bien no se ha demostrado de manera concluyente el objetivo molecular de estos efectos no genómicos de los receptores nucleares, se ha planteado la hipótesis de que existen variantes de receptores nucleares que están asociados a la membrana en lugar de estar localizados en el citosol o el núcleo. Además, estos receptores asociados a la membrana funcionan a través de mecanismos alternativos de transducción de señales que no implican la regulación génica.
Si bien se ha planteado la hipótesis de que existen varios receptores asociados a la membrana para las hormonas nucleares, se ha demostrado que muchos de los efectos rápidos requieren receptores nucleares canónicos. Sin embargo, la prueba de la importancia relativa de los mecanismos genómicos y no genómicos in vivo se ha visto impedida por la ausencia de mecanismos moleculares específicos para los efectos no genómicos que podrían bloquearse mediante la mutación del receptor sin alterar sus efectos directos sobre la expresión génica.
Un mecanismo molecular para la señalización no genómica a través del receptor nuclear de la hormona tiroidea TRβ involucra a la fosfatidilinositol 3-quinasa (PI3K). Esta señalización puede ser bloqueada por una sola sustitución de tirosina a fenilalanina en TRβ sin alterar la regulación génica directa. Cuando se crearon ratones con esta única sustitución conservadora de aminoácidos en TRβ, la maduración sináptica y la plasticidad en el hipocampo se vieron afectadas casi tan efectivamente como el bloqueo total de la síntesis de la hormona tiroidea. Este mecanismo parece estar conservado en todos los mamíferos, pero no en TRα ni en ningún otro receptor nuclear. Por lo tanto, la asociación dependiente de la fosfatirosina de TRβ con PI3K proporciona un mecanismo potencial para integrar la regulación del desarrollo y el metabolismo por la hormona tiroidea y las tirosina quinasas del receptor. Además, la señalización de la hormona tiroidea a través de PI3K puede alterar la expresión génica.
Miembros de familia
La siguiente es una lista de los 48 receptores nucleares humanos conocidos (y sus ortólogos en otras especies) clasificados según la homología de secuencia. La lista también incluye miembros seleccionados de la familia que carecen de ortólogos humanos (símbolo NRNC resaltado en amarillo).
| Subfamilia | Grupo | Miembro | ||||||
|---|---|---|---|---|---|---|---|---|
| Signatura NRNC | Abreviatura | Nombre | Gene | Ligand(s) | ||||
| 1 | Receptor de hormona tiroidea | A | Receptor de hormona tiroidea | NR1A1 | TR | Receptor de hormona tiroidea-α | THRA | hormona tiroidea |
| NR1A2 | TRβ | Receptor de hormona tiroidea-β | THRB | |||||
| B | Receptor de ácido retinoico | NR1B1 | RARα | Receptor ácido retinoico-α | RARA | vitamina A y compuestos conexos | ||
| NR1B2 | RARβ | Receptor de ácido retinoico-β | RARB | |||||
| NR1B3 | RARγ | Receptor de ácido retinoico-γ | RARG | |||||
| C | Receptor activado por proliferator peroxisome | NR1C1 | PPARα | Peroxisome proliferator-activado receptor-α | PPARA | ácidos grasos, prostaglandinas | ||
| NR1C2 | PPAR-β/δ | Peroxisome proliferator-activado receptor-β/δ | PPARD | |||||
| NR1C3 | PPARγ | Peroxisome proliferator-activado receptor-γ | PPARG | |||||
| D | Rev-ErbA | NR1D1 | Rev-ErbAα | Rev-ErbAα | NR1D1 | heme | ||
| NR1D2 | Rev-ErbAβ | Rev-ErbAα | NR1D2 | |||||
| E | E78C-like (artropo, trematodo, mullosc, nematodo) | NR1E1 | Eip78C | Proteína inducida por Ecdysone 78C | Eip78C | |||
| F | Receptor huérfano relacionado con RAR | NR1F1 | RαOR | Receptor huérfano relacionado con RAR-α | RORA | colesterol, ATRA | ||
| NR1F2 | RORβ | Receptor huérfano relacionado con RAR-β | RORB | |||||
| NR1F3 | RORγ | Receptor huérfano relacionado con RAR-γ | RORC | |||||
| G | CNR14-como (nematodo) | NR1G1 | sexo-1 | Receptor de hormona esteroides cnr14 | sexo-1 | |||
| H | Receptor de Liver X | NR1H1 | EcR | Receptor de Ecdysone, EcR (artropod) | EcR | ecdysteroids | ||
| NR1H2 | LXRβ | Receptor vivo X-β | NR1H2 | oxysterols | ||||
| NR1H3 | LXRα | Receptor vivo X-α | NR1H3 | |||||
| NR1H4 | FXR | Farnesoid Receptor X | NR1H4 | |||||
| NR1H5 | FXR-β | Receptor Farnesoid X-β (pseudogene in human) | NR1H5P | |||||
| I | Vitamina D como receptor | NR1I1 | VDR | Vitamina Receptor D | VDR | vitamina D | ||
| NR1I2 | PXR | Embarazo Receptor X | NR1I2 | xenobióticos | ||||
| NR1I3 | CAR | Receptor constitutivo yrostano | NR1I3 | androstane | ||||
| J | Hr96-como | NR1J1 | Hr96/Daf-12 | Receptor de hormona nuclear HR96 | Hr96 | colesterol/ácido dáfacrónico | ||
| NR1J2 | ||||||||
| NR1J3 | ||||||||
| K | Hr1-como | NR1K1 | Hr1 | Receptor de hormona nuclear HR1 | ||||
| 2 | Receptor Retinoid X | A | Factor nuclear hepatocito-4 | NR2A1 | HNF4α | Factor nuclear hepatocito-4-α | HNF4A | ácidos grasos |
| NR2A2 | HNF4γ | Factor nuclear hepatocito-4-γ | HNF4G | |||||
| B | Receptor Retinoide X | NR2B1 | RXRα | Receptor retinoide X-α | RXRA | retinoides | ||
| NR2B2 | RXRβ | Receptor retinoide X-β | RXRB | |||||
| NR2B3 | RXRγ | Retinoide X receptor-γ | RXRG | |||||
| NR2B4 | USP | Proteína ultraspirálica (artropodo) | usp | fosfolípidos | ||||
| C | Receptor testicular | NR2C1 | TR2 | Receptor testicular 2 | NR2C1 | |||
| NR2C2 | TR4 | Receptor testicular 4 | NR2C2 | |||||
| E | TLX/PNR | NR2E1 | TLX | Homologo del gen sin cola de Drosophila | NR2E1 | |||
| NR2E3 | PNR | Receptor nuclear específico de células fotorreceptoras | NR2E3 | |||||
| F | COUP/EAR | NR2F1 | COUP-TFI | Chicken ovalbumin upstream factor promotor-transcripción I | NR2F1 | |||
| NR2F2 | COUP-TFII | Chicken ovalbumin upstream promotor-transcription factor II | NR2F2 | ácido retinoico (modo) | ||||
| NR2F6 | EAR-2 | V-erbA-related | NR2F6 | |||||
| 3 | Estrógeno Receptor-like | A | Receptor de estrógeno | NR3A1 | ERα | Receptor estrógeno-α | ESR1 | estrógenos |
| NR3A2 | ERβ | Receptor de estrógeno-β | ESR2 | |||||
| B | Receptor relacionado con el estrógeno | NR3B1 | ERRα | Receptor-α relacionado con el estrógeno | ESRRA | |||
| NR3B2 | ERRβ | Receptor relacionado con el estrógeno-β | ESRRB | |||||
| NR3B3 | ERRγ | Receptor relacionado con el estrógeno-γ | ESRRG | |||||
| C | Receptores de 3-Ketosteroid | NR3C1 | GR | Receptor glucocorticoide | NR3C1 | cortisol | ||
| NR3C2 | MR | Receptor mineralocorticoide | NR3C2 | aldosterona | ||||
| NR3C3 | PR | Receptor de progesterona | PGR | progesterona | ||||
| NR3C4 | AR | Receptor de andrógeno | AR | testosterona | ||||
| D | Estrógeno Receptor-like (en lophotrochozoa) | NR3D | ||||||
| E | Estrógeno Receptor-like (en cnidaria) | NR3E | ||||||
| F | Estrógeno Receptor-like (en placozoa) | NR3F | ||||||
| 4 | Factor de crecimiento nervioso similar a IB | A | NGFIB/NURR1/NOR1 | NR4A1 | NGFIB | Factor de crecimiento nervioso IB | NR4A1 | |
| NR4A2 | NURR1 | Receptor nuclear relacionado 1 | NR4A2 | |||||
| NR4A3 | NOR1 | Receptor huérfano de origen neurono 1 | NR4A3 | |||||
| 5 | Esteroideogénico Factor-como | A | SF1/LRH1 | NR5A1 | SF1 | Factor esteroideo 1 | NR5A1 | fosfatidylinositoles |
| NR5A2 | LRH-1 | Receptor del hígado homolog-1 | NR5A2 | fosfatidylinositoles | ||||
| B | Hr39-like | NR5B1 | HR39/FTZ-F1 | Receptor de hormona nuclear fushi tarazu factor I beta | Hr39 | |||
| 6 | Germ Cell Nuclear Factor-like | A | GCNF | NR6A1 | GCNF | Factor nuclear de la célula germinal | NR6A1 | |
| 7 | NRs con dos dominios vinculantes de ADN | A | 2DBD-NRα | NR7A1 | 2DBD-NRA2 | |||
| B | 2DBD-NRβ | NR7B1 | 2DBD-NRA3 | |||||
| C | 2DBD-NRγ | NR7C1 | 2DBD-NRA1 | artrópodo "α/β" | ||||
| 8 | NR8 (eumetazoa) | A | NR8A | NR8A1 | CgNR8A1 | Receptor nuclear 8 | AKG49571 | |
| 0 | Misceláneas (lacks ya sea LBD o DBD) | A | knr/knrl/egon (artropods) | NR0A1 | KNI | Knirps de proteína de la brecha | knl | |
| B | DAX/SHP | NR0B1 | DAX1 | Reversión sexual sensible a la dosis, región crítica de hipoplasia suprarrenal, en el cromosoma X, gen 1 | NR0B1 | |||
| NR0B2 | SHP | Pequeño socio heterodimer | NR0B2 | |||||
De las dos familias 0, la 0A tiene un DBD similar al de la familia 1 y la 0B tiene un LBD único. El segundo DBD de la familia 7 probablemente esté relacionado con el DBD de la familia 1. Tres NR probablemente de la familia 1 de Biomphalaria glabrata poseen un DBD junto con un LBD similar al de la familia 0B. La ubicación del nhr-1 de C. elegans (Q21878) es objeto de controversia: aunque la mayoría de las fuentes lo ubican como NR1K1, la anotación manual en WormBase lo considera un miembro de NR2A. Solía haber un grupo 2D cuyo único miembro era Drosophila HR78/NR1D1 (Q24142) y ortólogos, pero luego se fusionó con el grupo 2C debido a su gran similitud, formando un "grupo 2C/D". Los estudios de eliminación de genes en ratones y moscas de la fruta respaldan la existencia de un grupo fusionado de este tipo.
Evolución
Un tema de debate ha sido la identidad del receptor nuclear ancestral como receptor huérfano o ligante de ligando. Este debate comenzó hace más de veinticinco años cuando se identificaron los primeros ligandos como hormonas tiroideas y esteroides de mamíferos. Poco después, la identificación del receptor de ecdisona en Drosophila introdujo la idea de que los receptores nucleares eran receptores hormonales que se unen a ligandos con una afinidad nanomolar. En ese momento, los tres ligandos de receptores nucleares conocidos eran esteroides, retinoides y hormona tiroidea, y de esos tres, tanto los esteroides como los retinoides eran productos del metabolismo de los terpenoides. Por lo tanto, se postuló que el receptor ancestral habría sido ligado por una molécula de terpenoide.
En 1992, una comparación del dominio de unión al ADN de todos los receptores nucleares conocidos condujo a la construcción de un árbol filogenético de los receptores nucleares que indicó que todos los receptores nucleares compartían un ancestro común. Como resultado, hubo un mayor esfuerzo por descubrir el estado del primer receptor nuclear y en 1997 se sugirió una hipótesis alternativa: el receptor nuclear ancestral era un receptor huérfano y adquirió la capacidad de unirse a ligandos con el tiempo. Esta hipótesis se propuso con base en los siguientes argumentos:
- Las secuencias de receptores nucleares que se habían identificado en los primeros metazoos (cnidarios y Schistosoma) todos los miembros de los grupos de receptores COUP-TF, RXR y FTZ-F1. Tanto COUP-TF como FTZ-F1 son receptores huérfanos, y RXR sólo se encuentra para atar un ligando en vertebrados.
- Mientras que los receptores huérfanos habían conocido homólogos artrópodos, no se habían identificado ortodoxos de receptores vertebrados ligados fuera de los vertebrados, lo que sugiere que los receptores huérfanos son mayores que los receptores liganded.
- Los receptores huérfanos se encuentran entre las seis subfamilias de los receptores nucleares, mientras que los receptores dependientes de ligando se encuentran entre tres. Así pues, dado que se creía que los receptores dependientes de los ligandos eran predominantemente miembros de las subfamilias recientes, parecía lógico que adquirieran la capacidad de ligands de forma independiente.
- La posición filogenética de un receptor nuclear dado dentro del árbol se correlaciona con sus capacidades de unión de ADN y de dimerización, pero no hay una relación identificada entre un receptor nuclear dependiente de ligando y la naturaleza química de su ligando. Además de esto, las relaciones evolutivas entre los receptores dependientes de los ligandos no tenían mucho sentido como receptores estrechamente relacionados de ligandos ligandos ligados a las subfamilias originarios de caminos biosintéticos completamente diferentes (por ejemplo, TRs y RARs). Por otro lado, las subfamilias que no están relacionadas evolucionariamente unen ligands similares (RAR y RXR ambos unen todo-trans y 9-cis ácido retinoico respectivamente).
- En 1997, se descubrió que los receptores nucleares no existían en la estática y en las conformaciones, pero que un ligando podría alterar el equilibrio entre los dos estados. Además, se encontró que los receptores nucleares podían ser regulados de manera dependiente del ligand, ya sea mediante la fosforilación u otras modificaciones post-translacionales. Así, esto proporcionó un mecanismo para cómo un receptor huérfano ancestral fue regulado de manera dependiente del ligand, y explicó por qué se conservaba el dominio de unión del ligand.
Durante los siguientes 10 años, se llevaron a cabo experimentos para probar esta hipótesis y pronto surgieron contraargumentos:
- Los receptores nucleares fueron identificados en el nuevo genoma secuenciado de la demosponja Amphimedon queenslandica, un miembro Porifera, el phylum metazoano más antiguo. El A. queenslandica genoma contiene dos receptores nucleares conocidos como AqNR1 y AqNR2 y ambos se caracterizaron por atar y ser regulados por ligands.
- Homologs for ligand-dependiente vertebrate receives were found outside vertebrates in mollusks and Platyhelminthes. Además, se encontró que los receptores nucleares encontrados en cnidarios tenían ligandos estructurales en mamíferos, que podían reflejar la situación ancestral.
- Se encontraron dos receptores orfanatos putantes, HNF4 y USP, mediante análisis estructural y masivo de espectrometría, para unir ácidos grasos y fosfolípidos respectivamente.
- Los receptores y ligandos nucleares se encuentran mucho menos específicos de lo que se pensaba anteriormente. Los retinoides pueden atar a los receptores mamíferos distintos de RAR y RXR, como PPAR, RORb o COUP-TFII. Además, RXR es sensible a una amplia gama de moléculas incluyendo retinoides, ácidos grasos y fosfolípidos.
- Estudio de la evolución de los receptores de esteroides reveló que el receptor de esteroides ancestrales podría atar un ligand, estradiol. Por el contrario, el receptor de estrógeno que se encuentra en moluscos es constitutivamente activo y no ató hormonas relacionadas con el estrógeno. Así, esto proporcionó un ejemplo de cómo un receptor dependiente de ligand ancestral podría perder su capacidad de ligando ligandos.
Una combinación de esta evidencia reciente, así como un estudio profundo de la estructura física del dominio de unión del ligando del receptor nuclear ha llevado al surgimiento de una nueva hipótesis sobre el estado ancestral del receptor nuclear. Esta hipótesis sugiere que el receptor ancestral puede actuar como un sensor de lípidos con capacidad para unirse, aunque de manera bastante débil, a varias moléculas hidrofóbicas diferentes, como retinoides, esteroides, hemo y ácidos grasos. Con su capacidad para interactuar con una variedad de compuestos, este receptor, a través de duplicaciones, perdería su capacidad para la actividad dependiente del ligando o se especializaría en un receptor altamente específico para una molécula en particular.
Historia
A continuación se presenta una breve selección de eventos clave en la historia de la investigación sobre receptores nucleares.
- 1905 – Ernest Starling acuñó la palabra hormona
- 1926 – Edward Calvin Kendall y Tadeus Reichstein aislaron y determinaron las estructuras de cortisona y tiroxina
- 1929 – Adolf Butenandt y Edward Adelbert Doisy – aislados independientemente y determinado la estructura del estrógeno
- 1958 – Elwood Jensen – aislado el receptor de estrógeno
- 1980 – clonación de los receptores de hormonas estrógeno, glucocorticoides y tiroides por Pierre Chambon, Ronald Evans y Björn Vennström respectivamente
- 2004 – Pierre Chambon, Ronald Evans y Elwood Jensen fueron galardonados con el Premio Albert Lasker de Investigación Médica Básica, un premio que frecuentemente precede a un Premio Nobel de Medicina
Véase también
- NucleaRDB
- Receptor de hormona tiroidea
- Receptor de hormona esteroides
- Receptores
Referencias
- ^ a b PDB: 3E00; Chandra V, Huang P, Hamuro Y, Raghuram S, Wang Y, Burris TP, Rastinejad F (noviembre de 2008). "Strutura del complejo nuclear de receptores PPAR-gamma-RXR- intacto en el ADN". Naturaleza. 456 (7220): 350–6. doi:10.1038/nature07413. PMC 2743566. PMID 19043829.
- ^ Evans RM (mayo de 1988). "El esteroide y la hormona tiroidea receptores superfamilia". Ciencia. 240 (4854): 889–95. Código:1988Sci...240..889E. doi:10.1126/ciencia.3283939. PMC 6159881. PMID 3283939.
- ^ Olefsky JM (octubre de 2001). "Nuclear receptor minireview series". El Diario de Química Biológica. 276 (40): 36863-4. doi:10.1074/jbc.R100047200PMID 11459855. S2CID 5497175.
- ^ a b c Mangelsdorf DJ, Thummel C, Beato M, Herrlich P, Schütz G, Umesono K, Blumberg B, Kastner P, Mark M, Chambon P, Evans RM (diciembre de 1995). "La superfamilia del receptor nuclear: la segunda década". Celular. 83 (6): 835-9. doi:10.1016/0092-8674(95)90199-X. PMC 6159888. PMID 8521507.
- ^ a b Novac N, Heinzel T (diciembre de 2004). "Receptores núcleos: panorama y clasificación". Metas de drogas actuales. Inflamación y alergia. 3 (4): 335–46. doi:10.2174/1568010042634541. PMID 15584884.
- ^ a b c Comité de Nomenclatura de Receptores Nucleares (abril de 1999). "Un sistema unificado de nomenclatura para la superfamilia del receptor nuclear". Celular. 97 (2): 161–3. doi:10.1016/S0092-8674(00)80726-6. PMID 10219237. S2CID 36659104.
- ^ a b c d Laudet V (diciembre de 1997). "Evolución de la superfamilia del receptor nuclear: diversificación temprana de un receptor huérfano ancestral". Journal of Molecular Endocrinology. 19 (3): 207–26. doi:10.1677/jme.0.0190207. PMID 9460643. S2CID 16419929.
- ^ Escriva H, Langlois MC, Mendonça RL, Pierce R, Laudet V (mayo de 1998). "Evolución y diversificación de la superfamilia del receptor nuclear". Annals of the New York Academy of Sciences. 839 (1): 143–6. Bibcode:1998NYASA.839..143E. doi:10.1111/j.1749-6632.1998.tb10747.x. PMID 9629140. S2CID 11164838.
- ^ Reitzel AM, Pang K, Ryan JF, Mullikin JC, Martindale MQ, Baxevanis AD, Tarrant AM (Febrero 2011). "Los receptores núcleos de la ctenofora Mnemiopsis leidyi carecen de un dominio de unión de ADN de zinc-finger: pérdida específica de linaje o condición ancestral en el surgimiento de la superfamilia del receptor nuclear?". EvoDevo. 2 (1): 3. doi:10.1186/2041-9139-2-3. PMC 3038971. PMID 21291545.
- ^ Bridgham JT, Eick GN, Larroux C, Deshpande K, Harms MJ, Gauthier ME, Ortlund EA, Degnan BM, Thornton JW (octubre de 2010). "Evolución de proteínas por tinkering molecular: diversificación de la superfamilia del receptor nuclear de un ancestro dependiente del ligand". PLOS Biología. 8 (10): e1000497. doi:10.1371/journal.pbio.1000497. PMC 2950128. PMID 20957188.
- ^ Sluder AE, Maina CV (abril de 2001). "Receptores núcleos en nematodos: temas y variaciones". Tendencias en la genética. 17 (4): 206–13. doi:10.1016/S0168-9525(01)02242-9. PMID 11275326.
- ^ Cheatle Jarvela AM, Pick L (2017). "La función y la evolución de los receptores nucleares en el desarrollo embrionario de insectos". Temas actuales en Developmental Biología. 125: 39–70. doi:10.1016/bs.ctdb.2017.01.003. ISBN 9780128021729. PMID 28527580.
- ^ Schaaf MJ (2017). "Investigación de receptores núcleos en peces cebra". Journal of Molecular Endocrinology. 59 (1): R65-R76. doi:10.1530/JME-17-0031. PMID 28438785.
- ^ a b Zhang Z, Burch PE, Cooney AJ, Lanz RB, Pereira FA, Wu J, Gibbs RA, Weinstock G, Wheeler DA (abril de 2004). "Análisis genómico de la familia de receptores nucleares: nuevas ideas sobre la estructura, regulación y evolución del genoma de ratas". Genome Research. 14 (4): 580-90. doi:10.1101/gr.2160004. PMC 383302. PMID 15059999.
- ^ Overington JP, Al-Lazikani B, Hopkins AL (diciembre de 2006). "¿Cuántos objetivos de drogas hay?". Reseñas de la naturaleza. Drug Discovery. 5 (12): 993–6. doi:10.1038/nrd2199. PMID 17139284. S2CID 11979420.
- ^ Benoit G, Cooney A, Giguere V, Ingraham H, Lazar M, Muscat G, Perlmann T, Renaud JP, Schwabe J, Sladek F, Tsai MJ, Laudet V (diciembre de 2006). "Unión Internacional de Farmacología. LXVI. Receptores nucleares huérfanos". Pharmacological Reseñas. 58 (4): 798-836. doi:10.1124/pr.58.4.10. PMID 17132856. S2CID 2619263.
- ^ Mohan R, Heyman RA (2003). "Moduladores de receptores nucleares huérfanos". Temas actuales en Química Medicinal. 3 (14): 1637–47. doi:10.2174/1568026033451709. PMID 14683519.
- ^ Kumar R, Thompson EB (mayo de 1999). "La estructura de los receptores de hormonas nucleares". esteroides. 64 (5): 310–9. doi:10.1016/S0039-128X(99)00014-8. PMID 10406480. S2CID 18333397.
- ^ Klinge CM (mayo de 2000). "Interacción del receptor estrógeno con co-activadores y co-represores". esteroides. 65 (5): 227–51. doi:10.1016/S0039-128X(99)00107-5. PMID 10751636. S2CID 41160722.
- ^ a b Wärnmark A, Treuter E, Wright AP, Gustafsson JA (octubre de 2003). "Activación funciona 1 y 2 de los receptores nucleares: estrategias moleculares para la activación transcripcional". Endocrinología molecular. 17 (10): 1901–9. doi:10.1210/me.2002-0384. PMID 12893880. S2CID 31314461.
- ^ Wu W, LoVerde PT (2021). "Identificación y evolución de los receptores nucleares en Platyhelminths". PLOS ONE. 16(8): e0250750 (8): e0250750. Bibcode:2021PLoSO..1650750W. doi:10.1371/journal.pone.0250750. PMC 8363021. PMID 34388160.
- ^ Weatherman RV, Fletterick RJ, Scanlan TS (1999). "Los ligandos de receptor núcleo y los dominios de unión de ligando". Examen anual de la bioquímica. 68: 559–81. doi:10.1146/annurev.biochem.68.1.559. PMID 10872460.
- ^ Chandra V, Huang P, Potluri N, Wu D, Kim Y, Rastinejad F (marzo de 2013). "Intección de Multidomain en la estructura del complejo nuclear de receptores HNF-4α". Naturaleza. 495 (7441): 394–8. Código:2013Natur.495..394C. doi:10.1038/nature11966. PMC 3606643. PMID 23485969.
- ^ Lou X, Toresson G, Benod C, Suh JH, Philips KJ, Webb P, Gustafsson JA (marzo de 2014). "Strutura del heterodimer retinoide receptor X α-liver X β (RXRα-LXRβ) en ADN". Naturaleza Estructural & Molecular Biología. 21 (3): 277–81. doi:10.1038/nsmb.2778. PMID 24561505. S2CID 23226682.
- ^ PDB: 2C7A; Roemer SC, Donham DC, Sherman L, Pon VH, Edwards DP, Churchill ME (diciembre de 2006). "Strutura del complejo ácido progesterona-deoxiribonucleico del receptor progesterona: interacciones novedosas necesarias para la unión a elementos de respuesta medio sitio". Endocrinología molecular. 20 (12): 3042–52. doi:10.1210/me.2005-0511. PMC 2532839. PMID 16931575.
- ^ PDB: 3L0L; Jin L, Martynowski D, Zheng S, Wada T, Xie W, Li Y (mayo de 2010). "Base estructural para los hidroxicolesteroles como ligandos naturales del receptor nuclear huérfano RORgamma". Endocrinología molecular. 24 (5): 923-9. doi:10.1210/me.2009-0507. PMC 2870936. PMID 20203100.
- ^ a b c d Amoutzias GD, Pichler EE, Mian N, De Graaf D, Imsiridou A, Robinson-Rechavi M, Bornberg-Bauer E, Robertson DL, Oliver SG (Julio 2007). "Un atlas de interacción de proteínas para los receptores nucleares: propiedades y calidad de una red de dimerización basada en centros". BMC Systems Biology. 1: 34. doi10.1186/1752-0509-1-34. PMC 1971058. PMID 17672894.
- ^ Linja MJ, Porkka KP, Kang Z, Savinainen KJ, Jänne OA, Tammela TL, Vessella RL, Palvimo JJ, Visakorpi T (febrero de 2004). "Expresión de coronadores de receptores de andrógeno en cáncer de próstata". Clinical Cancer Research. 10 (3): 1032–40. doi:10.1158/1078-0432.CCR-0990-3. PMID 14871982. S2CID 8038717.
- ^ Klinge CM, Bodenner DL, Desai D, Niles RM, Traish AM (mayo de 1997). "Binding of type II nuclear receives and estrogen recipient to full and half-site estrogen response elements in vitro". Nucleic Acids Research. 25 (10): 1903–12. doi:10.1093/nar/25.10.1903. PMC 146682. PMID 9115356.
- ^ Rual, Jean-François; Venkatesan, Kavitha; Hao, Tong; Hirozane-Kishikawa, Tomoko; Dricot, Amélie; Li, Ning; Berriz, Gabriel F.; Gibbons, Francis D.; Dreze, Matija; Ayivi-Guedehoussou, Nono; Klitgord, Niels (2005-10). "Hacia un mapa a escala proteome de la red de interacción de proteínas humanas". Naturaleza. 437 (7062): 1173–1178. Código:2005Natur.437.1173R. doi:10.1038/nature04209. ISSN 1476-4687. S2CID 4427026.
- ^ Albers, Michael; Kranz, Harald; Kober, Ingo; Kaiser, Carmen; Klink, Martin; Suckow, Jörg; Kern, Rainer; Koegl, Manfred (febrero de 2005). "Espección automatizada de dos hibridos de levadura para proteínas de intervención de receptores nucleares". Molecular & Celular Proteomics. 4 (2): 205–213. doi:10.1074/mcp.M400169-MCP200. ISSN 1535-9476. PMID 15604093. S2CID 14876486.
- ^ McKenna NJ, Lanz RB, O'Malley BW (junio de 1999). "Nuclear receptor coregulators: cellular and molecular biology". Reseñas endocrinas. 20 (3): 321-344. doi:10.1210/edrv.20.3.0366PMID 10368774. S2CID 10182146.
- ^ Glass CK, Rosenfeld MG (enero de 2000). "El intercambio de Coregulator en funciones transcripcionales de receptores nucleares". Genes " Development. 14 (2): 121–41. doi:10.1101/gad.14.2.121. PMID 10652267. S2CID 12793980.
- ^ Aranda A, Pascual A (julio de 2001). "Receptores hormonales núcleos y expresión genética". Fisiológica Reseñas. 81 (3): 1269–304. doi:10.1152/physrev.2001.81.3.1269. hdl:10261/79944. PMID 11427696. S2CID 5972234.
- ^ Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (junio de 2009). "Receptores de esteroides sexuales en diferenciación esquelética y neoplasia epitelial: ¿es posible la intervención específica del tejido?". BioEnsayos. 31 (6): 629–41. doi:10.1002/bies.200800138. PMID 19382224. S2CID 205469320.
- ^ Brzozowski AM, Pike AC, Dauter Z, Hubbard RE, Bonn T, Engström O, Ohman L, Greene GL, Gustafsson JA, Carlquist M (octubre de 1997). "La base molecular del agonismo y el antagonismo en el receptor del estrógeno". Naturaleza. 389 (6652): 753–8. Código:1997Natur.389..753B. doi:10.1038/39645. PMID 9338790. S2CID 4430999.
- ^ Shiau AK, Barstad D, Loria PM, Cheng L, Kushner PJ, Agard DA, Greene GL (diciembre de 1998). "La base estructural del reconocimiento del receptor/coactivador de estrógeno y el antagonismo de esta interacción por tamoxifeno". Celular. 95 (7): 927–37. doi:10.1016/S0092-8674(00)817171-1PMID 9875847. S2CID 10265320.
- ^ Gronemeyer H, Gustafsson JA, Laudet V (noviembre de 2004). "Principios para la modulación de la superfamilia del receptor nuclear". Reseñas de la naturaleza. Drug Discovery. 3 11): 950–64. doi:10.1038/nrd1551. PMID 15520817. S2CID 205475111.
- ^ Busch BB, Stevens WC, Martin R, Ordentlich P, Zhou S, Sapp DW, Horlick RA, Mohan R (noviembre de 2004). "Identificación de un agonista inverso selectivo para el alfa receptor del receptor nuclear huérfano relacionado con el estrógeno". Revista de Química Medicinal. 47 (23): 5593-6. doi:10.1021/jm049334f. PMID 15509154.
- ^ Smith CL, O'Malley BW (febrero de 2004). "Función del coregulator: una clave para entender la especificidad del tejido de los moduladores selectivos del receptor". Reseñas endocrinas. 25 (1): 45–71. doi:10.1210/er.2003-0023. PMID 14769827.
- ^ Pascual G, Glass CK (octubre de 2006). "Receptores núcleos contra inflamación: mecanismos de transrepresión". Tendencias en Endocrinología y Metabolismo. 17 (8): 321–7. doi:10.1016/j.tem.2006.08.005. PMID 16942889. S2CID 19612552.
- ^ Björnström L, Sjöberg M (junio de 2004). "Activación dependiente del receptor estrógeno de AP-1 a través de señalización no genómica". Receptor nuclear. 2 (1): 3. doi:10.1186/1478-1336-2-3. PMC 434532. PMID 15196329.
- ^ Zivadinovic D, Gametchu B, Watson CS (2005). "Los niveles de receptor-alfa de membrana estrógeno en las células de cáncer de mama MCF-7 predicen el cAMP y las respuestas a la proliferación". Investigación del cáncer de mama. 7 (1): R101–12. doi:10.1186/bcr958. PMC 1064104. PMID 15642158.
- ^ Kousteni S, Bellido T, Plotkin LI, O'Brien CA, Bodenner DL, Han L, Han K, DiGregorio GB, Katzenellenbogen JA, Katzenellenbogen BS, Roberson PK, Weinstein RS, Jilka RL, Manolagas SC (marzo de 2001). "Nongenotropic, sex-nonspecific signaling through the estrogen or androgen recipients: dissociation from transcriptional activity". Celular. 104 (5): 719–30. doi:10.1016/S0092-8674(01)00268-9. PMID 11257226. S2CID 10642274.
- ^ Storey NM, Gentile S, Ullah H, Russo A, Muessel M, Erxleben C, Armstrong DL (marzo de 2006). "Rapid señalización en la membrana plasmática por un receptor nuclear para la hormona tiroidea". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 103 (13): 5197–201. código:2006PNAS..103.5197S. doi:10.1073/pnas.0600089103. PMC 1458817. PMID 16549781.
- ^ Storey NM, O'Bryan JP, Armstrong DL (enero 2002). "Rac y Rho median la regulación hormonal opuesta del canal de potasio relacionado con el éter-a-go". Biología actual. 12 (1): 27–33. Bibcode:2002CBio...12...27S. doi:10.1016/S0960-9822(01)00625-X. PMID 11790300. S2CID 8608805.
- ^ a b Martin NP, Marron Fernandez de Velasco E, Mizuno F, Scappini EL, Gloss B, Erxleben C, Williams JG, Stapleton HM, Gentile S, Armstrong DL (septiembre de 2014). "Un mecanismo citoplasmático rápido para la regulación de la cinasa PI3 por el receptor de hormona tiroidea nuclear, TRβ, y evidencia genética por su papel en la maduración de sinapsis hipocampal del ratón in vivo". Endocrinología. 155 (9): 3713–24. doi:10.1210/en.2013-2058. PMC 4138568. PMID 24932806.
- ^ Gilbert ME (enero de 2004). "Alteraciones en transmisión sináptica y plasticidad en la zona CA1 del hipocampo adulto siguiendo hipotiroidismo de desarrollo". Investigación cerebral. Developmental Brain Research. 148 (1): 11–8. doi:10.1016/j.devbrainres.2003.09.018. PMID 14757514.
- ^ Moeller LC, Broecker-Preuss M (agosto 2011). "Regulación transcripcional por acción no clásica de hormona tiroidea". Thyroid Research. 4 (Supl 1): S6. doi:10.1186/1756-6614-4-S1-S6. PMC 3155112. PMID 21835053.
- ^ a b c d e f h i j k Kaur S, Jobling S, Jones CS, Noble LR, Routledge EJ, Lockyer AE (7 abril 2015). "Los receptores nucleares de Biomphalaria glabrata y Lottia gigantea: implicaciones para desarrollar nuevos organismos modelo". PLOS ONE. 10 (4): e0121259. Bibcode:2015PLoSO..1021259K. doi:10.1371/journal.pone.0121259. PMC 4388693. PMID 25849443.
- ^ Burris TP, de Vera IM, Cote I, Flaveny CA, Waninayake US, Chatterjee A, Walker JK, Steinauer N, Zhang J, Coons LA, Korach KS, Cain DW, Hollenberg AN, Webb Murray Stay D, Jetten AM, Edwards DP, Grimm SL, Hartig S, Lange Ohlstein E (ed.). "Unión Internacional de Farmacología Básica y Clínica CXIII: Superfamilia Receptor Nuclear—Actualizar 2023". Pharmacological Reseñas. 75 (6): 1233–1318. doi:10.1124/pharmrev.121.000436. ISSN 0031-6997. PMC 10595025. PMID 37586884.
- ^ Crossgrove K, Laudet V, Maina CV (febrero de 2002). "Dirofilaria immitis codifica Di-nhr-7, un ortodologo putative del gen E78 de Drosophila ecdysone regulado". Parasitología molecular y bioquímica. 119 (2): 169–77. doi:10.1016/s0166-6851(01)00412-1. PMID 11814569.
- ^ "sex-1 (gene)". WormBase: Nematode Information Resource.
- ^ Otte K, Kranz H, Kober I, Thompson P, Hoefer M, Haubold B, Remmel B, Voss H, Kaiser C, Albers M, Cheruvallath Z, Jackson D, Casari G, Koegl M, Päääbo S, Mous J, Kremoser C, Deuschle U (Febrero de 2003). "Identificar la beta del receptor de farnesoide X como un nuevo receptor nuclear de mamíferos lanosterol de detección". Biología molecular y celular. 23 (3): 864–72. doi:10.1128/mcb.23.3.864-872.2003. PMC 140718. PMID 12529392.
- ^ "FlyBase Gene Report: Dmel\Hr96". FlyBase. Retrieved 14 de agosto 2019.
- ^ a b Schwabe JW, Teichmann SA (enero de 2004). "Receptores núcleos: la evolución de la diversidad". Ciencia STKE. 2004 (217): pe4. doi:10.1126/stke.2172004pe4. PMID 14747695. S2CID 20835274.
- ^ Kruse SW, Suino-Powell K, Zhou XE, Kretschman JE, Reynolds R, Vonrhein C, et al. (septiembre de 2008). "Identificación de los receptores nucleares huérfanos de COUP-TFII como receptor reactivado de ácido retinoico". PLOS Biología. 6 (9): e227. doi:10.1371/journal.pbio.0060227. PMC 2535662. PMID 18798693.
- ^ Markov GV, Gutierrez-Mazariegos J, Pitrat D, Billas IM, Bonneton F, Moras D, et al. (marzo de 2017). "Origina de una antigua pareja hormonal/receptor revelada por la resurrección de un estrógeno ancestral". Avances científicos. 3 (3): e1601778. Bibcode:2017SciA....3E1778M. doi:10.1126/sciadv.1601778. PMC 5375646. PMID 28435861.
- ^ a b Khalturin K, Billas I, Chebaro Y, Reitzel AM, Tarrant AM, Laudet V, Markov GV (noviembre 2018). "Receptores NR3E en cnidarios: una nueva familia de parientes de receptores de esteroides extiende los posibles mecanismos para ligando". J Esteroide Biochem Mol Biol. 184: 11–19. doi:10.1016/j.jsbmb.2018.06.014. PMC 6240368. PMID 29940311.
- ^ Wu, W, LoVerde PT (septiembre 2023). "El conocimiento actualizado y la nomenclatura propuesta para los receptores nucleares con dos dominios vinculantes de ADN (2DBD-NRs)". PLOS ONE. 18 (9): e0286107. Bibcode:2023PLoSO..1886107W. doi:10.1371/journal.pone.0286107. PMC 10497141. PMID 37699039.
- ^ Wu W, Niles EG, Hirai H, LoVerde PT (febrero de 2007). "Evolución de una nueva subfamilia de receptores nucleares con miembros que cada uno contiene dos dominios vinculantes de ADN". BMC Evol Biol. 7 27. Bibcode:2007BMCEE...7...27W. doi:10.1186/1471-2148-7-27. PMC 1810520. PMID 17319953.
- ^ Huang W, Xu F, Li J, Li L, Que H, Zhang G (agosto de 2015). "Evolución de una nueva subfamilia de receptores nucleares con énfasis en el miembro del ostra del Pacífico Crassostrea gigas". Gene. 567 (2): 164–72. doi:10.1016/j.gene.2015.04.082. PMID 25956376.
- ^ "nhr-1 (gene)". WormBase: Nematode Information Resource.
- ^ Marxreiter S, Thummel CS (febrero de 2018). "Adulto funciones para el receptor nuclear Drosophila DHR78". Dinámica del desarrollo. 247 (2): 315–322. doi:10.1002/dvdy.24608. PMC 5771960. PMID 29171103.
- ^ Evans RM (mayo de 1988). "El esteroide y la hormona tiroidea receptores superfamilia". Ciencia. 240 (4854): 889–95. Código:1988Sci...240..889E. doi:10.1126/ciencia.3283939. PMC 6159881. PMID 3283939.
- ^ Moore DD (enero de 1990). "Diversidad y unidad en los receptores de hormonas nucleares: una superfamilia de receptores terpenoides". El nuevo biólogo. 2 (1): 100–5. PMID 1964083.
- ^ Laudet V, Hänni C, Coll J, Catzeflis F, Stéhelin D (marzo de 1992). "Evolución de la superfamilia del gen del receptor nuclear". El Diario EMBO. 11 (3): 1003–13. doi:10.1002/j.1460-2075.1992.tb05139.x. PMC 556541. PMID 1312460.
- ^ Escriva H, Safi R, Hänni C, Langlois MC, Saumitou-Laprade P, Stehelin D, Capron A, Pierce R, Laudet V (junio de 1997). "Ligand binding fue adquirida durante la evolución de los receptores nucleares". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 94 13): 6803-8. Código: 1997PNAS...94.6803E. doi:10.1073/pnas.94.13.6803. PMC 21239. PMID 9192646.
- ^ a b c Escriva H, Delaunay F, Laudet V (agosto 2000). "Evolución de los receptores nucleares y vinculantes". BioEnsayos. 22 (8): 717–27. doi:10.1002/1521-1878(200008)22:8 made717::AID-BIES5 confidencial3.0.CO;2-I. PMID 10918302. S2CID 45891497.
- ^ Bridgham JT, Eick GN, Larroux C, Deshpande K, Harms MJ, Gauthier ME, Ortlund EA, Degnan BM, Thornton JW (octubre de 2010). "Evolución de proteínas por tinkering molecular: diversificación de la superfamilia del receptor nuclear de un ancestro dependiente del ligand". PLOS Biología. 8 (10): e1000497. doi:10.1371/journal.pbio.1000497. PMC 2950128. PMID 20957188.
- ^ a b Markov GV, Laudet V (marzo de 2011). "Origina y evolución de la capacidad de unión de ligandos de los receptores nucleares". Endocrinología molecular y celular. Evolución de receptores de hormonas nucleares. 334 (1–2): 21–30. doi:10.1016/j.mce.2010.10.017. PMID 21055443. S2CID 33537979.
- ^ Thornton JW, Need E, Crews D (septiembre de 2003). "Resucitando el receptor ancestral de esteroides: origen antiguo de la señalización de estrógeno". Ciencia. 301 (5640): 1714-7. Código:2003...301.1714T. doi:10.1126/ciencia.1086185. PMID 14500980. S2CID 37628350.
- ^ Tata JR (junio de 2005). "Cien años de hormonas". EMBO Informes. 6 (6): 490–6. doi:10.1038/sj.embor.7400444. PMC 1369102. PMID 15940278.
Enlaces externos
- Receptores nucleares en la Biblioteca Nacional de Medicina de EE.UU.
- Vincent Laudet (2006). "El Compendio IUPHAR de la Farmacología y Clasificación de la Superfamilia del Receptor Nuclear 2006E". Compendio de receptores nucleares. Unión Internacional de Farmacología Básica y Clínica. Archivado desde el original en 2015-04-02. Retrieved 2008-02-21.
- "Nuclear Receptor online diario". Página. publicado por BioMed Central (ya no aceptando presentaciones desde mayo de 2007). Retrieved 2008-02-21.
- "Nuclear Receptor Resource". Universidad Georgetown. Archivado desde el original el 2008-05-11. Retrieved 2008-02-21.
- "Nuclear Receptor Signaling Atlas (Receptores, Coactivadores, Corepressors y Ligands)". El consorcio NURSA. Retrieved 2008-02-21.
un consorcio de investigación y una base de datos financiados por NIH; incluye una revista de acceso abierto PubMed-indexed, Firma del receptor nuclear
- "Nuclear Receptor Resource". Jack Vanden Heuvel. Retrieved 2009-09-21.
