Reacción en cadena de la polimerasa
La reacción en cadena de la polimerasa (PCR) es un método ampliamente utilizado para hacer rápidamente millones o miles de millones de copias (completas o parciales) de una muestra de ADN específica, lo que permite a los científicos tomar una muestra muy pequeña de ADN y amplificarla (o una parte de ella) a una cantidad lo suficientemente grande como para estudiarla en detalle. PCR fue inventado en 1983 por el bioquímico estadounidense Kary Mullis en Cetus Corporation; Mullis y el bioquímico Michael Smith, que habían desarrollado otras formas esenciales de manipular el ADN, recibieron conjuntamente el Premio Nobel de Química en 1993.
La PCR es fundamental para muchos de los procedimientos utilizados en las pruebas e investigaciones genéticas, incluido el análisis de muestras antiguas de ADN y la identificación de agentes infecciosos. Usando PCR, las copias de cantidades muy pequeñas de secuencias de ADN se amplifican exponencialmente en una serie de ciclos de cambios de temperatura. La PCR es ahora una técnica común y, a menudo, indispensable que se utiliza en la investigación de laboratorio médico para una amplia variedad de aplicaciones, incluida la investigación biomédica y la ciencia forense criminal.
La mayoría de los métodos de PCR se basan en ciclos térmicos. El ciclo térmico expone a los reactivos a ciclos repetidos de calentamiento y enfriamiento para permitir diferentes reacciones dependientes de la temperatura, específicamente, la fusión del ADN y la replicación del ADN impulsada por enzimas. La PCR emplea dos reactivos principales: cebadores (que son fragmentos cortos de ADN de cadena sencilla conocidos como oligonucleótidos que son una secuencia complementaria a la región de ADN objetivo) y una ADN polimerasa. En el primer paso de la PCR, las dos hebras de la doble hélice del ADN se separan físicamente a alta temperatura en un proceso llamado desnaturalización del ácido nucleico. En el segundo paso, se baja la temperatura y los cebadores se unen a las secuencias complementarias de ADN. Luego, las dos cadenas de ADN se convierten en plantillas para que la ADN polimerasa ensamble enzimáticamente una nueva cadena de ADN a partir de nucleótidos libres, los componentes básicos del ADN. A medida que avanza la PCR, el ADN generado se utiliza como plantilla para la replicación, lo que pone en marcha una reacción en cadena en la que la plantilla de ADN original se amplifica exponencialmente.
Casi todas las aplicaciones de PCR emplean una polimerasa de ADN estable al calor, como la polimerasa Taq, una enzima aislada originalmente de la bacteria termófila Thermus aquaticus. Si la polimerasa usada fuera sensible al calor, se desnaturalizaría bajo las altas temperaturas del paso de desnaturalización. Antes del uso de la polimerasa Taq, la polimerasa de ADN tenía que agregarse manualmente en cada ciclo, lo que era un proceso tedioso y costoso.
Las aplicaciones de la técnica incluyen la clonación de ADN para la secuenciación, la clonación y manipulación de genes, la mutagénesis de genes; construcción de filogenias basadas en ADN o análisis funcional de genes; diagnóstico y seguimiento de trastornos genéticos; amplificación de ADN antiguo; análisis de huellas dactilares genéticas para perfiles de ADN (por ejemplo, en ciencia forense y pruebas de paternidad); y detección de patógenos en pruebas de ácido nucleico para el diagnóstico de enfermedades infecciosas.
Principios
La PCR amplifica una región específica de una cadena de ADN (el objetivo de ADN). La mayoría de los métodos de PCR amplifican fragmentos de ADN de entre 0,1 y 10 kilopares de bases (kpb) de longitud, aunque algunas técnicas permiten la amplificación de fragmentos de hasta 40 kpb. La cantidad de producto amplificado está determinada por los sustratos disponibles en la reacción, que se vuelve limitante a medida que avanza la reacción.
Una configuración básica de PCR requiere varios componentes y reactivos, entre ellos:
- a Plantilla de ADN que contiene la región objetivo de ADN para amplificar
- a polimerasa de ADN; una enzima que polimeriza nuevas cadenas de ADN; polimerasa Taq resistente al calor es especialmente común, ya que es más probable que permanezca intacta durante el proceso de desnaturalización de ADN de alta temperatura
- dos ADN cepas que son complementarios a los 3' (tres primeros) extremos de cada uno de los hilos sensoriales y anti-sentidos del objetivo de ADN (la polimerasa DNA sólo puede atar y elongar de una región de ADN de doble fisura; sin imprimaciones, no hay un sitio de iniciación de doble fisura en el que la polimerasa puede atar); las imprimaciones específicas que son complementarias de la región de ADNquímica son seleccionadas de antemano, y son a menudo un laboratorio
- deoxynucleoside triphosphates, o dNTPs (a veces llamado "desoxinucleótido triphosfatos"; nucleótidos que contienen grupos de triphosfato), los bloques de construcción de los cuales la polimerasa ADN sintetiza una nueva cadena de ADN
- a solución de amortiguación proporcionar un entorno químico adecuado para una actividad óptima y estabilidad de la polimerasa de ADN
- bivalent cations, generalmente magnesio (Mg) o manganeso (Mn) iones; Mg2+ es el más común, pero Mn2+ se puede utilizar para la mutagénesis de ADN mediada por PCR, como Mn superior2+ concentración aumenta la tasa de error durante la síntesis del ADN; y monovallent cationsiones de potasio (K)
La reacción suele llevarse a cabo en un volumen de 10 a 200 μl en pequeños tubos de reacción (volúmenes de 0,2 a 0,5 ml) en un termociclador. El termociclador calienta y enfría los tubos de reacción para alcanzar las temperaturas requeridas en cada paso de la reacción (ver más abajo). Muchos termocicladores modernos utilizan el efecto Peltier, que permite calentar y enfriar el bloque que contiene los tubos de PCR simplemente invirtiendo la corriente eléctrica. Los tubos de reacción de paredes delgadas permiten una conductividad térmica favorable para permitir un rápido equilibrio térmico. La mayoría de los termocicladores tienen tapas calentadas para evitar la condensación en la parte superior del tubo de reacción. Los termocicladores más antiguos que carecen de una tapa calentada requieren una capa de aceite encima de la mezcla de reacción o una bola de cera dentro del tubo.
Procedimiento
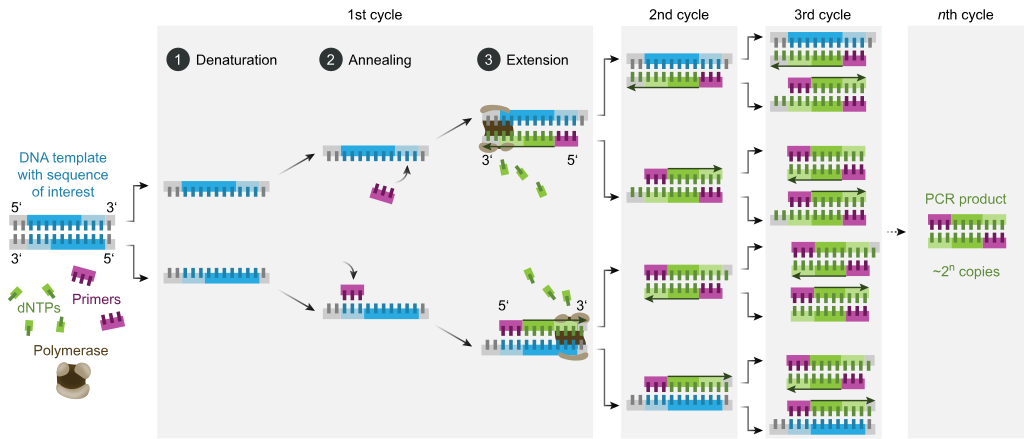
Por lo general, la PCR consta de una serie de 20 a 40 cambios de temperatura repetidos, llamados ciclos térmicos, y cada ciclo suele constar de dos o tres pasos de temperatura discretos (consulte la figura a continuación). El ciclo suele estar precedido por un solo paso de temperatura a una temperatura muy alta (>90 °C (194 °F)) y seguido de una retención al final para la extensión del producto final o un breve almacenamiento. Las temperaturas utilizadas y el tiempo que se aplican en cada ciclo dependen de una variedad de parámetros, incluida la enzima utilizada para la síntesis de ADN, la concentración de iones bivalentes y dNTP en la reacción y la temperatura de fusión (T< sub>m) de los cebadores. Los pasos individuales comunes a la mayoría de los métodos de PCR son los siguientes:
- Iniciación: Este paso sólo es necesario para las polimeras de ADN que requieren activación de calor por PCR de arranque caliente. Consiste en calentar la cámara de reacción a una temperatura de 94–96 °C (201–205 °F), o 98 °C (208 °F) si se utilizan polimeras extremadamente termoestables, que se mantiene durante 1–10 minutos.
- Denaturación: Este paso es el primer evento ciclista regular y consiste en calentar la cámara de reacción a 94–98 °C (201–208 °F) durante 20–30 segundos. Esto causa la fusión de ADN, o la desnaturalización, de la plantilla de ADN de doble tirada, rompiendo los vínculos de hidrógeno entre bases complementarias, produciendo dos moléculas de ADN de un solo tirado.
- Annealing: En el siguiente paso, la temperatura de reacción se reduce a 50–65 °C (122–149 °F) durante 20–40 segundos, lo que permite el acaparamiento de las cepas a cada una de las plantillas de ADN de una sola hoja. En la mezcla de reacción se incluyen dos imprimaciones diferentes: una para cada uno de los dos complementos monoplazados que contienen la región objetivo. Las imprimaciones son secuencias únicas, pero son mucho más cortas que la longitud de la región de destino, complementando sólo secuencias muy cortas en el extremo 3 de cada cadena.
- Es crítico determinar una temperatura adecuada para el paso de aneación porque la eficiencia y la especificidad se ven fuertemente afectadas por la temperatura aislante. Esta temperatura debe ser lo suficientemente baja como para permitir la hibridación del primer al hilo, pero lo suficientemente alta para que la hibridación sea específica, es decir, el primer debe atar sólo a una parte perfectamente complementaria del hilo, y en ninguna otra parte. Si la temperatura es demasiado baja, la imprimación puede atar imperfectamente. Si es demasiado alto, la cartilla puede no atar en absoluto. Una temperatura típica de aneación es de aproximadamente 3-5 °C debajo de Tm de las cartillas utilizadas. Los enlaces de hidrógeno estables entre bases complementarias se forman sólo cuando la secuencia de inicio coincide muy estrechamente con la secuencia de plantilla. Durante este paso, la polimerasa se une al híbrido de primera templanza y comienza la formación de ADN.
- Extension/elongation: La temperatura en este paso depende de la polimerasa de ADN utilizada; la temperatura de actividad óptima para la polimerasa de ADN termostable Taq polimerasa es aproximadamente 75–80 °C (167–176 °F), aunque una temperatura de 72 °C (162 °F) se utiliza comúnmente con esta enzima. En este paso, la polimerasa de ADN sintetiza una nueva cadena de ADN complementaria a la plantilla de ADN agregando dNTPs gratis de la mezcla de reacción que es complementaria a la plantilla en la dirección 5'-a-3', condensando el grupo 5'-fosfato de los dNTPs con el grupo 3'-hidroxy al final de la cadena de ADN naciente (perteneciente). El tiempo preciso necesario para el alargamiento depende tanto de la polimerasa de ADN utilizada como de la longitud de la región de destino de ADN para amplificar. Como regla de pulgar, a su temperatura óptima, la mayoría de las polimeras de ADN polimerizan mil bases por minuto. En condiciones óptimas (es decir, si no hay limitaciones debido a la limitación de sustratos o reactivos), en cada etapa de extensión/elongación, se duplica el número de secuencias de objetivos de ADN. Con cada ciclo sucesivo, los hilos de plantilla originales más todos los hilos recién generados se convierten en hilos de plantilla para la próxima ronda de alargamiento, lo que conduce a la amplificación exponencial (geométrica) de la región específica del objetivo de ADN.
- Los procesos de desnaturalización, aniquilación y alargamiento constituyen un único ciclo. Se requieren múltiples ciclos para amplificar el objetivo de ADN a millones de copias. La fórmula utilizada para calcular el número de copias de ADN formadas después de un número determinado de ciclos es 2n, donde n es el número de ciclos. Así, una reacción de 30 ciclos resulta en 230, o 1.073.741.824 copias de la región original de destino de ADN de doble tirada.
- Elongación final: Este solo paso es opcional, pero se realiza a una temperatura de 70–74 °C (158–165 °F) (el rango de temperatura requerido para la actividad óptima de la mayoría de las polimeras utilizadas en PCR) durante 5–15 minutos después del último ciclo de PCR para asegurar que cualquier ADN único restante esté completamente alargado.
- Final hold: El paso final enfría la cámara de reacción a 4–15 °C (39–59 °F) por un tiempo indefinido, y puede ser empleado para el almacenamiento a corto plazo de los productos PCR.

Para verificar si la PCR generó con éxito la región objetivo de ADN anticipada (a veces también denominada amplímero o amplicón), se puede emplear la electroforesis en gel de agarosa para la separación por tamaño de los productos de la PCR. El tamaño de los productos de la PCR se determina por comparación con una escalera de ADN, un marcador de peso molecular que contiene fragmentos de ADN de tamaños conocidos, que corre en el gel junto con los productos de la PCR.
Etapas
Al igual que con otras reacciones químicas, la velocidad de reacción y la eficiencia de la PCR se ven afectadas por factores limitantes. Por lo tanto, todo el proceso de PCR se puede dividir en tres etapas según el progreso de la reacción:
- Amplificación exponencial: En cada ciclo, la cantidad de producto se duplica (asumiendo la eficiencia de reacción del 100%). Después de 30 ciclos, una sola copia de ADN puede aumentar hasta 1.000.000.000 (un billón) copias. En cierto sentido, entonces, la replicación de un hilo discreto de ADN está siendo manipulada en un tubo bajo condiciones controladas. La reacción es muy sensible: sólo las cantidades minuciosas de ADN deben estar presentes.
- Nivelación fuera del escenario: La reacción disminuye a medida que la polimerasa del ADN pierde actividad y como consumo de reactivos, como dNTPs y cepas, los hace más limitados.
- Plateau: Ningún producto se acumula debido al agotamiento de reactivos y enzimas.
Optimización
En la práctica, la PCR puede fallar por varios motivos, como la sensibilidad o la contaminación. La contaminación con ADN extraño puede dar lugar a productos falsos y se aborda con protocolos y procedimientos de laboratorio que separan las mezclas previas a la PCR de los posibles contaminantes del ADN. Por ejemplo, si se analiza el ADN de la escena de un crimen, una sola molécula de ADN del personal de laboratorio podría amplificarse y desviar la investigación. Por lo tanto, las áreas de configuración de PCR están separadas del análisis o la purificación de otros productos de PCR, se utiliza material de plástico desechable y la superficie de trabajo entre las configuraciones de reacción debe limpiarse a fondo.
Especificidad se puede ajustar mediante condiciones experimentales para que no se generen productos falsos. Las técnicas de diseño de cebadores son importantes para mejorar el rendimiento del producto de PCR y evitar la formación de productos inespecíficos. El uso de componentes de tampón alternativos o enzimas de polimerasa puede ayudar con la amplificación de regiones de ADN largas o problemáticas. Por ejemplo, se dice que la polimerasa Q5 es aproximadamente 280 veces menos propensa a errores que la polimerasa Taq. Tanto los parámetros de ejecución (por ejemplo, la temperatura y la duración de los ciclos) como la adición de reactivos, como la formamida, pueden aumentar la especificidad y el rendimiento de la PCR. Se pueden realizar simulaciones informáticas de los resultados teóricos de la PCR (PCR electrónica) para ayudar en el diseño del cebador.
Aplicaciones
Aislamiento selectivo de ADN
La PCR permite el aislamiento de fragmentos de ADN a partir de ADN genómico mediante la amplificación selectiva de una región específica de ADN. Este uso de PCR aumenta muchas formas, como la generación de sondas de hibridación para la hibridación del sur o del norte y la clonación de ADN, que requieren mayores cantidades de ADN, que representan una región de ADN específica. La PCR proporciona a estas técnicas grandes cantidades de ADN puro, lo que permite el análisis de muestras de ADN incluso a partir de cantidades muy pequeñas de material de partida.
Otras aplicaciones de la PCR incluyen la secuenciación de ADN para determinar secuencias amplificadas por PCR desconocidas en las que uno de los cebadores de amplificación se puede usar en la secuenciación de Sanger, el aislamiento de una secuencia de ADN para acelerar las tecnologías de ADN recombinante que implican la inserción de una secuencia de ADN en un plásmido, fago o cósmido (dependiendo del tamaño) o el material genético de otro organismo. Las colonias bacterianas (como E. coli) pueden examinarse rápidamente mediante PCR para determinar las construcciones correctas del vector de ADN. La PCR también se puede usar para la toma de huellas genéticas; una técnica forense utilizada para identificar a una persona u organismo mediante la comparación de ADN experimental a través de diferentes métodos basados en PCR.
Algunos métodos de huellas dactilares de PCR tienen un alto poder de discriminación y se pueden usar para identificar relaciones genéticas entre individuos, como padre-hijo o entre hermanos, y se usan en pruebas de paternidad (Fig. 4). Esta técnica también se puede usar para determinar las relaciones evolutivas entre organismos cuando se usan ciertos relojes moleculares (es decir, los genes 16S rRNA y recA de microorganismos).
Amplificación y cuantificación de ADN
Debido a que la PCR amplifica las regiones de ADN a las que se dirige, la PCR se puede utilizar para analizar cantidades de muestra extremadamente pequeñas. Esto suele ser fundamental para el análisis forense, cuando solo se dispone de una pequeña cantidad de ADN como prueba. La PCR también se puede usar en el análisis de ADN antiguo que tiene decenas de miles de años. Estas técnicas basadas en PCR se han utilizado con éxito en animales, como un mamut de cuarenta mil años, y también en ADN humano, en aplicaciones que van desde el análisis de momias egipcias hasta la identificación de un zar ruso y el cuerpo de El rey inglés Ricardo III.
Los métodos de PCR cuantitativa o PCR en tiempo real (qPCR, que no debe confundirse con RT-PCR) permiten estimar la cantidad de una secuencia dada presente en una muestra, una técnica que a menudo se aplica para determinar cuantitativamente los niveles de expresión génica. La PCR cuantitativa es una herramienta establecida para la cuantificación de ADN que mide la acumulación de producto de ADN después de cada ronda de amplificación por PCR.
qPCR permite la cuantificación y detección de una secuencia de ADN específica en tiempo real ya que mide la concentración mientras se lleva a cabo el proceso de síntesis. Existen dos métodos para la detección y cuantificación simultáneas. El primer método consiste en utilizar colorantes fluorescentes que se retienen de forma inespecífica entre las dobles hebras. El segundo método implica sondas que codifican secuencias específicas y están marcadas con fluorescencia. La detección de ADN utilizando estos métodos solo se puede ver después de que tenga lugar la hibridación de las sondas con su ADN complementario (ADNc). Una combinación técnica interesante es la PCR en tiempo real y la transcripción inversa. Esta sofisticada técnica, denominada RT-qPCR, permite la cuantificación de una pequeña cantidad de ARN. A través de esta técnica combinada, el ARNm se convierte en ADNc, que luego se cuantifica mediante qPCR. Esta técnica reduce la posibilidad de error al final de la PCR, lo que aumenta las posibilidades de detección de genes asociados con enfermedades genéticas como el cáncer. Los laboratorios utilizan RT-qPCR con el fin de medir con sensibilidad la regulación génica. Los fundamentos matemáticos para la cuantificación confiable de PCR y RT-qPCR facilitan la implementación de procedimientos de ajuste precisos de datos experimentales en aplicaciones de investigación, médicas, diagnósticas y de enfermedades infecciosas.
Aplicaciones médicas y de diagnóstico
Los futuros padres pueden someterse a pruebas para determinar si son portadores genéticos, o sus hijos pueden someterse a pruebas para determinar si realmente están afectados por una enfermedad. Las muestras de ADN para las pruebas prenatales se pueden obtener mediante amniocentesis, muestreo de vellosidades coriónicas o incluso mediante el análisis de células fetales raras que circulan en el torrente sanguíneo de la madre. El análisis de PCR también es esencial para el diagnóstico genético previo a la implantación, en el que se analizan las células individuales de un embrión en desarrollo para detectar mutaciones.
- PCR también se puede utilizar como parte de una prueba sensible Tejido, vital para el trasplante de órganos. A partir de 2008, incluso hay una propuesta para reemplazar las pruebas anticuerpos tradicionales para el tipo de sangre con pruebas basadas en PCR.
- Muchas formas de cáncer implican alteraciones a oncogenes. Al utilizar pruebas basadas en PCR para estudiar estas mutaciones, los regímenes de terapia a veces se pueden personalizar individualmente a un paciente. El PCR permite el diagnóstico temprano de enfermedades malignas como la leucemia y los linfomas, que es actualmente el más desarrollado en la investigación del cáncer y ya se está utilizando rutinariamente. Los ensayos PCR se pueden realizar directamente en muestras de ADN genómico para detectar células malignas específicas de translocación en una sensibilidad que es al menos 10.000 veces superior a la de otros métodos. PCR es muy útil en el campo médico ya que permite el aislamiento y la amplificación de los supresores tumorales. PCR cuantitativa, por ejemplo, se puede utilizar para cuantificar y analizar células individuales, así como reconocer ADN, MRNA y confirmaciones y combinaciones de proteínas.
Aplicaciones de enfermedades infecciosas
La PCR permite un diagnóstico rápido y altamente específico de enfermedades infecciosas, incluidas las causadas por bacterias o virus. La PCR también permite la identificación de microorganismos no cultivables o de crecimiento lento, como micobacterias, bacterias anaerobias o virus a partir de ensayos de cultivo de tejidos y modelos animales. La base para las aplicaciones de diagnóstico de PCR en microbiología es la detección de agentes infecciosos y la discriminación de cepas no patógenas de patógenas en virtud de genes específicos.
La PCR ha revolucionado la caracterización y detección de organismos de enfermedades infecciosas de las siguientes maneras:
- El virus de inmunodeficiencia humana (o VIH), es un objetivo difícil de encontrar y erradicar. Las primeras pruebas de infección se basaron en la presencia de anticuerpos al virus circulando en el torrente sanguíneo. Sin embargo, los anticuerpos no aparecen hasta muchas semanas después de la infección, los anticuerpos maternales enmascaran la infección de un recién nacido, y los agentes terapéuticos para combatir la infección no afectan a los anticuerpos. Se han desarrollado pruebas PCR que pueden detectar tan poco como un genoma viral entre el ADN de más de 50.000 células anfitrionas. Las infecciones se pueden detectar antes, la sangre donada puede ser analizada directamente para el virus, los recién nacidos pueden ser probados inmediatamente para la infección, y los efectos de los tratamientos antivirales pueden ser cuantificados.
- Algunos organismos de la enfermedad, como el tuberculosis, son difíciles de muestra de pacientes y lento para ser cultivado en el laboratorio. Las pruebas basadas en PCR han permitido la detección de pequeñas cantidades de organismos de enfermedad (tanto vivos como muertos), en muestras convenientes. El análisis genético detallado también se puede utilizar para detectar la resistencia a los antibióticos, permitiendo una terapia inmediata y eficaz. Los efectos de la terapia también se pueden evaluar inmediatamente.
- La propagación de un organismo de enfermedad a través de poblaciones de animales domésticos o silvestres puede ser monitoreada por pruebas PCR. En muchos casos, se puede detectar y supervisar la aparición de nuevos subtipos virulentos. Los subtipos de un organismo responsable de epidemias anteriores también pueden ser determinados por el análisis de PCR.
- El ADN viral puede ser detectado por PCR. Las cartillas utilizadas deben ser específicas para las secuencias apuntadas en el ADN de un virus, y PCR se puede utilizar para análisis de diagnóstico o secuenciación de ADN del genoma viral. La alta sensibilidad de PCR permite la detección de virus poco después de la infección e incluso antes del inicio de la enfermedad. Tal detección temprana puede dar a los médicos un tiempo de plomo significativo en el tratamiento. La cantidad de virus ("carga viral") en un paciente también puede ser cuantificada por las técnicas de cuantitación de ADN basadas en PCR (ver abajo). Una variante de PCR (RT-PCR) se utiliza para detectar ARN viral en lugar de ADN: en esta prueba la enzima transcriptasa inversa se utiliza para generar una secuencia de ADN que coincide con el ARN viral; este ADN se amplifica según el método PCR habitual. RT-PCR es ampliamente utilizado para detectar el genoma viral SARS-CoV-2.
- Las enfermedades como la pertussis (o la tos ferina) son causadas por la bacteria Bordetella pertussis. Esta bacteria está marcada por una infección respiratoria aguda grave que afecta a varios animales y humanos y ha provocado la muerte de muchos niños pequeños. La toxina de la pertussis es una exotoxina de proteína que se une a los receptores celulares por dos centavos y reacciona con diferentes tipos de células como los linfocitos T que juegan un papel en la inmunidad celular. PCR es una herramienta de prueba importante que puede detectar secuencias dentro del gen para la toxina de la pertussis. Debido a que PCR tiene una alta sensibilidad para la toxina y un tiempo de giro rápido, es muy eficiente para diagnosticar la pertussis en comparación con la cultura.
Aplicaciones forenses
El desarrollo de protocolos de huellas dactilares genéticas (o ADN) basadas en PCR ha visto una aplicación generalizada en la ciencia forense:
- En su forma más discriminatoria, huella genética puede discriminar singularmente a cualquier persona de toda la población del mundo. Las muestras minutas de ADN pueden ser aisladas de una escena del crimen, y en comparación con las de sospechosos, o de una base de datos de ADN de pruebas anteriores o convictos. Las versiones más simples de estos exámenes se utilizan a menudo para descartar rápidamente a los sospechosos durante una investigación criminal. Las pruebas de crímenes de décadas pueden ser probadas, confirmando o exonerando a las personas condenadas originalmente.
- La escritura forense de ADN ha sido una manera eficaz de identificar o exonerar a sospechosos criminales debido al análisis de pruebas descubiertas en una escena del crimen. El genoma humano tiene muchas regiones repetitivas que se pueden encontrar dentro de secuencias genéticas o en regiones no codientes del genoma. Específicamente, hasta el 40% del ADN humano es repetitivo. Hay dos categorías distintas para estas regiones repetitivas y no codificación en el genoma. La primera categoría se llama número variable repeticiones tándem (VNTR), que son 10–100 pares base largos y la segunda categoría se llama repeticiones tándem cortas (STR) y estos consisten en secciones repetidos 2–10 pares base. PCR se utiliza para amplificar varios VNTRs y STR conocidos utilizando cejas que flanquean cada una de las regiones repetitivas. Los tamaños de los fragmentos obtenidos de cualquier individuo para cada uno de los STRs indicarán qué alelos están presentes. Al analizar varios STRs para un individuo, se encontrará un conjunto de alelos para cada persona que estadísticamente es probable que sea único. Los investigadores han identificado la secuencia completa del genoma humano. Esta secuencia se puede acceder fácilmente a través del sitio web de NCBI y se utiliza en muchas aplicaciones de la vida real. Por ejemplo, el FBI ha compilado un conjunto de sitios de marcadores de ADN usados para la identificación, y estos se llaman la base de datos de ADN del Sistema Combinado de Índice de ADN (CODIS). Utilizando esta base de datos, el análisis estadístico se puede utilizar para determinar la probabilidad de que una muestra de ADN coincida. PCR es una herramienta analítica muy potente y significativa para usar para escribir ADN forense porque los investigadores sólo necesitan una cantidad muy pequeña del ADN objetivo para ser usados para el análisis. Por ejemplo, un solo pelo humano con folículo capilar adjunto tiene suficiente ADN para realizar el análisis. Del mismo modo, algunos espermatozoides, muestras de piel de debajo de las uñas, o una pequeña cantidad de sangre pueden proporcionar suficiente ADN para el análisis concluyente.
- Las formas menos discriminatorias de la huella dactilar del ADN pueden ayudar en Pruebas de paternidad de ADN, donde un individuo se combina con sus parientes cercanos. El ADN de restos humanos no identificados puede ser probado, y comparado con el de posibles padres, hermanos o niños. Se pueden utilizar pruebas similares para confirmar a los padres biológicos de un niño adoptado (o secuestrado). El padre biológico real de un recién nacido también puede ser confirmado (o descartado).
- El diseño PCR AMGX/AMGY se ha demostrado que no sólo facilitar en amplificar las secuencias de ADN de una cantidad muy minúscula de genoma. Sin embargo, también se puede utilizar para la determinación sexual en tiempo real de muestras forenses óseas. Esto proporciona una manera poderosa y eficaz de determinar el género en casos forenses y especímenes antiguos.
Aplicaciones de investigación
La PCR se ha aplicado a muchas áreas de investigación en genética molecular:
- PCR permite la rápida producción de piezas cortas de ADN, incluso cuando no es más que la secuencia de las dos cartillas se conoce. Esta capacidad de PCR aumenta muchos métodos, como la generación de sondas de hibridación para la hibridación de lote sur o norte. PCR suministra estas técnicas con grandes cantidades de ADN puro, a veces como una sola hebra, permitiendo el análisis incluso de cantidades muy pequeñas de material inicial.
- La tarea de secuenciación de ADN también puede ser asistido por PCR. Se pueden producir fácilmente segmentos conocidos de ADN de un paciente con mutación genética. Las modificaciones a la técnica de amplificación pueden extraer segmentos de un genoma completamente desconocido, o pueden generar sólo una sola cadena de un área de interés.
- PCR tiene numerosas aplicaciones al proceso más tradicional clonación de ADN. Puede extraer segmentos para la inserción en un vector de un genoma más grande, que sólo puede estar disponible en pequeñas cantidades. Utilizando un único conjunto de 'pantallas vencedoras', también puede analizar o extraer fragmentos que ya se han insertado en vectores. Algunas alteraciones del protocolo PCR pueden generar mutaciones (general o dirigida por el sitio) de un fragmento insertado.
- Sitios marcados por secuencias es un proceso donde PCR se utiliza como un indicador que un segmento particular de un genoma está presente en un clon en particular. El Proyecto Genoma Humano encontró esta aplicación vital para mapear los clones acogedores que estaban secuenciando y coordinar los resultados de diferentes laboratorios.
- Una aplicación de PCR es el análisis fitogénico del ADN fuentes antiguas, como el que se encuentra en los huesos recuperados de Neanderthals, de tejidos congelados de mamuts, o del cerebro de momias egipcias. En algunos casos, el ADN altamente degradado de estas fuentes podría ser reagrupado durante las primeras etapas de amplificación.
- Una aplicación común de PCR es el estudio de patrones expresión. Los tejidos (o incluso las células individuales) se pueden analizar en diferentes etapas para ver qué genes se han vuelto activos, o que se han apagado. Esta aplicación también puede utilizar PCR cuantitativa para cuantificar los niveles reales de expresión
- La capacidad de PCR para amplificar simultáneamente varios loci de esperma individual ha mejorado mucho la tarea más tradicional de Cartografía genética estudiando cruces cromosómicas después de la meiosis. Rare crossover eventos entre loci muy cercanos han sido observados directamente analizando miles de espermatozoides individuales. Del mismo modo, se pueden analizar borraciones, inserciones, translocaciones o inversiones inusuales, sin tener que esperar (o pagar) por los procesos largos y laboriosos de fertilización, embriogénesis, etc.
- mutagenesis dirigida por el sitio: PCR se puede utilizar para crear genes mutantes con mutaciones elegidas por científicos a voluntad. Estas mutaciones se pueden elegir para entender cómo las proteínas cumplen sus funciones, y para cambiar o mejorar la función proteica.
Ventajas
PCR tiene una serie de ventajas. Es bastante simple de entender y usar, y produce resultados rápidamente. La técnica es muy sensible y tiene el potencial de producir de millones a miles de millones de copias de un producto específico para secuenciación, clonación y análisis. qRT-PCR comparte las mismas ventajas que la PCR, con la ventaja añadida de la cuantificación del producto sintetizado. Por lo tanto, tiene sus usos para analizar alteraciones de los niveles de expresión génica en tumores, microbios u otros estados patológicos.
PCR es una herramienta de investigación muy poderosa y práctica. La PCR está determinando la secuenciación de etiologías desconocidas de muchas enfermedades. La técnica puede ayudar a identificar la secuencia de virus previamente desconocidos relacionados con los ya conocidos y así darnos una mejor comprensión de la enfermedad en sí. Si el procedimiento se puede simplificar aún más y se pueden desarrollar sistemas sensibles de detección no radiométrica, la PCR ocupará un lugar destacado en el laboratorio clínico en los años venideros.
Limitaciones
Una de las principales limitaciones de la PCR es que se necesita información previa sobre la secuencia diana para generar los cebadores que permitirán su amplificación selectiva. Esto significa que, por lo general, los usuarios de PCR deben conocer la(s) secuencia(s) precisa(s) aguas arriba de la región objetivo en cada una de las dos plantillas monocatenarias para garantizar que la ADN polimerasa se una correctamente a los híbridos cebador-plantilla y, posteriormente, genere la toda la región diana durante la síntesis de ADN.
Como todas las enzimas, las ADN polimerasas también son propensas a errores, lo que a su vez provoca mutaciones en los fragmentos de PCR que se generan.
Otra limitación de la PCR es que se puede amplificar incluso la cantidad más pequeña de ADN contaminante, lo que genera resultados engañosos o ambiguos. Para minimizar la posibilidad de contaminación, los investigadores deben reservar salas separadas para la preparación de reactivos, la PCR y el análisis del producto. Los reactivos deben dispensarse en alícuotas de un solo uso. Deben utilizarse de forma rutinaria pipetas con émbolos desechables y puntas de pipeta extralargas. Además, se recomienda asegurarse de que la configuración del laboratorio siga un flujo de trabajo unidireccional. Ningún material o reactivo utilizado en las salas de análisis y PCR debe llevarse nunca a la sala de preparación de PCR sin una descontaminación completa.
Las muestras ambientales que contienen ácidos húmicos pueden inhibir la amplificación por PCR y generar resultados inexactos.
Variaciones
- PCR específico de Allele: una técnica de diagnóstico o clonación basada en variaciones de un solo núcleoide (SNVs que no deben confundirse con SNPs) (diferencias de base única en un paciente). Requiere conocimiento previo de una secuencia de ADN, incluyendo diferencias entre los alelos, y utiliza cepas cuyos 3' extremos abarcan el SNV (base par buffer alrededor de SNV generalmente incorporado). La amplificación PCR bajo condiciones estrictas es mucho menos eficiente en presencia de un desajuste entre plantilla y cartilla, así que la amplificación exitosa con una presencia de imprimación específica SNP indica la presencia del SNP específico en una secuencia. Consulte el genotipado SNP para obtener más información.
- Assembly PCR o Polymerase Cycling Assembly (PCA): síntesis artificial de largas secuencias de ADN realizando PCR en una piscina de oligonucleótidos largos con segmentos de superposición cortos. Los oligonucleótidos se alternan entre direcciones sensoriales y antisensatas, y los segmentos superpuestos determinan el orden de los fragmentos de PCR, produciendo selectivamente el producto final de ADN largo.
- PCR asimétrico: Amplifica preferentemente una cadena de ADN en una plantilla de ADN doble. Se utiliza en secuenciación e hibridación probing donde se requiere amplificación de sólo uno de los dos hilos complementarios. PCR se lleva a cabo como de costumbre, pero con un gran exceso de la imprimación para el hilo apuntado para la amplificación. Debido a la amplificación lenta (aritmética) más tarde en la reacción después de que se ha utilizado el primer límite, se requieren ciclos adicionales de PCR. Una modificación reciente de este proceso, conocido como LInear-AFter...TÉl...Exponential-PCR (LATE-PCR), utiliza una imprimación limitante con una temperatura de fusión más alta (Tm) que la imprimación de exceso para mantener la eficiencia de reacción ya que la concentración de imprimación limita disminuye la reacción media.
- PCR convectivo: una forma pseudo-isotérmica de realizar PCR. En lugar de calentar y enfriar repetidamente la mezcla PCR, la solución está sujeta a un gradiente térmico. La inestabilidad térmica resultante impulsada por el flujo convectivo elimina automáticamente los reactivos PCR de las regiones calientes y frías que permiten repetidamente PCR. Los parámetros como las condiciones de los límites térmicos y la geometría del recinto PCR pueden optimizarse para producir PCR robusto y rápido aprovechando el surgimiento de campos de flujo caótico. Tal configuración PCR de flujo convectivo reduce significativamente el requisito de potencia del dispositivo y el tiempo de operación.
- PCR de eliminación: un método muy paralelo para recuperar moléculas de ADN precisas para la síntesis de genes. Una biblioteca compleja de moléculas de ADN se modifica con etiquetas de flanque único antes de secuenciar masivamente paralelo. Las cartillas dirigidas por la etiqueta permiten la recuperación de moléculas con secuencias deseadas por PCR.
- PCR digital (dPCR): utilizado para medir la cantidad de una secuencia de ADN objetivo en una muestra de ADN. La muestra de ADN está muy diluida para que después de ejecutar muchos PCR en paralelo, algunos de ellos no reciban una sola molécula del ADN objetivo. La concentración de ADN objetivo se calcula utilizando la proporción de resultados negativos. Por lo tanto, el nombre "PCR digital".
- Amplificación dependiente de la helicasa: similar a la PCR tradicional, pero utiliza una temperatura constante en lugar de ciclismo a través de ciclos de desnaturalización y aniquilación/extensión. Helicasa de ADN, una enzima que desenrolla el ADN, se utiliza en lugar de desnaturalización térmica.
- Comienzo caliente PCR: una técnica que reduce la amplificación no específica durante las etapas iniciales del PCR. Se puede realizar manualmente calentando los componentes de reacción a la temperatura de desnaturalización (por ejemplo, 95 °C) antes de añadir la polimerasa. Se han desarrollado sistemas de enzimas especializados que inhiben la actividad de la polimerasa a temperatura ambiente, ya sea por la unión de un anticuerpo o por la presencia de inhibidores ligados covalentemente que se disocian sólo después de un paso de activación de alta temperatura. PCR de arranque caliente / frío se consigue con nuevas polimeras híbridas que son inactivas a temperatura ambiente y se activan instantáneamente a temperatura de alargamiento.
- En silico PCR (digital PCR, PCR virtual, PCR electrónico, e-PCR) se refiere a las herramientas computacionales utilizadas para calcular los resultados teóricos de la reacción de la cadena de polimerasa utilizando un conjunto dado de imprimaciones (probes) para amplificar secuencias de ADN de un genoma secuenciado o transcriptoma. En silico PCR se propuso como una herramienta educativa para la biología molecular.
- Intersequence-specific PCR (ISSR): un método PCR para la huella de ADN que amplifica regiones entre secuencias simples repite para producir una huella única de longitudes de fragmento amplificadas.
- PCR inverso: se utiliza comúnmente para identificar las secuencias de flanqueo alrededor de insertos genómicos. Se trata de una serie de digestión de ADN y de auto ligación, dando lugar a secuencias conocidas al final de la secuencia desconocida.
- PCR mediado por la ligación: utiliza pequeños vinculos de ADN ligados al ADN de interés y múltiples cepas acuáticas a los vinculos de ADN; se ha utilizado para secuenciación de ADN, andación de genomas y huella de ADN.
- Metilación específica PCR (MSP): desarrollado por Stephen Baylin y James G. Herman en la Johns Hopkins School of Medicine, y se utiliza para detectar la metilación de las islas CpG en ADN genómico. El ADN se trata por primera vez con bisulfito de sodio, que convierte bases de citosina no metilada a uracil, que es reconocida por las imprimaciones PCR como timina. Dos PCR se llevan a cabo en el ADN modificado, utilizando conjuntos de imprimación idénticos excepto en cualquier islas CpG dentro de las secuencias de imprimación. En estos puntos, un conjunto de imprimación reconoce el ADN con citocinas para amplificar el ADN metilado, y un conjunto reconoce el ADN con uracil o timina para amplificar el ADN no atenuado. MSP usando qPCR también se puede realizar para obtener información cuantitativa y no cualitativa sobre la metilación.
- Miniprimer PCR: utiliza una polimerasa termoestable (S-Tbr) que puede extenderse desde las cartillas cortas ("smalligos") tan cortas como 9 o 10 nucleótidos. Este método permite que el PCR se oriente hacia regiones más pequeñas de primera unión, y se utiliza para amplificar secuencias de ADN conservadas, como el gen 16S (o eucariotic 18S) rRNA.
- Amplificación de sonda dependiente de ligación múltiple ()MLPA): permite amplificar múltiples objetivos con un solo par de imprimación, evitando así las limitaciones de resolución de multix PCR (ver abajo).
- Multiplexx-PCR: consiste en múltiples conjuntos de imprimación dentro de una sola mezcla PCR para producir amplicons de diferentes tamaños que son específicos a diferentes secuencias de ADN. Al seleccionar varios genes a la vez, puede obtenerse información adicional de una sola prueba que de otra manera requeriría varias veces los reactivos y más tiempo para realizar. Las temperaturas de recocido para cada uno de los conjuntos de imprimación deben ser optimizadas para funcionar correctamente dentro de una sola reacción, y tamaños de amplicon. Es decir, su longitud de par base debe ser lo suficientemente diferente para formar bandas distintas cuando se visualiza por electroforesis gel.
- PCR con ayuda de nanopartícula (nanoPCR): algunas nanopartículas (PN) pueden mejorar la eficiencia de PCR (por lo que se llama nanoPCR), y algunos pueden incluso superar los potenciadores PCR originales. It was reported that quantum dots (QDs) can improve PCR specificity and efficiency. Los nanotubos de carbono de paredes únicas (SWCNTs) y los nanotubos de carbono de paredes múltiples (MWCNTs) son eficaces para mejorar la amplificación de PCR largo. La nanopolvosa de carbono (CNP) puede mejorar la eficiencia de PCR repetidas y PCR largas, mientras que el óxido de zinc, dióxido de titanio y Ag NPs se encontraron para aumentar el rendimiento de PCR. Los datos anteriores indicaron que los NP no metálicos mantenían una fidelidad aceptable de amplificación. Dado que muchos NP son capaces de mejorar la eficiencia de PCR, es evidente que es probable que haya un gran potencial para las mejoras de la tecnología nanoPCR y el desarrollo de productos.
- PCR anidado: aumenta la especificidad de la amplificación del ADN, reduciendo el fondo debido a la amplificación no específica del ADN. Se utilizan dos conjuntos de imprimaciones en dos PCR sucesivos. En la primera reacción, se utiliza un par de imprimaciones para generar productos de ADN, que además del objetivo previsto, pueden consistir todavía en fragmentos de ADN no especificamente amplificados. El producto(s) se utiliza en un segundo PCR con un conjunto de imprimaciones cuyos sitios de unión son completamente o parcialmente diferentes de y ubicados 3' de cada una de las imprimaciones utilizadas en la primera reacción. PCR anidado es a menudo más exitoso en amplificar fragmentos de ADN largos específicamente que PCR convencional, pero requiere un conocimiento más detallado de las secuencias de destino.
- Overlap-extension PCR o Splicing by overlap extension (SOEing) : una técnica de ingeniería genética que se utiliza para juntar dos o más fragmentos de ADN que contienen secuencias complementarias. Se utiliza para unir piezas de ADN que contienen genes, secuencias regulatorias o mutaciones; la técnica permite la creación de construcciones específicas y largas de ADN. También puede introducir eliminaciones, inserciones o mutaciones de puntos en una secuencia de ADN.
- PAN-AC: utiliza condiciones isotérmicas para la amplificación, y puede ser utilizado en células vivas.
- PCR cuantitativa (qPCR): utilizado para medir la cantidad de una secuencia de destino (comúnmente en tiempo real). Mide cuantitativamente las cantidades iniciales de ADN, cDNA o ARN. El PCR cuantitativo se utiliza comúnmente para determinar si una secuencia de ADN está presente en una muestra y el número de sus copias en la muestra. PCR cuantitativa tiene un alto grado de precisión. Los métodos de PCR cuantitativos usan tintes fluorescentes, como Sybr Green, EvaGreen o sondas de ADN que contienen fluorofre, como TaqMan, para medir la cantidad de producto amplificado en tiempo real. También a veces se abrevia a RT-PCR (RT-PCR)en tiempo real PCR) pero esta abreviatura debe usarse sólo para la transcripción inversa PCR. qPCR son las contracciones apropiadas para PCR cuantitativa (PCR en tiempo real).
- PCR (RC-PCR): Permite que la adición de dominios funcionales o secuencias de elección sea anexada independientemente al extremo del amplicon generado en una sola reacción de tubo cerrado. Este método genera imprimaciones específicas apuntadas dentro de la reacción por la interacción de las cartillas universales (que contienen las secuencias deseadas o dominios a ser anexados) y sondas RC.
- Transcripción PCR (RT-PCR): para amplificar el ADN del ARN. La transcripción inversa transcribe RNA en cDNA, que luego es amplificada por PCR. RT-PCR es ampliamente utilizado en perfiles de expresión, para determinar la expresión de un gen o para identificar la secuencia de una transcripción del ARN, incluyendo sitios de inicio de transcripción y terminación. Si se conoce la secuencia genómica de ADN de un gen, RT-PCR se puede utilizar para mapear la ubicación de exones e intrones en el gen. El final de 5' de un gen (correspondiente al sitio de inicio de la transcripción) es generalmente identificado por RACE-PCR (Amplificación rápida de cDNA Ends).
- RNase H-dependiente PCR (rhPCR): una modificación de PCR que utiliza las imprimaciones con un bloque de extensión de 3' que puede ser eliminado por una enzima termostable RNase HII. Este sistema reduce los ímpetu-dimers y permite realizar reacciones multiplexadas con mayor número de imprimaciones.
- Imprimación única específica (SSP-PCR): permite la amplificación del ADN de doble tirada incluso cuando la información de secuencia está disponible sólo en un extremo. Este método permite la amplificación de genes para los cuales sólo se dispone de una información de secuencia parcial, y permite el genoma unidireccional caminando desde regiones conocidas hasta regiones desconocidas del cromosoma.
- PCR de fase sólida: abarca múltiples significados, incluyendo Polony Amplification (donde las colonias PCR se derivan en una matriz de gel, por ejemplo), Bridge PCR (los primitivos están covalentemente vinculados a una superficie de soporte sólido), PCR de fase sólida convencional (donde el PCR asimétrico se aplica en la presencia de soporte sólido primitivo con secuencia que coincida con una de las imprimaciones acuosas) y Sólido mejorado PCR de fase sólida
- Suicidio PCR: típicamente utilizado en paleogenética u otros estudios donde evitar falsos positivos y asegurar la especificidad del fragmento amplificado es la máxima prioridad. Se describió originalmente en un estudio para verificar la presencia del microbio Yersinia pestis en muestras dentales obtenidas de tumbas del siglo XIV de personas supuestamente asesinadas por la plaga durante la epidemia medieval de la muerte negra. El método prescribe el uso de cualquier combinación de imprimación sólo una vez en un PCR (de ahí el término "suicidio"), que nunca debe haber sido utilizado en cualquier reacción PCR de control positivo, y las imprimaciones siempre deben dirigirse a una región genómica nunca amplificada antes en el laboratorio utilizando este o cualquier otro conjunto de imprimaciones. Esto asegura que no contaminar el ADN de las reacciones PCR anteriores está presente en el laboratorio, que de otro modo podría generar falsos positivos.
- PCR entrelazado térmico asimétrico (TAIL-PCR): para el aislamiento de una secuencia desconocida flanqueando una secuencia conocida. Dentro de la secuencia conocida, TAIL-PCR utiliza un par anidado de imprimaciones con diferentes temperaturas aniquiladoras; una imprimación degenerada se utiliza para amplificar en la otra dirección de la secuencia desconocida.
- Touchdown PCR ()PCR desplegable): una variante de PCR que tiene como objetivo reducir el fondo no específico reduciendo gradualmente la temperatura de acaparamiento a medida que avanza el ciclismo PCR. La temperatura acuática en los ciclos iniciales es generalmente unos pocos grados (3–5 °C) por encima de la Tm de las imprimaciones utilizadas, mientras que en los ciclos posteriores, es unos pocos grados (3–5 °C) debajo de la imprimación Tm. Las temperaturas más altas dan mayor especificidad para la fijación de la imprimación, y las temperaturas más bajas permiten una amplificación más eficiente de los productos específicos formados durante los ciclos iniciales.
- Paseo rápido universal: para el genoma caminando y la huella genética usando un PCR más específico "dos-sided" que los enfoques convencionales "uno-sided" (utilizando sólo una sola cartilla específica para genes y una cartilla general, que puede llevar a la "noise" artificial en virtud de un mecanismo que implica la formación de la estructura de laría. Los derivados Streamlined de UFW son LaNe RAGE (lariat-dependiente de PCR anidado para una rápida amplificación de los fines genómicos del ADN), 5'RACE LaNe y 3'RACE LaNe.
Historia
Las enzimas resistentes al calor que son un componente clave en la reacción en cadena de la polimerasa se descubrieron en la década de 1960 como producto de una forma de vida microbiana que vivía en las aguas sobrecalentadas de Mushroom Spring de Yellowstone.
Un artículo de 1971 en el Journal of Molecular Biology escrito por Kjell Kleppe y colaboradores en el laboratorio de H. Gobind Khorana describió por primera vez un método de uso de un ensayo enzimático para replicar una plantilla corta de ADN con cebadores in vitro. Sin embargo, esta primera manifestación del principio básico de PCR no recibió mucha atención en ese momento y la invención de la reacción en cadena de la polimerasa en 1983 generalmente se atribuye a Kary Mullis.
Cuando Mullis desarrolló la PCR en 1983, trabajaba en Emeryville, California, para Cetus Corporation, una de las primeras empresas de biotecnología, donde era responsable de sintetizar cadenas cortas de ADN. Mullis ha escrito que concibió la idea de PCR mientras recorría la Pacific Coast Highway una noche en su automóvil. Estaba jugando en su mente con una nueva forma de analizar los cambios (mutaciones) en el ADN cuando se dio cuenta de que, en cambio, había inventado un método para amplificar cualquier región del ADN a través de ciclos repetidos de duplicación impulsados por la ADN polimerasa. En Scientific American, Mullis resumió el procedimiento: “Comenzando con una sola molécula del material genético ADN, la PCR puede generar 100 mil millones de moléculas similares en una tarde. La reacción es fácil de ejecutar. No requiere más que un tubo de ensayo, algunos reactivos simples y una fuente de calor." La toma de huellas dactilares de ADN se utilizó por primera vez para las pruebas de paternidad en 1988.
Mullis ha acreditado su uso de LSD como parte integral de su desarrollo de PCR: "¿Habría inventado PCR si no hubiera tomado LSD? Lo dudo seriamente. Podría sentarme en una molécula de ADN y ver pasar los polímeros. Aprendí eso en parte con drogas psicodélicas."
Mullis y el bioquímico Michael Smith, que había desarrollado otras formas esenciales de manipular el ADN, recibieron conjuntamente el Premio Nobel de Química en 1993, siete años después de que Mullis y sus colegas de Cetus pusieran en práctica su propuesta por primera vez. El artículo de Mullis de 1985 con R. K. Saiki y H. A. Erlich, 'Enzymatic Amplification of β-globin Genomic Sequences and Restriction Site Analysis for Diagnosis of Sickle Cell Anemia', la invención de la reacción en cadena de la polimerasa (PCR), fue honrado con una citación para el premio Chemical Breakthrough Award de la División de Historia de la Química de la American Chemical Society en 2017.
En el centro del método de PCR se encuentra el uso de una ADN polimerasa adecuada capaz de soportar las altas temperaturas de >90 °C (194 °F) requeridas para la separación de las dos cadenas de ADN en la doble hélice de ADN después de cada ciclo de replicación. Las polimerasas de ADN empleadas inicialmente para experimentos in vitro que presagiaban PCR no pudieron soportar estas altas temperaturas. Por lo tanto, los primeros procedimientos para la replicación del ADN eran muy ineficientes y requerían mucho tiempo, y requerían grandes cantidades de ADN polimerasa y un manejo continuo durante todo el proceso.
El descubrimiento en 1976 de la polimerasa Taq, una ADN polimerasa purificada de la bacteria termófila, Thermus aquaticus, que vive naturalmente en ambientes cálidos (50 a 80 °C (122 a 176 °F)) como aguas termales— allanaron el camino para mejoras dramáticas del método PCR. La ADN polimerasa aislada de T. aquaticus es estable a altas temperaturas manteniéndose activo incluso después de la desnaturalización del ADN, obviando así la necesidad de añadir nueva ADN polimerasa después de cada ciclo. Esto permitió un proceso basado en un termociclador automatizado para la amplificación de ADN.
Disputas de patentes
La técnica PCR fue patentada por Kary Mullis y asignada a Cetus Corporation, donde Mullis trabajaba cuando inventó la técnica en 1983. La enzima polimerasa Taq también estaba cubierta por patentes. Ha habido varias demandas de alto perfil relacionadas con la técnica, incluida una demanda sin éxito presentada por DuPont. La compañía farmacéutica suiza Hoffmann-La Roche compró los derechos de las patentes en 1992. La última de las patentes comerciales de PCR expiró en 2017.
Una batalla relacionada con la patente sobre la enzima polimerasa Taq sigue en curso en varias jurisdicciones de todo el mundo entre Roche y Promega. Los argumentos legales se han extendido más allá de la vigencia de las patentes originales de PCR y Taq polimerasa, que expiraron el 28 de marzo de 2005.
Contenido relacionado
Ave marina
Quebrada de Samariá
Justicia brandegeeana