PyMOL
PyMOL es un sistema de visualización molecular de código abierto pero patentado creado por Warren Lyford DeLano. Fue comercializado inicialmente por DeLano Scientific LLC, que era una empresa de software privada dedicada a crear herramientas útiles que sean universalmente accesibles para las comunidades científicas y educativas. Actualmente lo comercializa Schrödinger, Inc. Como la licencia de software original era una licencia permisiva, pudieron eliminarla; Las nuevas versiones ya no se publican bajo la licencia Python, sino bajo una licencia personalizada (que otorga amplios derechos de uso, redistribución y modificación, pero asigna derechos de autor a cualquier versión a Schrodinger, LLC.), y parte del código fuente ya no se publica. . PyMOL puede producir imágenes 3D de alta calidad de moléculas pequeñas y macromoléculas biológicas, como las proteínas. Según el autor original, en 2009, casi una cuarta parte de todas las imágenes publicadas de estructuras de proteínas 3D en la literatura científica se crearon utilizando PyMOL.
PyMOL es una de las pocas herramientas de visualización de modelos, en su mayoría de código abierto, disponibles para su uso en biología estructural. La parte Py del nombre del software se refiere a que el programa fue escrito en el lenguaje de programación Python.
PyMOL utiliza la biblioteca OpenGL Extension Wrangler (GLEW) y FreeGLUT, y puede resolver ecuaciones de Poisson-Boltzmann utilizando el solucionador adaptable de Poisson Boltzmann. PyMOL usó Tk para los widgets GUI y tenía binarios Aqua nativos para macOS a través de Schrödinger, que fueron reemplazados por una interfaz de usuario PyQt en todas las plataformas con el lanzamiento de la versión 2.0.
Historia y comercialización
Las primeras versiones de PyMol se lanzaron bajo la licencia Python. El 1 de agosto de 2006, DeLano Scientific adoptó un sistema de descarga de acceso controlado para compilaciones PyMOL precompiladas (incluidas las betas) distribuidas por la empresa. El acceso a estos ejecutables ahora está limitado a usuarios registrados que sean clientes de pago; Las compilaciones educativas están disponibles de forma gratuita para estudiantes y profesores. Sin embargo, la mayor parte del código fuente actual sigue estando disponible de forma gratuita, al igual que las versiones precompiladas más antiguas. Si bien los sistemas de compilación para otras plataformas están abiertos, el sistema de compilación API de Windows (WinAPI, Win32) no lo está, aunque hay binarios no oficiales de Windows disponibles en línea. Cualquiera puede compilar un ejecutable a partir del código fuente con licencia de Python o pagar una suscripción a servicios de soporte para obtener acceso a ejecutables precompilados.
El 8 de enero de 2010, Schrödinger, Inc. llegó a un acuerdo para adquirir PyMOL. La firma asumió el desarrollo, mantenimiento, soporte y ventas de PyMOL, incluidas todas las suscripciones válidas en ese momento. También continúan apoyando activamente a la comunidad de código abierto PyMOL. En 2017, Schrödinger renovó el sistema de distribución para unificar la interfaz de usuario en Qt y la gestión de paquetes en Anaconda, y lo lanzó como PyMol v2. Esta versión restringe algunas funcionalidades nuevas y agrega una marca de agua a la visualización si se usa sin licencia más allá del período de prueba de 30 días; La política general de licencias es similar a la del sistema DeLano. La mayor parte del código fuente sigue estando disponible, esta vez bajo una licencia tipo BSD. Al igual que con la distribución anterior, hay disponibles binarios no oficiales de Windows en formato de rueda y, de hecho, las distribuciones de Linux continúan proporcionando sus propias versiones del código fuente abierto.
Colores de elementos
PyMOL aplica coloración de bolas por elemento.
| Tabla periódica usando los colores del elemento PyMOL como fondo | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | Hydro-genH#e5e5e5 | HelioHe#d8ffff | |||||||||||||||||
| 2 | Lith-iumLi#cc7fff | Be#c2ff00 | BoronB#ffb5b5 | CarbonC#33ff33 | NitrogenN#3333ff | Oxy-genO#ff4c4c | Fluor-ineF#b2ffff | NeonNe#b2e2f5 | |||||||||||
| 3 | SodiumNa#aaa5cf2 | MagnesiumMg#8aff00 | AluminioAl#bfa5a | Sili-conSi#f0c79f | PhosphorusP#ff7f00 | SulfurS#e5c53f | ChlorineCl#1ef01e | ArgonAr#7fd0e2 | |||||||||||
| 4 | PotasioK#8f3fd3 | CalcioCa#3cff00 | ScandiumSc#e5e5e5 | TitanioTi#bfc2c7 | VanadiumV#a5a5aaa | Chrom-iumCr#8a99c7 | Manga-neseMn#9c7ac7 | IronFe#e06633 | CobaltoCo#f08f9f | NickelNi#4fd04f | CopperCu#c77f33 | ZincZn#7c7faf | GalliumGa#c28f8f | GermaniumGe#668f8f | ArsenicAs#bd7fe2 | SelenioSe#ffa000 | BromineBr#a529 | Kryp-tonKr#5cb7d0 | |
| 5 | RubidiumRb#6f2eaf | Stront-iumSr.#00ff00 | YttriumY#94ffffff | Zirco-niumZr#94e0e0 | Nio-biumNb#72c2c8 | MolibdenoMo#54b5b | TechnetiumTc#3a9d9d | Ruthe-niumRu#248f8f | RhodiumRh#097c8c | Pallad-iumPd#006984 | SilverAg#bfbf | Cad-miumCd#ffd88f | IndiumIn#a57472 | TinSn#667f7f | Anti-monySb#9d62b5 | Tellur-iumTe#d37a00 | IodineI#940094 | XenonXe#419daf | |
| 6 | Cae-siumCs#57168f | BariumBa#00c800 | LutetiumLu#00aa24 | Haf-niumHf#4cc2ff | Tanta-lumTa#4ca5ff | Tung-stenW#2194d5 | RheniumRe#267caa | OsmiumOs#266695 | IridiumIr#165487 | PlatinumPt#d0d0e0 | GoldAu#ffd023 | Mer-curyHg#b7b7d0 | ThalliumTl#a5544c | LeadPb#575961 | Bis-muthBi#9d4fb5 | Polonio | AstatineAt#744f44 | RadonRn#418295 | |
| 7 | Fran-ciumFr#410066 | RadiumRa#007c00 | Lawren-ciumLr#c70066 | Ruther-fordiumRf#cc0059 | Dub-niumDb#d0004f | Sea—borgiumSg#d80044 | Bohr-iumBh#e00037 | HassiumHs#e5002e | Meit -neriumMt#ea0026 | Darm-stadtium Ds... | Roentgenium Rg... | Coper-nicium Cn... | Nihonio Nh... | Flerov.iumFl. | Moscov -iumMc- | Liver-moriumLv – | TennessineTs – | Oga-nessonOg- | |
| Lan-thanumLa#6fd3ff | CeriumCe#ffffc7 | Praseo-dymiumPr#d8ffc7 | NeodymiumNd#c7ffc7 | PrometioPm#a2ffc7 | SamariumSm#8fffc7 | EuropiumEu#61ffc7 | Gadolin-iumGd#44ffc7 | Ter-biumTb#2fffc7 | Dyspro-siumDy#1effc7 | HolmiumHo#00ff9c | ErbiumEr#00e574 | ThuliumTm#00d351 | Ytter-biumYb#00bf37 | ||||||
| Actin-iumAc#6faafa | ThorioTh#00baff | ProtactiniumPa#00a0ff | UranioU#008fff | NeptunioNp#007fff | PlutónioPu#006aff | AmericiumAm#545cf2 | CuriumCm#775ce2 | Berkel-iumBk#8a4fe2 | CalifornioCf#a036d3 | EinsteiniumEs#b21ed3 | FermiumFm#b21eba | Mende-leviumMd#b20ca5 | Nobel No#bd0c87 | ||||||
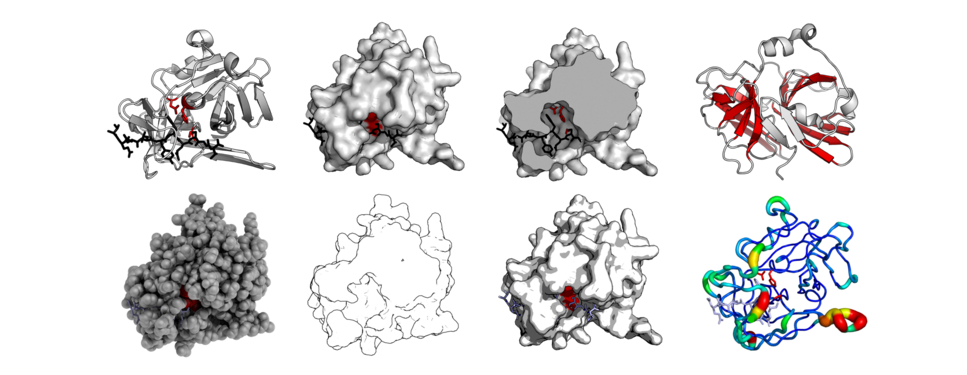
Galería
- Ejemplo de algunas características de edición de moléculas de PyMOL, rotación de bonos dihedral y relajación molecular interactiva con Modo de escultura. Estas son características útiles para preparar la geometría de entrada para el software de química cuántica
- La misma estructura proteica (TEV proteasa - PDB: 1LVB) renderizado en diferentes modos. Caricatura estándar, superficie, corte de superficie, barriles destacados, 'QuteMol'-como, 'Goodsell'-como, brillante-superficie, y b-factor putty.