Puente salino (proteico y supramolecular)

En química, un puente salino es una combinación de dos interacciones no covalentes: enlaces de hidrógeno y enlaces iónicos (Figura 1). El apareamiento iónico es una de las fuerzas no covalentes más importantes en química, en sistemas biológicos, en diferentes materiales y en muchas aplicaciones como la cromatografía de pares iónicos. Es una contribución observada más comúnmente a la estabilidad de la conformación plegada entrópicamente desfavorable de las proteínas. Aunque se sabe que las interacciones no covalentes son interacciones relativamente débiles, pequeñas interacciones estabilizadoras pueden sumar una contribución importante a la estabilidad general de un confórmero. Los puentes salinos no sólo se encuentran en las proteínas, sino que también se pueden encontrar en la química supramolecular. La termodinámica de cada uno se explora a través de procedimientos experimentales para acceder a la contribución de energía libre del puente salino a la energía libre general del estado.
Puentes salinos en enlaces químicos
En el agua, la formación de puentes salinos o pares iónicos está impulsada principalmente por la entropía, generalmente acompañada de contribuciones desfavorables de ΔH debido a la desolvatación de los iones que interactúan tras la asociación. Los enlaces de hidrógeno contribuyen a la estabilidad de los pares iónicos, p. iones de amonio protonados, y con aniones se forma por desprotonación como en el caso del carboxilato, fosfato, etc.; entonces las constantes de asociación dependen del pH. Las fuerzas impulsoras entrópicas para el apareamiento iónico (en ausencia de contribuciones significativas de enlaces H) también se encuentran en el metanol como disolvente. En el contacto con disolventes no polares se forman pares iónicos con constantes de asociación muy altas; en la fase gaseosa las energías de asociación de p.e. los haluros alcalinos alcanzan hasta 200 kJ/mol. La ecuación de Bjerrum o de Fuoss describe la asociación de pares iónicos en función de las cargas iónicas zA y zB y la constante dieléctrica ε del medio; un gráfico correspondiente de la estabilidad ΔG frente a zAzB muestra para más de 200 pares de iones la correlación lineal esperada para una gran variedad de iones. Los iones inorgánicos y orgánicos se muestran con una fuerza iónica moderada y una asociación de puente salino similar. Valores de ΔG de alrededor de 5 a 6 kJ/mol para una combinación 1:1 de anión y catión, casi independiente de la naturaleza (tamaño, polarizabilidad, etc.) de los iones. . Los valores de ΔG son aditivos y aproximadamente una función lineal de las cargas, la interacción de, por ejemplo, un anión fosfato doblemente cargado con un catión amonio con carga única representa aproximadamente 2x5 = 10 kJ/mol. Los valores de ΔG dependen de la fuerza iónica I de la solución, como se describe en la ecuación de Debye-Hückel, con fuerza iónica cero se observa ΔG = 8 kJ/mol. Las estabilidades de los pares de iones álcali en función de la carga del anión zby se pueden describir mediante una ecuación más detallada.
Puentes salinos que se encuentran en las proteínas
El puente salino surge con mayor frecuencia del carboxilato aniónico (RCOO-) del ácido aspártico o del ácido glutámico y el amonio catiónico (RNH3+< /sup>) de la lisina o el guanidinio (RNHC(NH2)2+) de la arginina (Figura 2). Aunque estos son los más comunes, también pueden participar otros residuos con cadenas laterales ionizables como histidina, tirosina y serina, dependiendo de factores externos que perturben su pKa&#. 39;s. También se considera importante la distancia entre los residuos que participan en el puente salino. La distancia requerida es inferior a 4 Å (400 pm). Los aminoácidos que están separados por más de esta distancia no califican como formadores de un puente salino. Debido a las numerosas cadenas laterales ionizables de aminoácidos que se encuentran en toda una proteína, el pH al que se coloca una proteína es crucial para su estabilidad.
Puentes salinos encontrados en complejos proteína-ligando
También se pueden formar puentes salinos entre una proteína y ligandos de moléculas pequeñas. Se descubrió que más de 1100 complejos proteína-ligando únicos del Protein Databank forman puentes salinos con sus objetivos proteicos, lo que indica que los puentes salinos son frecuentes en la interacción fármaco-proteína. Estos contienen estructuras de diferentes clases de enzimas, incluidas hidrolasas, transferasas, quinasas, reductasa, oxidorreductasa, liasas y receptores acoplados a proteína G (GPCR). Estudios recientes han demostrado que los puentes salinos desempeñan un papel importante en la estabilización de los complejos proteína-ligando al actuar como clips moleculares que estabilizan la conformación de la proteína y pueden aumentar la actividad inhibidora en cientos de veces. Por lo tanto, la incorporación de puentes salinos entre los ligandos y sus proteínas dianas podría ser una herramienta valiosa en el diseño racional de fármacos.
Métodos para cuantificar la estabilidad del puente salino en proteínas

La contribución de un puente salino a la estabilidad general del estado plegado de una proteína se puede evaluar a través de datos termodinámicos recopilados de estudios de mutagénesis y técnicas de resonancia magnética nuclear. Usando una proteína mutada de tipo pseudo-salvaje específicamente mutada para evitar la precipitación a pH alto, la contribución del puente salino a la energía libre general del estado de la proteína plegada se puede determinar realizando una mutación puntual, alterando y, en consecuencia, rompiendo la sal. puente. Por ejemplo, se identificó la existencia de un puente salino en la lisozima T4 entre el ácido aspártico (Asp) en el residuo 70 y una histidina (His) en el residuo 31 (Figura 3). Se realizó mutagénesis dirigida con asparagina (Asn) (Figura 4) obteniendo tres nuevos mutantes: Asp70Asn His31 (Mutante 1), Asp70 His31Asn (Mutante 2) y Asp70Asn His31Asn (Doble Mutante).

Una vez que se han establecido los mutantes, se pueden emplear dos métodos para calcular la energía libre asociada con un puente salino. Un método implica la observación de la temperatura de fusión de la proteína de tipo salvaje frente a la de los tres mutantes. La desnaturalización se puede controlar mediante un cambio en el dicroísmo circular. Una reducción en la temperatura de fusión indica una reducción en la estabilidad. Esto se cuantifica mediante un método descrito por Becktel y Schellman donde la diferencia de energía libre entre ambos se calcula mediante ΔTΔS. Existen algunos problemas con este cálculo y solo se puede utilizar con datos muy precisos. En el ejemplo de lisozima T4, el ΔS del tipo pseudosalvaje se había informado previamente a pH 5,5, por lo que la diferencia de temperatura del punto medio de 11 °C a este pH se multiplicó por el ΔS informado. de 360 cal/(mol·K) (1,5 kJ/(mol·K)) produce un cambio de energía libre de aproximadamente −4 kcal/mol (−17 kJ/mol). Este valor corresponde a la cantidad de energía libre aportada a la estabilidad de la proteína por el puente salino.
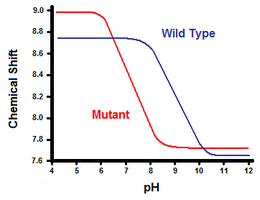
El segundo método utiliza espectroscopia de resonancia magnética nuclear para calcular la energía libre del puente salino. Se realiza una titulación, mientras se registra el desplazamiento químico correspondiente a los protones del carbono adyacente al grupo carboxilato o amonio. El punto medio de la curva de titulación corresponde al pKa, o el pH donde la proporción de moléculas protonadas: desprotonadas es 1:1. Siguiendo con el ejemplo de la lisozima T4, se obtiene una curva de titulación mediante la observación de un cambio en el protón C2 de la histidina 31 (Figura 5). La Figura 5 muestra el cambio en la curva de titulación entre el tipo salvaje y el mutante en el que Asp70 es Asn. El puente salino formado se encuentra entre el Asp70 desprotonado y el His31 protonado. Esta interacción provoca el cambio observado en pKa de His31. En la proteína de tipo salvaje desplegada, donde el puente salino está ausente, se informa que His31 tiene un pKa de 6,8 en H2O. tampones de fuerza iónica moderada. La Figura 5 muestra un pKa del tipo salvaje de 9,05. Esta diferencia en pKa está respaldada por la interacción de His31 con Asp70. Para mantener el puente salino, His31 intentará conservar su protón el mayor tiempo posible. Cuando se rompe el puente salino, como en el mutante D70N, el pKa vuelve a un valor de 6,9, mucho más cercano al de His31 en el estado desplegado.
La diferencia en pKa se puede cuantificar para reflejar la contribución del puente de sal a la energía libre. Usando la energía gratuita de Gibbs: ΔG = −RT In...Keq), donde R es la constante de gas universal, T es la temperatura en kelvins, y Keq es la constante del equilibrio de una reacción en el equilibrio. La deprotonación de His31 es una reacción de equilibrio ácido con una reacción especial Keq conocido como la constante de disociación de ácido, Ka: His31-H+ ⇌ His31 + H+. La pKa entonces está relacionado con Ka por el siguiente: pKa = −log(Ka). Cálculo de la diferencia de energía libre del mutante y del tipo salvaje ahora se puede hacer utilizando la ecuación de energía libre, la definición de pKa, la p observadaKa valores, y la relación entre logaritmos naturales y logaritmos. En el ejemplo de lysozyme T4, este enfoque dio una contribución calculada de aproximadamente 3 kcal/mol a la energía libre general. Se puede adoptar un enfoque similar con el otro participante en el puente de sal, como el Asp70 en el ejemplo T4 lysozyme, monitoreando su cambio en pKa después de la mutación de His31.
Una palabra de precaución al elegir el experimento apropiado implica la ubicación del puente salino dentro de la proteína. El entorno juega un papel importante en la interacción. Con fuerzas iónicas elevadas, el puente salino puede quedar completamente enmascarado, ya que se trata de una interacción electrostática. El puente salino His31-Asp70 en la lisozima T4 estaba enterrado dentro de la proteína. La entropía juega un papel más importante en los puentes salinos superficiales donde los residuos que normalmente tienen la capacidad de moverse están restringidos por su interacción electrostática y enlaces de hidrógeno. Se ha demostrado que esto disminuye la entropía lo suficiente como para casi borrar la contribución de la interacción. Los puentes salinos superficiales se pueden estudiar de manera similar a los puentes salinos enterrados, empleando ciclos dobles mutantes y valoraciones de RMN. Aunque existen casos en los que los puentes de sal enterrados contribuyen a la estabilidad, como cualquier otra cosa, existen excepciones y los puentes de sal enterrados pueden mostrar un efecto desestabilizador. Además, los puentes salinos superficiales, bajo ciertas condiciones, pueden mostrar un efecto estabilizador. El efecto estabilizador o desestabilizador debe evaluarse caso por caso y pocas declaraciones generales pueden hacerse.
Química supramolecular


La química supramolecular es un campo que se ocupa de las interacciones no covalentes entre macromoléculas. Los químicos de este campo han utilizado los puentes salinos de formas diversas y creativas, incluida la detección de aniones, la síntesis de cápsulas moleculares y polímeros de doble hélice.
Complejación aniónica
Las principales contribuciones de la química supramolecular se han dedicado al reconocimiento y detección de aniones. El emparejamiento iónico es la fuerza impulsora más importante para la complejación de aniones, pero la selectividad, p. dentro de la serie de haluros se ha logrado, principalmente mediante contribuciones de enlaces de hidrógeno.
Cápsulas moleculares
Las cápsulas moleculares son estructuras químicas diseñadas para capturar y retener una molécula huésped (ver encapsulación molecular). Szumna y sus compañeros desarrollaron una nueva cápsula molecular con un interior quiral. Esta cápsula está formada por dos mitades, como un huevo de Pascua de plástico (Figura 6). Las interacciones del puente salino entre las dos mitades hacen que se autoensamblen en solución (Figura 7). Son estables incluso cuando se calientan a 60 °C.
Polímeros de doble hélice
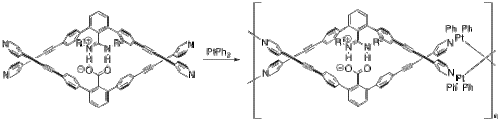
Yashima y sus compañeros han utilizado puentes salinos para construir varios polímeros que adoptan una conformación de doble hélice muy parecida al ADN. En un ejemplo, incorporaron platino para crear un metalopolímero de doble hélice. A partir de su monómero y bifenilo de platino (II) (Figura 8), su metalopolímero se autoensambla a través de una serie de reacciones de intercambio de ligandos. Las dos mitades del monómero están ancladas entre sí a través del puente salino entre el carboxilato desprotonado y los nitrógenos protonados.