Proteína fluorescente verde
La proteína fluorescente verde (GFP) es una proteína que exhibe una fluorescencia verde brillante cuando se expone a la luz en el rango azul a ultravioleta. La etiqueta GFP se refiere tradicionalmente a la proteína aislada por primera vez de la medusa Aequorea victoria y, en ocasiones, se denomina avGFP. Sin embargo, se han encontrado GFP en otros organismos, incluidos corales, anémonas de mar, zoanítidos, copépodos y lancetas.
La GFP de A. victoria tiene un pico de excitación mayor a una longitud de onda de 395 nm y uno menor a 475 nm. Su pico de emisión está en 509 nm, que se encuentra en la porción verde inferior del espectro visible. El rendimiento cuántico de fluorescencia (QY) de GFP es 0,79. La GFP del pensamiento de mar (Renilla reniformis) tiene un único pico de excitación principal a 498 nm. GFP es una herramienta excelente en muchas formas de biología debido a su capacidad para formar un cromóforo interno sin requerir cofactores accesorios, productos genéticos o enzimas/sustratos que no sean oxígeno molecular.
En biología celular y molecular, el gen GFP se usa con frecuencia como indicador de expresión. Se ha utilizado en formas modificadas para fabricar biosensores, y se han creado muchos animales que expresan GFP, lo que demuestra una prueba de concepto de que un gen se puede expresar en un organismo determinado, en órganos seleccionados o en células de interés. La GFP puede introducirse en animales u otras especies a través de técnicas transgénicas y mantenerse en su genoma y en el de su descendencia. Hasta la fecha, GFP se ha expresado en muchas especies, incluidas bacterias, levaduras, hongos, peces y mamíferos, incluso en células humanas. Los científicos Roger Y. Tsien, Osamu Shimomura y Martin Chalfie recibieron el Premio Nobel de Química 2008 el 10 de octubre de 2008 por su descubrimiento y desarrollo de la proteína fluorescente verde.
La mayoría de los genes disponibles comercialmente para GFP y proteínas fluorescentes similares tienen una longitud de alrededor de 730 pares de bases. La proteína natural tiene 238 aminoácidos. Su masa molecular es de 27 kD. Por lo tanto, fusionar el gen GFP con el gen de una proteína de interés puede aumentar significativamente el tamaño y la masa molecular de la proteína, y puede afectar la función natural de la proteína o cambiar su ubicación o trayectoria de transporte dentro de la célula..
Antecedentes
GFP de tipo salvaje (wtGFP)
En las décadas de 1960 y 1970, la GFP, junto con la proteína luminiscente separada aequorina (una enzima que cataliza la descomposición de la luciferina y libera luz), se purificó por primera vez a partir de la medusa Aequorea victoria y sus propiedades estudiado por Osamu Shimomura. En A. victoria, la fluorescencia de GFP ocurre cuando la aecuorina interactúa con los iones Ca2+, lo que induce un brillo azul. Parte de esta energía luminiscente se transfiere a la GFP, cambiando el color general hacia el verde. Sin embargo, su utilidad como herramienta para los biólogos moleculares no comenzó a darse cuenta hasta 1992 cuando Douglas Prasher informó sobre la clonación y la secuencia de nucleótidos de wtGFP en Gene. Los fondos para este proyecto se habían agotado, por lo que Prasher envió muestras de ADNc a varios laboratorios. El laboratorio de Martin Chalfie expresó la secuencia codificante de wtGFP, con los primeros aminoácidos eliminados, en células heterólogas de E. coli y C. elegans, publicando los resultados en Science en 1994. El laboratorio de Frederick Tsuji informó de forma independiente la expresión de la proteína recombinante un mes después. Sorprendentemente, la molécula de GFP se plegó y fue fluorescente a temperatura ambiente, sin necesidad de cofactores exógenos específicos de las medusas. Aunque esta GFP cercana a la wt era fluorescente, tenía varios inconvenientes, incluidos espectros de excitación de doble pico, sensibilidad al pH, sensibilidad al cloruro, rendimiento cuántico de fluorescencia deficiente, fotoestabilidad deficiente y plegamiento deficiente a 37 °C (99 °F).
La primera estructura cristalina informada de una GFP fue la del mutante S65T por el grupo de Remington en Science en 1996. Un mes después, el grupo de Phillips informó de forma independiente la estructura de GFP de tipo salvaje en Biotecnología de la Naturaleza. Estas estructuras cristalinas proporcionaron antecedentes vitales sobre la formación de cromóforos y las interacciones de residuos vecinos. Los investigadores han modificado estos residuos mediante mutagénesis aleatoria y dirigida para producir la amplia variedad de derivados de GFP que se utilizan en la actualidad. Investigaciones adicionales sobre GFP han demostrado que es resistente a detergentes, proteasas, tratamientos con cloruro de guanidinio (GdmCl) y cambios drásticos de temperatura.
Derivados de GFP
Debido al potencial de uso generalizado y las necesidades cambiantes de los investigadores, se han diseñado muchos mutantes diferentes de GFP. La primera mejora importante fue una mutación puntual única (S65T) informada en 1995 en Nature por Roger Tsien. Esta mutación mejoró drásticamente las características espectrales de GFP, lo que resultó en un aumento de la fluorescencia, la fotoestabilidad y un cambio del pico de excitación principal a 488 nm, con la emisión máxima mantenida en 509 nm. Esto coincidió con las características espectrales de los conjuntos de filtros FITC comúnmente disponibles, aumentando la practicidad de uso por parte del investigador general. En 1995, los laboratorios de Thastrup y Falkow descubrieron un punto mutante de eficiencia de plegado a 37 °C (F64L) para este andamio, que produce GFP mejorada (EGFP). EGFP permitió el uso práctico de GFP en células de mamíferos. EGFP tiene un coeficiente de extinción (denotado ε) de 55 000 M−1cm−1. El rendimiento cuántico de fluorescencia (QY) de EGFP es 0,60. El brillo relativo, expresado como ε•QY, es 33 000 M−1cm−1.
Superfolder GFP (sfGFP), una serie de mutaciones que permiten que GFP se pliegue y madure rápidamente incluso cuando se fusiona con péptidos que se pliegan mal, se informó en 2006.
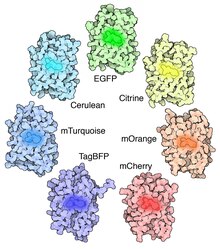
Se han realizado muchas otras mutaciones, incluidos mutantes de color; en particular, proteína fluorescente azul (EBFP, EBFP2, Azurite, mKalama1), proteína fluorescente cian (ECFP, Cerulean, CyPet, mTurquoise2) y derivados de proteína fluorescente amarilla (YFP, Citrine, Venus, YPet). Los derivados de BFP (excepto mKalama1) contienen la sustitución Y66H. Presentan una amplia banda de absorción en el ultravioleta centrada cerca de los 380 nanómetros y un máximo de emisión de 448 nanómetros. Se ha desarrollado una proteína mutante verde fluorescente (BFPms1) que se une preferentemente a Zn(II) y Cu(II). BFPms1 tiene varias mutaciones importantes, incluido el cromóforo BFP (Y66H), Y145F para un mayor rendimiento cuántico, H148G para crear un agujero en el barril beta y varias otras mutaciones que aumentan la solubilidad. La unión de Zn(II) aumenta la intensidad de la fluorescencia, mientras que la unión de Cu(II) apaga la fluorescencia y cambia el máximo de absorbancia de 379 a 444 nm. Por lo tanto, pueden usarse como biosensores de Zn.
Unión de cromóforos. La mutación crítica en los derivados del cian es la sustitución Y66W, que hace que el cromóforo se forme con un componente de indol en lugar de fenol. Se requieren varias mutaciones compensatorias adicionales en el barril circundante para restaurar el brillo de este cromóforo modificado debido al aumento del volumen del grupo indol. En ECFP y Cerulean, la mitad N-terminal de la séptima cadena exhibe dos conformaciones. Ambas conformaciones tienen un conjunto complejo de interacciones de van der Waals con el cromóforo. Las mutaciones Y145A y H148D en Cerulean estabilizan estas interacciones y permiten que el cromóforo sea más plano, mejor empaquetado y menos propenso a la extinción por colisión.
La mutagénesis aleatoria dirigida al sitio adicional en combinación con la detección basada en la vida útil de la fluorescencia ha estabilizado aún más la séptima hebra β, lo que da como resultado una variante brillante, mTurquoise2, con un rendimiento cuántico (QY) de 0,93. La longitud de onda desplazada hacia el rojo de los derivados de YFP se logra mediante la mutación T203Y y se debe a las interacciones de apilamiento de electrones π entre el residuo de tirosina sustituido y el cromóforo. Estas dos clases de variantes espectrales se emplean a menudo para los experimentos de transferencia de energía de resonancia de Förster (FRET). Los reporteros FRET codificados genéticamente sensibles a las moléculas de señalización celular, como el calcio o el glutamato, el estado de fosforilación de proteínas, la complementación de proteínas, la dimerización de receptores y otros procesos proporcionan lecturas ópticas altamente específicas de la actividad celular en tiempo real.
La mutagénesis semiracional de una serie de residuos condujo a mutantes sensibles al pH conocidos como pHluorinas y, más tarde, pHluorinas supereclípticas. Al explotar el cambio rápido en el pH tras la fusión de vesículas sinápticas, las pHluorinas etiquetadas con sinaptobrevina se han utilizado para visualizar la actividad sináptica en las neuronas.
La GFP sensible a redox (roGFP) se diseñó mediante la introducción de cisteínas en la estructura de barril beta. El estado redox de las cisteínas determina las propiedades fluorescentes de roGFP.
Nomenclatura
La nomenclatura de las GFP modificadas suele ser confusa debido a la superposición de asignaciones de varias versiones de GFP en un solo nombre. Por ejemplo, mGFP a menudo se refiere a una GFP con una palmitoilación N-terminal que hace que la GFP se una a las membranas celulares. Sin embargo, el mismo término también se usa para referirse a la GFP monomérica, que a menudo se logra mediante la mutación A206K que rompe la interfaz del dímero. La GFP de tipo salvaje tiene una débil tendencia a la dimerización en concentraciones superiores a 5 mg/mL. mGFP también significa "GFP modificado" que ha sido optimizado a través del intercambio de aminoácidos para una expresión estable en células vegetales.
En la naturaleza
Se desconoce el propósito tanto de la bioluminiscencia (primaria) (de la acción de la aecuorina sobre la luciferina) como de la fluorescencia (secundaria) de GFP en las medusas. GFP se coexpresa con aecuorina en pequeños gránulos alrededor del borde de la campana de la medusa. El pico de excitación secundario (480 nm) de GFP absorbe parte de la emisión azul de aecuorina, lo que le da a la bioluminiscencia un tono más verde. El residuo de serina 65 del cromóforo GFP es responsable de los espectros de excitación de doble pico de GFP de tipo salvaje. Se conserva en las tres isoformas de GFP originalmente clonadas por Prasher. Casi todas las mutaciones de este residuo consolidan los espectros de excitación en un solo pico a 395 nm o 480 nm. El mecanismo preciso de esta sensibilidad es complejo, pero, al parecer, implica la donación de un hidrógeno de la serina 65 al glutamato 222, que influye en la ionización del cromóforo. Dado que una sola mutación puede mejorar drásticamente el pico de excitación de 480 nm, lo que convierte a GFP en un socio mucho más eficiente de la aecuorina, A. victoria parece preferir evolutivamente el espectro de excitación de doble pico menos eficiente. Roger Tsien ha especulado que la variación de la presión hidrostática con la profundidad puede afectar la capacidad de la serina 65 para donar hidrógeno al cromóforo y cambiar la proporción de los dos picos de excitación. Así, la medusa puede cambiar el color de su bioluminiscencia con la profundidad. Sin embargo, un colapso en la población de medusas en Friday Harbor, donde se descubrió originalmente GFP, ha obstaculizado un mayor estudio del papel de GFP en el entorno natural de las medusas.
Se sabe que la mayoría de las especies de lancetas producen GFP en varias regiones de su cuerpo. A diferencia de A. victoria, las lancetas no producen su propia luz azul y aún se desconoce el origen de su GFP endógena. Algunos especulan que atrae plancton hacia la boca de la lanceta, sirviendo como un mecanismo de caza pasivo. También puede servir como agente fotoprotector en las larvas, previniendo el daño causado por la luz azul de alta intensidad al convertirla en luz verde de menor intensidad. Sin embargo, estas teorías no han sido probadas.
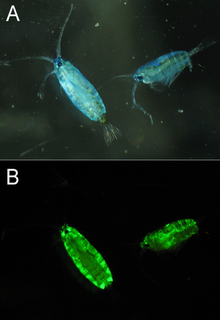
Se han encontrado proteínas similares a GFP en múltiples especies de copépodos marinos, particularmente de las familias Pontellidae y Aetideidae. La GFP aislada de Pontella mimocerami ha mostrado altos niveles de brillo con un rendimiento cuántico de 0,92, lo que la hace casi dos veces más brillante que la EGFP de uso común aislada de A. victoria.
Otras proteínas fluorescentes
Hay muchas proteínas similares a GFP que, a pesar de pertenecer a la misma familia de proteínas que GFP, no se derivan directamente de Aequorea victoria. Estos incluyen dsRed, eqFP611, Dronpa, TagRFPs, KFP, EosFP/IrisFP, Dendra, etc. Habiendo sido desarrolladas a partir de proteínas en diferentes organismos, estas proteínas a veces pueden mostrar enfoques inesperados para la formación de cromóforos. Algunos de estos, como KFP, se desarrollan a partir de proteínas naturalmente no fluorescentes o débilmente fluorescentes para ser mejorados en gran medida mediante mutagénesis. Cuando se usan barriles similares a GFP de diferentes características espectrales, los espectros de excitación de un cromóforo se pueden usar para alimentar otro cromóforo (FRET), lo que permite la conversión entre longitudes de onda de luz.
Las proteínas fluorescentes de unión a FMN (FbFP) se desarrollaron en 2007 y son una clase de proteínas fluorescentes pequeñas (11-16 kDa) independientes del oxígeno que se derivan de los receptores de luz azul. Están destinados especialmente al uso en condiciones anaeróbicas o hipóxicas, ya que la formación y unión del cromóforo Flavin no requiere oxígeno molecular, como sí ocurre con la síntesis del cromóforo GFP.
Las proteínas fluorescentes con otros cromóforos, como UnaG con bilirrubina, pueden mostrar propiedades únicas, como la emisión desplazada hacia el rojo por encima de 600 nm o la fotoconversión de un estado emisor de luz verde a un estado emisor de luz roja. Pueden tener longitudes de onda de excitación y emisión lo suficientemente separadas como para lograr la conversión entre luz roja y verde.
Una nueva clase de proteína fluorescente evolucionó a partir de una ficobiliproteína cianobacteriana (Trichodesmium erythraeum), α-aloficocianina, y se denominó proteína fluorescente ultra roja pequeña (smURFP) en 2016. smURFP autocatalíticamente autoincorpora la cromóforo biliverdina sin necesidad de una proteína externa, conocida como liasa. Las proteínas similares a GFP derivadas de medusas y corales requieren oxígeno y producen una cantidad estequiométrica de peróxido de hidrógeno tras la formación de cromóforos. smURFP no requiere oxígeno ni produce peróxido de hidrógeno y utiliza el cromóforo biliverdina. smURFP tiene un gran coeficiente de extinción (180 000 M−1 cm−1) y tiene un rendimiento cuántico modesto (0,20), lo que lo hace comparable con el brillo biofísico de eGFP y ~2 - veces más brillante que la mayoría de las proteínas fluorescentes rojas o de color rojo lejano derivadas del coral. Las propiedades espectrales de smURFP son similares a las del colorante orgánico Cy5.
Las reseñas sobre nuevas clases de proteínas fluorescentes y sus aplicaciones se pueden encontrar en las reseñas citadas.
Estructura
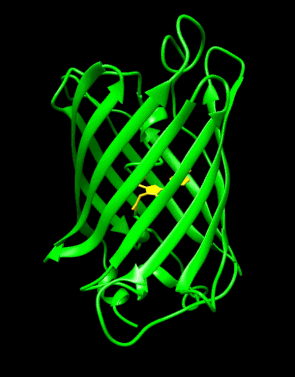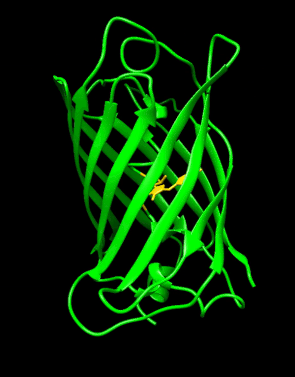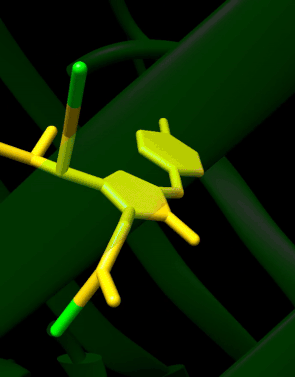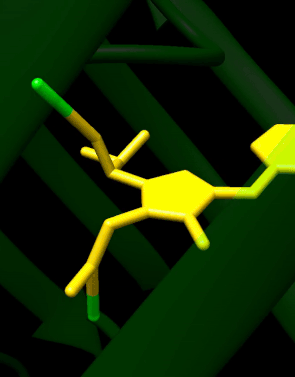
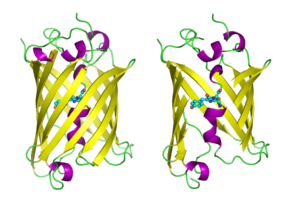
GFP tiene una estructura de barril beta que consta de once hebras β con una disposición de hoja plegada, con una hélice alfa que contiene el cromóforo 4-(p-hidroxibencilideno)imidazolidin-5-ona (HBI) corriendo por el centro. Cinco hélices alfa más cortas forman tapas en los extremos de la estructura. La estructura de barril beta es un cilindro casi perfecto, de 42 Å de largo y 24 Å de diámetro (algunos estudios han informado un diámetro de 30 Å), lo que crea lo que se conoce como un "β-can" formación, que es exclusiva de la familia tipo GFP. HBI, la forma modificada espontáneamente del tripéptido Ser65–Tyr66–Gly67, no es fluorescente en ausencia del andamio GFP correctamente plegado y existe principalmente en forma de fenol no ionizado en wtGFP. Las cadenas laterales del barril que miran hacia adentro inducen reacciones de ciclación específicas en Ser65–Tyr66–Gly67 que inducen la ionización de HBI a la forma de fenolato y la formación de cromóforos. Este proceso de modificación postraduccional se denomina maduración. La red de enlaces de hidrógeno y las interacciones de apilamiento de electrones con estas cadenas laterales influyen en el color, la intensidad y la fotoestabilidad de GFP y sus numerosos derivados. La naturaleza estrechamente empaquetada del cilindro excluye las moléculas de solvente, protegiendo la fluorescencia del cromóforo de la extinción por el agua. Además de la autociclación de Ser65-Tyr66-Gly67, se produce una reacción de 1,2-deshidrogenación en el residuo Tyr66. Además de los tres residuos que forman el cromóforo, residuos como Gln94, Arg96, His148, Thr203 y Glu222 actúan como estabilizadores. Los residuos de Gln94, Arg96 e His148 pueden estabilizarse deslocalizando la carga del cromóforo. Arg96 es el residuo estabilizador más importante debido al hecho de que provoca que se produzcan los realineamientos estructurales necesarios del anillo HBI. Cualquier mutación en el residuo Arg96 daría como resultado una disminución en la tasa de desarrollo del cromóforo porque se perderían las interacciones electrostáticas y estéricas adecuadas. Tyr66 es el receptor de los enlaces de hidrógeno y no se ioniza para producir una electrostática favorable.
Formación autocatalítica del cromóforo en wtGFP
Desde el punto de vista mecánico, el proceso implica una ciclación mediada por una base seguida de deshidratación y oxidación. En la reacción de 7a a 8 se produce la formación de una enamina a partir de la imina, mientras que en la reacción de 7b a 9 se extrae un protón. El fluoróforo HBI formado se resalta en verde.
Las reacciones son catalizadas por los residuos Glu222 y Arg96. También es posible un mecanismo análogo con treonina en lugar de Ser65.
Aplicaciones
Ensayos de reportera
(feminine)La proteína fluorescente verde se puede utilizar como gen informador.
Por ejemplo, GFP se puede utilizar como indicador de los niveles de toxicidad ambiental. Se ha demostrado que esta proteína es una forma eficaz de medir los niveles de toxicidad de varios productos químicos, incluidos el etanol, el p-formaldehído, el fenol, el triclosán y el parabeno. La GFP es excelente como proteína informadora porque no tiene ningún efecto en el huésped cuando se introduce en el entorno celular del huésped. Debido a esta capacidad, no se necesita tinción de visualización externa, ATP o cofactores. Con respecto a los niveles de contaminantes, se midió la fluorescencia para medir el efecto que los contaminantes tienen sobre la célula huésped. También se midió la densidad celular de la célula huésped. Los resultados del estudio realizado por Song, Kim, & Seo (2016) mostró que hubo una disminución tanto en la fluorescencia como en la densidad celular a medida que aumentaron los niveles de contaminantes. Esto era indicativo del hecho de que la actividad celular había disminuido. Más investigación sobre esta aplicación específica para determinar el mecanismo por el cual la GFP actúa como marcador de contaminantes. Se han observado resultados similares en el pez cebra porque el pez cebra al que se le inyectó GFP fue aproximadamente veinte veces más susceptible de reconocer el estrés celular que el pez cebra al que no se le inyectó GFP.
Ventajas
La mayor ventaja de GFP es que puede ser heredable, dependiendo de cómo se introdujo, lo que permite un estudio continuo de las células y los tejidos en los que se expresa. La visualización de GFP no es invasiva y solo requiere iluminación con luz azul. La GFP por sí sola no interfiere con los procesos biológicos, pero cuando se fusiona con proteínas de interés, se requiere un diseño cuidadoso de los enlazadores para mantener la función de la proteína de interés. Además, si se usa con un monómero, puede difundirse fácilmente por todas las células.
Microscopía de fluorescencia
La disponibilidad de GFP y sus derivados ha redefinido completamente la microscopía de fluorescencia y la forma en que se utiliza en biología celular y otras disciplinas biológicas. Mientras que la mayoría de las moléculas fluorescentes pequeñas como FITC (isotiocianato de fluoresceína) son fuertemente fototóxicas cuando se usan en células vivas, las proteínas fluorescentes como GFP suelen ser mucho menos dañinas cuando se iluminan en células vivas. Esto ha desencadenado el desarrollo de sistemas de microscopía de fluorescencia de células vivas altamente automatizados, que se pueden usar para observar células a lo largo del tiempo que expresan una o más proteínas etiquetadas con proteínas fluorescentes.
Existen muchas técnicas para utilizar GFP en un experimento de obtención de imágenes de células vivas. La forma más directa de utilizar GFP es unirlo directamente a una proteína de interés. Por ejemplo, GFP se puede incluir en un plásmido que expresa otros genes para indicar una transfección exitosa de un gen de interés. Otro método es utilizar una GFP que contenga una mutación en la que la fluorescencia cambiará de verde a amarillo con el tiempo, lo que se conoce como temporizador fluorescente. Con el temporizador fluorescente, los investigadores pueden estudiar el estado de la producción de proteínas, como activación reciente, activación continua o desactivación reciente, según el color informado por la proteína fluorescente. En otro ejemplo más, los científicos han modificado la GFP para que se active solo después de la exposición a la irradiación, lo que brinda a los investigadores una herramienta para activar selectivamente ciertas partes de una célula y observar dónde se mueven las proteínas etiquetadas con la GFP desde la ubicación inicial. Estos son solo dos ejemplos en un campo floreciente de microcopia fluorescente y aquí se puede encontrar una revisión más completa de los biosensores que utilizan GFP y otras proteínas fluorescentes.
Por ejemplo, GFP ha sido ampliamente utilizado en el etiquetado de los espermatozoides de varios organismos con fines de identificación como en Drosophila melanogaster, donde la expresión de GFP se puede utilizar como marcador de una característica particular. GFP también se puede expresar en diferentes estructuras que permiten la distinción morfológica. En tales casos, el gen para la producción de GFP se incorpora al genoma del organismo en la región del ADN que codifica para las proteínas diana y que está controlada por la misma secuencia reguladora; es decir, la secuencia reguladora del gen ahora controla la producción de GFP, además de la(s) proteína(s) marcada(s). En las células donde se expresa el gen y se producen las proteínas etiquetadas, se produce GFP al mismo tiempo. Por lo tanto, sólo aquellas células en las que se expresa el gen marcado, o se producen las proteínas diana, serán fluorescentes cuando se observen bajo microscopía de fluorescencia. El análisis de tales películas de lapso de tiempo ha redefinido la comprensión de muchos procesos biológicos, incluido el plegamiento de proteínas, el transporte de proteínas y la dinámica del ARN, que en el pasado se habían estudiado utilizando material fijo (es decir, muerto). Los datos obtenidos también se utilizan para calibrar modelos matemáticos de sistemas intracelulares y para estimar tasas de expresión génica. De manera similar, GFP se puede usar como indicador de la expresión de proteínas en sistemas heterólogos. En este escenario, las proteínas de fusión que contienen GFP se introducen indirectamente, utilizando el ARN de la construcción, o directamente, con la propia proteína etiquetada. Este método es útil para estudiar las características estructurales y funcionales de la proteína etiquetada en una escala macromolecular o de una sola molécula con microscopía de fluorescencia.
El microscopio Vertico SMI que utiliza la tecnología SPDM Phymod utiliza el llamado "fotoblanqueo reversible" efecto de tintes fluorescentes como GFP y sus derivados para localizarlos como moléculas individuales en una resolución óptica de 10 nm. Esto también se puede realizar como una co-localización de dos derivados de GFP (2CLM).
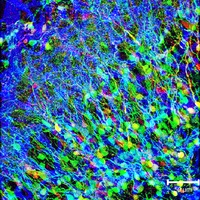
Otro poderoso uso de GFP es expresar la proteína en pequeños conjuntos de células específicas. Esto permite a los investigadores detectar ópticamente tipos específicos de células in vitro (en una placa), o incluso in vivo (en el organismo vivo). Combinar genéticamente varias variantes espectrales de GFP es un truco útil para el análisis de circuitos cerebrales (Brainbow). Otros usos interesantes de las proteínas fluorescentes en la literatura incluyen el uso de FP como sensores del potencial de la membrana neuronal, el seguimiento de los receptores AMPA en las membranas celulares, la entrada viral y la infección de virus de influenza individuales y virus lentivirales, etc.
También se ha descubierto que las nuevas líneas de ratas GFP transgénicas pueden ser relevantes para la terapia génica y la medicina regenerativa. Mediante el uso de "alta expresión" GFP, las ratas transgénicas muestran una alta expresión en la mayoría de los tejidos y muchas células que no se han caracterizado o se han caracterizado de forma deficiente en ratas transgénicas GFP anteriores.
Se ha demostrado que GFP es útil en criobiología como ensayo de viabilidad. La correlación de viabilidad medida mediante ensayos con azul de tripano fue de 0,97. Otra aplicación es el uso de co-transfección de GFP como control interno para la eficiencia de transfección en células de mamífero.
Un nuevo uso posible de GFP incluye usarlo como un monitor sensible de procesos intracelulares a través de un sistema láser eGFP hecho de una línea celular de riñón embrionario humano. El primer láser vivo diseñado está hecho por una célula que expresa eGFP dentro de una cavidad óptica reflectante y la golpea con pulsos de luz azul. En un cierto umbral de pulso, la salida óptica del eGFP se vuelve más brillante y completamente uniforme en color verde puro con una longitud de onda de 516 nm. Antes de ser emitida como luz láser, la luz rebota de un lado a otro dentro de la cavidad del resonador y pasa por la celda varias veces. Al estudiar los cambios en la actividad óptica, los investigadores pueden comprender mejor los procesos celulares.
GFP se usa ampliamente en la investigación del cáncer para etiquetar y rastrear células cancerosas. Las células cancerosas marcadas con GFP se han utilizado para modelar la metástasis, el proceso por el cual las células cancerosas se propagan a órganos distantes.
GFP dividido
GFP se puede utilizar para analizar la colocalización de proteínas. Esto se logra "dividiendo" la proteína en dos fragmentos que pueden autoensamblarse y luego fusionar cada uno de estos con las dos proteínas de interés. Solos, estos fragmentos de GFP incompletos no pueden emitir fluorescencia. Sin embargo, si las dos proteínas de interés se colocalizan, los dos fragmentos de GFP se ensamblan para formar una estructura similar a GFP que es capaz de emitir fluorescencia. Por lo tanto, midiendo el nivel de fluorescencia es posible determinar si las dos proteínas de interés se colocalizan.
Macro-fotografía
Los procesos biológicos a macroescala, como la propagación de infecciones por virus, se pueden seguir mediante el etiquetado GFP. En el pasado, la luz ultravioleta (UV) mutagénica se ha utilizado para iluminar organismos vivos (p. ej., ver) para detectar y fotografiar la expresión de GFP. Recientemente, se ha desarrollado una técnica que utiliza luces LED no mutagénicas para macrofotografía. La técnica utiliza un accesorio de cámara de epifluorescencia basado en el mismo principio utilizado en la construcción de microscopios de epifluorescencia.
Mascotas transgénicas
Alba, un conejo verde fluorescente, fue creado por un laboratorio francés encargado por Eduardo Kac utilizando GFP con fines artísticos y de comentario social. La empresa estadounidense Yorktown Technologies comercializa a las tiendas de acuarios el pez cebra verde fluorescente (GloFish) que se desarrolló inicialmente para detectar la contaminación en las vías fluviales. NeonPets, una empresa con sede en EE. UU., ha comercializado ratones fluorescentes verdes para la industria de las mascotas como NeonMice. Los cerdos fluorescentes verdes, conocidos como Noels, fueron criados por un grupo de investigadores dirigido por Wu Shinn-Chih en el Departamento de Ciencia y Tecnología Animal de la Universidad Nacional de Taiwán. Un equipo japonés-estadounidense creó gatos verdes fluorescentes como prueba de concepto para usarlos potencialmente como organismos modelo para enfermedades, particularmente el VIH. En 2009, un equipo surcoreano de la Universidad Nacional de Seúl crió los primeros beagles transgénicos con células de fibroblastos de anémonas de mar. Los perros emiten una luz fluorescente roja y están destinados a permitir que los científicos estudien los genes que causan enfermedades humanas como la narcolepsia y la ceguera.
Arte
Julian Voss-Andreae, un artista nacido en Alemania que se especializa en "esculturas de proteínas" creó esculturas basadas en la estructura de GFP, incluida la "Proteína fluorescente verde" de 1,70 m (5'6") de altura (2004) y la "Steel Jellyfish" de 1,40 m (4'7") de altura (2006). La última escultura se encuentra en el lugar del descubrimiento de GFP por parte de Shimomura en 1962, los Laboratorios Friday Harbor de la Universidad de Washington.
Contenido relacionado
Cavidad corporal
Deslizador de orejas rojas
Arthrobacter