Polimorfismo de longitud de fragmentos de restricción
En biología molecular, el polimorfismo de longitud de fragmentos de restricción (RFLP) es una técnica que explota variaciones en secuencias de ADN homólogas, conocidas como polimorfismos, para distinguir individuos, poblaciones, o especies o para señalar las ubicaciones de los genes dentro de una secuencia. El término puede referirse a un polimorfismo en sí mismo, detectado a través de las diferentes ubicaciones de los sitios de las enzimas de restricción, o a una técnica de laboratorio relacionada mediante la cual se pueden ilustrar tales diferencias. En el análisis RFLP, una o más enzimas de restricción digieren una muestra de ADN en fragmentos, y los fragmentos de restricción resultantes se separan luego mediante electroforesis en gel de acuerdo con su tamaño.
El análisis RFLP ahora está en gran medida obsoleto debido a la aparición de tecnologías económicas de secuenciación de ADN, pero fue la primera técnica de perfilado de ADN lo suficientemente económica como para ver una aplicación generalizada. El análisis RFLP fue una herramienta temprana importante en el mapeo del genoma, la localización de genes para trastornos genéticos, la determinación del riesgo de enfermedad y las pruebas de paternidad.
Análisis RFLP
La técnica básica para la detección de RFLP consiste en fragmentar una muestra de ADN con la aplicación de una enzima de restricción, que puede escindir selectivamente una molécula de ADN siempre que se reconozca una secuencia corta y específica en un proceso conocido como digestión de restricción. Los fragmentos de ADN producidos por la digestión se separan luego por longitud a través de un proceso conocido como electroforesis en gel de agarosa y se transfieren a una membrana mediante el procedimiento de transferencia Southern. La hibridación de la membrana con una sonda de ADN marcada determina entonces la longitud de los fragmentos que son complementarios a la sonda. Se dice que ocurre un polimorfismo de longitud de fragmento de restricción cuando la longitud de un fragmento detectado varía entre individuos, lo que indica homologías de secuencia no idénticas. Cada longitud de fragmento se considera un alelo, ya sea que contenga una región codificante o no, y puede usarse en análisis genéticos posteriores.
Ejemplos
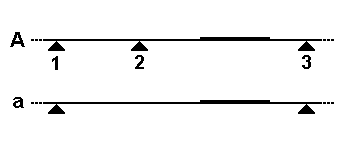
Hay dos mecanismos comunes por los cuales el tamaño de un fragmento de restricción en particular puede variar. En el primer esquema, una sonda de ADN detecta un pequeño segmento del genoma (línea más gruesa). En el alelo A, una enzima de restricción escinde el genoma en tres sitios cercanos (triángulos), pero la sonda solo detectará el fragmento más a la derecha. En el alelo a, el sitio de restricción 2 se ha perdido por una mutación, por lo que la sonda ahora detecta el fragmento fusionado más grande que va desde los sitios 1 a 3. El segundo diagrama muestra cómo se vería esta variación de tamaño de fragmento en un Southern blot, y cómo cada alelo (dos por individuo) puede ser heredado en los miembros de una familia.
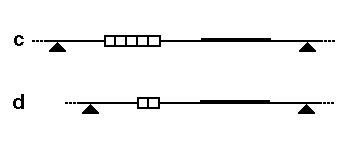
En el tercer esquema, la sonda y la enzima de restricción se eligen para detectar una región del genoma que incluye un segmento de repetición en tándem de número variable (VNTR) (cuadros en el diagrama esquemático). En el alelo c, hay cinco repeticiones en el VNTR y la sonda detecta un fragmento más largo entre los dos sitios de restricción. En el alelo d, solo hay dos repeticiones en el VNTR, por lo que la sonda detecta un fragmento más corto entre los mismos dos sitios de restricción. Otros procesos genéticos, como inserciones, deleciones, translocaciones e inversiones, también pueden dar lugar a polimorfismos. Las pruebas de RFLP requieren muestras de ADN mucho más grandes que las pruebas de repetición corta en tándem (STR).
Aplicaciones
Anteriormente, el análisis de la variación de RFLP en los genomas era una herramienta vital en el mapeo del genoma y el análisis de enfermedades genéticas. Si los investigadores intentaran determinar inicialmente la ubicación cromosómica de un gen de una enfermedad en particular, analizarían el ADN de los miembros de una familia afectada por la enfermedad y buscarían alelos RFLP que mostraran un patrón de herencia similar al de la enfermedad (ver enlace genético). Una vez que se localizó un gen de la enfermedad, el análisis RFLP de otras familias podría revelar quién estaba en riesgo de contraer la enfermedad o quién era probable que fuera portador de los genes mutantes. La prueba RFLP se utiliza en la identificación y diferenciación de organismos mediante el análisis de patrones únicos en el genoma. También se utiliza en la identificación de la tasa de recombinación en los loci entre sitios de restricción.
El análisis RFLP también fue la base de los primeros métodos de huellas dactilares genéticas, útiles en la identificación de muestras recuperadas de la escena del crimen, en la determinación de la paternidad y en la caracterización de la diversidad genética o patrones de reproducción en poblaciones animales.
Alternativas
Sin embargo, la técnica para el análisis de RFLP es lenta y engorrosa. Requiere una gran cantidad de muestra de ADN, y el proceso combinado de marcaje de sonda, fragmentación de ADN, electroforesis, transferencia, hibridación, lavado y autorradiografía puede tardar hasta un mes en completarse. En 1985 se informó sobre una versión limitada del método RFLP que usaba sondas de oligonucleótidos. Los resultados del Proyecto Genoma Humano han reemplazado en gran medida la necesidad del mapeo RFLP y la identificación de muchos polimorfismos de un solo nucleótido (SNP) en ese proyecto (así como como la identificación directa de muchos genes de enfermedades y mutaciones) ha reemplazado la necesidad de análisis de ligamiento de enfermedades RFLP (ver genotipificación de SNP). El análisis de los alelos VNTR continúa, pero ahora generalmente se realiza mediante métodos de reacción en cadena de la polimerasa (PCR). Por ejemplo, los protocolos estándar para la toma de huellas dactilares de ADN implican el análisis de PCR de paneles de más de una docena de VNTR.
RFLP todavía se usa en la selección asistida por marcadores. El polimorfismo de longitud de fragmentos de restricción terminal (TRFLP o, a veces, T-RFLP) es una técnica desarrollada inicialmente para caracterizar comunidades bacterianas en muestras de especies mixtas. La técnica también se ha aplicado a otros grupos, incluidos los hongos del suelo. TRFLP funciona mediante la amplificación por PCR del ADN utilizando pares de cebadores que han sido marcados con etiquetas fluorescentes. Los productos de PCR luego se digieren usando enzimas RFLP y los patrones resultantes se visualizan usando un secuenciador de ADN. Los resultados se analizan simplemente contando y comparando bandas o picos en el perfil de TRFLP, o haciendo coincidir las bandas de una o más ejecuciones de TRFLP con una base de datos de especies conocidas. La técnica es similar en algunos aspectos a la electroforesis en gel de gradiente de temperatura o gradiente desnaturalizante (TGGE y DGGE).
Los cambios de secuencia directamente relacionados con un RFLP también pueden analizarse más rápidamente mediante PCR. La amplificación puede dirigirse a través del sitio de restricción alterado y los productos digeridos con la enzima de restricción. Este método se ha denominado secuencia polimórfica amplificada escindida (CAPS). Alternativamente, el segmento amplificado se puede analizar mediante sondas de oligonucleótidos específicos de alelo (ASO), un proceso que a menudo se puede realizar mediante una simple transferencia de puntos.
Contenido relacionado
Sarcopterygii
Mesozoos
Ludwig de Bertalanffy