Poliadenilación

poliadenilación es la adición de una cola poli(A) a una transcripción de ARN, normalmente un ARN mensajero (ARNm). La cola poli(A) consta de múltiples monofosfatos de adenosina; en otras palabras, es un tramo de ARN que sólo tiene bases de adenina. En eucariotas, la poliadenilación es parte del proceso que produce ARNm maduro para la traducción. En muchas bacterias, la cola poli(A) promueve la degradación del ARNm. Por tanto, forma parte de un proceso más amplio de expresión génica.
El proceso de poliadenilación comienza cuando termina la transcripción de un gen. El segmento más 3′ del pre-ARNm recién creado se escinde primero mediante un conjunto de proteínas; Estas proteínas luego sintetizan la cola poli(A) en el extremo 3' del ARN. En algunos genes, estas proteínas añaden una cola poli(A) en uno de varios sitios posibles. Por lo tanto, la poliadenilación puede producir más de una transcripción de un solo gen (poliadenilación alternativa), similar al empalme alternativo.
La cola poli(A) es importante para la exportación nuclear, la traducción y la estabilidad del ARNm. La cola se acorta con el tiempo y, cuando es lo suficientemente corta, el ARNm se degrada enzimáticamente. Sin embargo, en algunos tipos de células, los ARNm con colas cortas de poli(A) se almacenan para su posterior activación mediante repoliadenilación en el citosol. Por el contrario, cuando ocurre la poliadenilación en bacterias, promueve la degradación del ARN. A veces, este también es el caso de los ARN no codificantes eucariotas.
Las moléculas de ARNm tanto en procariotas como en eucariotas tienen extremos 3' poliadenilados, con las colas poli(A) procarióticas generalmente más cortas y menos moléculas de ARNm poliadeniladas.
Antecedentes del ARN
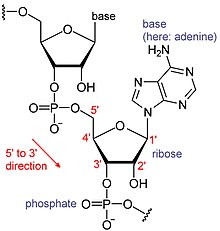
Los ARN son un tipo de moléculas biológicas grandes, cuyos componentes básicos individuales se denominan nucleótidos. El nombre cola poli(A) (para cola de ácido poliadenílico) refleja la forma en que se abrevian los nucleótidos de ARN, con una letra para la base que contiene el nucleótido (A para adenina, C para citosina, G para guanina y U de uracilo). Los ARN se producen (transcriben) a partir de una plantilla de ADN. Por convención, las secuencias de ARN se escriben en dirección 5′ a 3′. El extremo 5' es la parte de la molécula de ARN que se transcribe primero y el extremo 3' se transcribe al final. El extremo 3' es también donde se encuentra la cola poli(A) en los ARN poliadenilados.
El ARN mensajero (ARNm) es un ARN que tiene una región codificante que actúa como plantilla para la síntesis de proteínas (traducción). El resto del ARNm, las regiones no traducidas, ajustan la actividad del ARNm. También hay muchos ARN que no se traducen, llamados ARN no codificantes. Al igual que las regiones no traducidas, muchos de estos ARN no codificantes tienen funciones reguladoras.
Poliadenilación nuclear
Función
En la poliadenilación nuclear, se añade una cola poli(A) a un ARN al final de la transcripción. En los ARNm, la cola poli(A) protege la molécula de ARNm de la degradación enzimática en el citoplasma y ayuda en la terminación de la transcripción, la exportación del ARNm desde el núcleo y la traducción. Casi todos los ARNm de eucariotas están poliadenilados, con la excepción de los ARNm de histonas dependientes de la replicación animal. Estos son los únicos ARNm en eucariotas que carecen de una cola poli(A), y que terminan en una estructura de tallo-bucle seguida de una secuencia rica en purinas, denominada elemento histona aguas abajo, que indica dónde se corta el ARN para que el extremo 3' Se forma el ARNm de la histona.
Muchos ARN no codificantes eucarióticos siempre están poliadenilados al final de la transcripción. Hay ARN pequeños donde la cola poli(A) se ve sólo en formas intermedias y no en el ARN maduro, ya que los extremos se eliminan durante el procesamiento, siendo los más notables los microARN. Pero, para muchos ARN largos no codificantes (un grupo aparentemente grande de ARN reguladores que, por ejemplo, incluye el ARN Xist, que media la inactivación del cromosoma X), una cola poli(A) es parte del ARN maduro.
Mecanismo
| Proteínas implicadas: CPSF: factor de especificidad de la división/polyadenylation |
El complejo de poliadenilación procesiva en el núcleo de los eucariotas actúa sobre productos de la ARN polimerasa II, como el ARNm precursor. Aquí, un complejo multiproteico (ver componentes a la derecha) escinde la parte más 3′ de un ARN recién producido y poliadenila el extremo producido por esta escisión. La escisión es catalizada por la enzima CPSF y ocurre entre 10 y 30 nucleótidos aguas abajo de su sitio de unión. Este sitio a menudo tiene la secuencia señal de poliadenilación AAUAAA en el ARN, pero existen variantes de la misma que se unen más débilmente a CPSF. Otras dos proteínas añaden especificidad a la unión a un ARN: CstF y CFI. CstF se une a una región rica en GU más abajo del sitio de CPSF. CFI reconoce un tercer sitio en el ARN (un conjunto de secuencias UGUAA en mamíferos) y puede reclutar CPSF incluso si falta la secuencia AAUAAA. La señal de poliadenilación (el motivo de secuencia reconocido por el complejo de escisión del ARN) varía entre grupos de eucariotas. La mayoría de los sitios de poliadenilación humanos contienen la secuencia AAUAAA, pero esta secuencia es menos común en plantas y hongos.
El ARN normalmente se escinde antes de la terminación de la transcripción, ya que CstF también se une a la ARN polimerasa II. A través de un mecanismo poco comprendido (a partir de 2002), indica que la ARN polimerasa II se salga de la transcripción. La escisión también involucra a la proteína CFII, aunque se desconoce cómo. El sitio de escisión asociado con una señal de poliadenilación puede variar hasta unos 50 nucleótidos.
Cuando se escinde el ARN, comienza la poliadenilación, catalizada por la poliadenilato polimerasa. La poliadenilato polimerasa construye la cola poli(A) añadiendo unidades de monofosfato de adenosina del trifosfato de adenosina al ARN, escindiendo el pirofosfato. Otra proteína, PAB2, se une a la nueva y corta cola poli(A) y aumenta la afinidad de la poliadenilato polimerasa por el ARN. Cuando la cola de poli(A) tiene aproximadamente 250 nucleótidos de largo, la enzima ya no puede unirse al CPSF y la poliadenilación se detiene, determinando así la longitud de la cola de poli(A). CPSF está en contacto con la ARN polimerasa II, lo que le permite indicarle a la polimerasa que termine la transcripción. Cuando la ARN polimerasa II alcanza una "secuencia de terminación" (⁵'TTTATT3' en la plantilla de ADN y ⁵'AAUAAA3' en la transcripción primaria), el final de la transcripción está señalizado. La maquinaria de poliadenilación también está físicamente unida al espliceosoma, un complejo que elimina los intrones de los ARN.
Efectos posteriores
La cola de poli(A) actúa como sitio de unión para la proteína de unión a poli(A). La proteína de unión a poli(A) promueve la exportación desde el núcleo y la traducción, e inhibe la degradación. Esta proteína se une a la cola poli(A) antes de la exportación del ARNm desde el núcleo y, en la levadura, también recluta poli(A) nucleasa, una enzima que acorta la cola poli(A) y permite la exportación del ARNm. La proteína de unión a poli(A) se exporta al citoplasma con el ARN. Los ARNm que no se exportan son degradados por el exosoma. La proteína de unión a poli(A) también puede unirse y, por tanto, reclutar, varias proteínas que afectan la traducción, una de ellas es el factor de iniciación 4G, que a su vez recluta la subunidad ribosómica 40S. Sin embargo, no se requiere una cola poli(A) para la traducción de todos los ARNm. Además, la cola poli(A) (oligoadenilación) puede determinar el destino de las moléculas de ARN que generalmente no tienen cola poli(A) (como los (pequeños) ARN no codificantes (sn), etc.) y, por lo tanto, inducir su ARN. decadencia.
Deadenilación
En las células somáticas eucariotas, las colas poli(A) de la mayoría de los ARNm en el citoplasma se acortan gradualmente, y los ARNm con cola poli(A) más corta se traducen menos y se degradan antes. Sin embargo, pueden pasar muchas horas antes de que se degrade un ARNm. Este proceso de deadenilación y degradación puede acelerarse mediante microARN complementarios a la región 3 'no traducida de un ARNm. En óvulos inmaduros, los ARNm con colas poli(A) acortadas no se degradan, sino que se almacenan y son traduccionalmente inactivos. Estos ARNm de cola corta se activan mediante poliadenilación citoplasmática después de la fertilización, durante la activación del óvulo.
En animales, la poli(A) ribonucleasa (PARN) puede unirse a la tapa 5' y eliminar nucleótidos de la cola de poli(A). El nivel de acceso a la tapa 5′ y a la cola poli(A) es importante para controlar la rapidez con la que se degrada el ARNm. PARN se muere menos si el ARN está unido por los factores de iniciación 4E (en la tapa 5 ') y 4G (en la cola poli (A)), razón por la cual la traducción reduce la muerte. La tasa de deadenilación también puede estar regulada por proteínas de unión a ARN. Además, las estructuras de triple hélice del ARN y los motivos de ARN, como el bolsillo de unión del extremo 3' de la cola poli(A), retardan el proceso de deadenilación e inhiben la eliminación de la cola poli(A). Una vez que se elimina la cola de poli(A), el complejo de descapsulación elimina la tapa 5', lo que provoca una degradación del ARN. Varias otras proteínas participan en la desactivación de células humanas y de levadura en ciernes, en particular el complejo CCR4-Not.
Poliadenilación citoplasmática
Existe poliadenilación en el citosol de algunos tipos de células animales, concretamente en la línea germinal, durante la embriogénesis temprana y en los sitios postsinápticos de las células nerviosas. Esto alarga la cola poli(A) de un ARNm con una cola poli(A) acortada, de modo que el ARNm se traduzca. Estas colas de poli(A) acortadas suelen tener menos de 20 nucleótidos y se alargan hasta alrededor de 80 a 150 nucleótidos.
En el embrión temprano de ratón, la poliadenilación citoplasmática de los ARN maternos del óvulo permite que la célula sobreviva y crezca aunque la transcripción no comience hasta la mitad de la etapa de 2 células (etapa de 4 células en humanos). En el cerebro, la poliadenilación citoplasmática está activa durante el aprendizaje y podría desempeñar un papel en la potenciación a largo plazo, que es el fortalecimiento de la transmisión de señales de una célula nerviosa a otra en respuesta a los impulsos nerviosos y es importante para el aprendizaje y la formación de la memoria.
La poliadenilación citoplasmática requiere las proteínas de unión a ARN CPSF y CPEB, y puede involucrar otras proteínas de unión a ARN como Pumilio. Dependiendo del tipo de célula, la polimerasa puede ser el mismo tipo de poliadenilato polimerasa (PAP) que se utiliza en el proceso nuclear, o la polimerasa citoplasmática GLD-2.
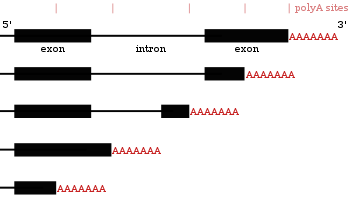
Poliadenilación alternativa
Muchos genes codificadores de proteínas tienen más de un sitio de poliadenilación, por lo que un gen puede codificar varios ARNm que difieren en su extremo 3'. La región 3' de una transcripción contiene muchas señales de poliadenilación (PAS). Cuando se utilizan sitios PAS más proximales (más cerca del extremo 5'), se acorta la longitud de la región 3' no traducida (3' UTR) de una transcripción. Los estudios tanto en humanos como en moscas han demostrado APA específicos de tejido. Los tejidos neuronales prefieren el uso de PAS distal, lo que lleva a UTR 3' más largas y los tejidos testiculares prefieren el PAS proximal, lo que lleva a UTR 3' más cortas. Los estudios han demostrado que existe una correlación entre el nivel de conservación de un gen y su tendencia a realizar una poliadenilación alternativa, y los genes altamente conservados exhiben más APA. De manera similar, los genes altamente expresados siguen este mismo patrón. Los datos de ribosecuenciación (secuenciación de solo ARNm dentro de los ribosomas) han demostrado que es más probable que se traduzcan las isoformas de ARNm con UTR 3' más cortas.
Dado que la poliadenilación alternativa cambia la longitud del segmento 3' UTR, también puede cambiar qué sitios de unión están disponibles para los microARN en la UTR 3'. Los microARN tienden a reprimir la traducción y promover la degradación de los ARNm a los que se unen, aunque existen ejemplos de microARN que estabilizan las transcripciones. La poliadenilación alternativa también puede acortar la región codificante, haciendo así que el ARNm codifique una proteína diferente, pero esto es mucho menos común que simplemente acortar la región 3' no traducida.
La elección del sitio poli(A) puede verse influenciada por estímulos extracelulares y depende de la expresión de las proteínas que participan en la poliadenilación. Por ejemplo, la expresión de CstF-64, una subunidad del factor estimulador de escisión (CstF), aumenta en los macrófagos en respuesta a los lipopolisacáridos (un grupo de compuestos bacterianos que desencadenan una respuesta inmune). Esto da como resultado la selección de sitios poli(A) débiles y, por tanto, transcripciones más cortas. Esto elimina elementos reguladores en las regiones 3' no traducidas de los ARNm para productos relacionados con la defensa como la lisozima y el TNF-α. Estos ARNm tienen vidas medias más largas y producen más de estas proteínas. Las proteínas de unión a ARN distintas a las de la maquinaria de poliadenilación también pueden afectar la utilización de un sitio de poliadenilación, al igual que la metilación del ADN cerca de la señal de poliadenilación. Además, muchos otros componentes implicados en la transcripción, el empalme u otros mecanismos que regulan la biología del ARN pueden afectar al APA.
Etiquetado para degradación en eucariotas
Para muchos ARN no codificantes, incluidos ARNt, ARNr, ARNsn y ARNsno, la poliadenilación es una forma de marcar el ARN para su degradación, al menos en la levadura. Esta poliadenilación se realiza en el núcleo mediante el complejo TRAMP, que mantiene una cola de alrededor de 4 nucleótidos de largo hasta el extremo 3 '. Luego, el exosoma degrada el ARN. También se han encontrado colas poli(A) en fragmentos de ARNr humano, tanto en forma de colas homopoliméricas (solo A) como heteropoliméricas (principalmente A).
En procariotas y orgánulos

En muchas bacterias, tanto los ARNm como los ARN no codificantes pueden poliadenilarse. Esta cola poli(A) promueve la degradación por parte del degradosoma, que contiene dos enzimas que degradan el ARN: polinucleótido fosforilasa y RNasa E. La polinucleótido fosforilasa se une al extremo 3' de los ARN y la extensión 3' proporcionada por la cola poli(A) le permite unirse a los ARN cuya estructura secundaria bloquearía el extremo 3'. Las sucesivas rondas de poliadenilación y degradación del extremo 3' por la polinucleótido fosforilasa permiten que el degradosoma supere estas estructuras secundarias. La cola poli(A) también puede reclutar RNasas que cortan el ARN en dos. Estas colas bacterianas poli(A) tienen aproximadamente 30 nucleótidos de largo.
En grupos tan diferentes como los animales y los tripanosomas, las mitocondrias contienen colas poli(A) tanto estabilizadoras como desestabilizadoras. La poliadenilación desestabilizadora se dirige tanto al ARNm como a los ARN no codificantes. Las colas de poli(A) tienen una longitud media de 43 nucleótidos. Los estabilizadores comienzan en el codón de parada, y sin ellos el codón de parada (UAA) no está completo ya que el genoma sólo codifica la parte U o UA. Las mitocondrias de las plantas sólo tienen poliadenilación desestabilizadora. Nunca se ha observado poliadenilación mitocondrial ni en levaduras de gemación ni de fisión.
Si bien muchas bacterias y mitocondrias tienen poliadenilato polimerasas, también tienen otro tipo de poliadenilación, realizada por la propia polinucleótido fosforilasa. Esta enzima se encuentra en bacterias, mitocondrias, plastidios y como constituyente del exosoma de las arqueas (en aquellas arqueas que tienen un exosoma). Puede sintetizar una extensión 3′ donde la gran mayoría de las bases son adeninas. Al igual que en las bacterias, la poliadenilación por la polinucleótido fosforilasa promueve la degradación del ARN en los plastidios y probablemente también en las arqueas.
Evolución
Aunque la poliadenilación se observa en casi todos los organismos, no es universal. Sin embargo, la amplia distribución de esta modificación y el hecho de que esté presente en organismos de los tres dominios de la vida implica que se supone que el último ancestro común universal de todos los organismos vivos tenía alguna forma de sistema de poliadenilación. Algunos organismos no poliadenilan el ARNm, lo que implica que han perdido sus maquinarias de poliadenilación durante la evolución. Aunque no se conocen ejemplos de eucariotas que carezcan de poliadenilación, los ARNm de la bacteria Mycoplasma gallisepticum y la arcaica tolerante a la sal Haloferax volcanii carecen de esta modificación.
La enzima poliadenilación más antigua es la polinucleótido fosforilasa. Esta enzima es parte tanto del degradosoma bacteriano como del exosoma de arqueas, dos complejos estrechamente relacionados que reciclan el ARN en nucleótidos. Esta enzima degrada el ARN atacando el enlace entre los nucleótidos más en 3' con un fosfato, rompiendo un nucleótido difosfato. Esta reacción es reversible, por lo que la enzima también puede extender el ARN con más nucleótidos. La cola heteropolimérica añadida por la polinucleótido fosforilasa es muy rica en adenina. La elección de la adenina es probablemente el resultado de concentraciones de ADP más altas que otros nucleótidos como resultado del uso de ATP como moneda energética, lo que hace que sea más probable que se incorpore a esta cola en las primeras formas de vida. Se ha sugerido que la participación de colas ricas en adenina en la degradación del ARN impulsó la evolución posterior de las poliadenilato polimerasas (las enzimas que producen colas poli(A) sin otros nucleótidos en ellas).
Las poliadenilato polimerasas no son tan antiguas. Han evolucionado por separado tanto en bacterias como en eucariotas a partir de la enzima que agrega CCA, que es la enzima que completa los extremos 3 'de los ARNt. Su dominio catalítico es homólogo al de otras polimerasas. Se presume que la transferencia horizontal de la enzima bacteriana que agrega CCA a eucariotas permitió que la enzima que agrega CCA similar a las arqueas cambiara su función a una poli (A) polimerasa. Algunos linajes, como las arqueas y las cianobacterias, nunca desarrollaron una poliadenilato polimerasa.
Se observan colas de poliadenilato en varios virus de ARN, incluidos la influenza A, el coronavirus, el virus del mosaico de la alfalfa y la hepatitis A del pato. Algunos virus, como el VIH-1 y el poliovirus, inhiben la proteína de unión poli-A de la célula. (PABPC1) para enfatizar sus propios genes' expresión sobre la de la célula huésped.
Historia
La poli(A)polimerasa se identificó por primera vez en 1960 como una actividad enzimática en extractos elaborados a partir de núcleos celulares que podían polimerizar ATP, pero no ADP, en poliadenina. Aunque se identificó en muchos tipos de células, esta actividad no tuvo función conocida hasta 1971, cuando se encontraron secuencias poli(A) en los ARNm. Al principio se pensó que la única función de estas secuencias era la protección del extremo 3 'del ARN contra las nucleasas, pero más tarde se identificaron las funciones específicas de la poliadenilación en la exportación y traducción nuclear. Las polimerasas responsables de la poliadenilación se purificaron y caracterizaron por primera vez en las décadas de 1960 y 1970, pero la gran cantidad de proteínas accesorias que controlan este proceso no se descubrieron hasta principios de la década de 1990.