Poblamiento de China
Historia genética

Sinopsis
Estudios arqueológicos en las llanuras centrales
Estudios arqueológicos en el sur de China
- Personas arcaicas de 12.000 a 10.000 BP
- Longlin ()隆) (10,686–10,439 cal BP; Laomocao Cave ♫, El Condado Autónomo de Longlin, Guangxi, China) es un individuo paleolítico superior con ascendente ascendencia de Asia oriental. Aunque los restos de Longlin tienen fenotipos excepcionalmente arcaicos, los análisis genéticos muestran que Longlin llevaba niveles similares de ascendencia humana arcaica como los asiáticos orientales neolíticos y actuales. Sin embargo, otro estudio sugiere que sus fenotipos se derivan de la hibridación con homininas arcaicas. Longlin parece estar estrechamente relacionado con la gente de Maludong o Red Deer Cave. También se relacionan estrechamente con Ikawazu (ca. 2,700 BP; Ikawazu Shell Midden site (伊 traiciona貝塚), Atsumi Peninsula, Southern Aichi Prefecture, central Honshu, Japón), un individuo Jōmon, que con linajes basales asiáticos como Hoabinhian. Longlin, Ikawazu, y Neolithic East Asians costeros de Shandong y Fujian probablemente todos se divergieron unos de otros al mismo tiempo, con Longlin se encuentra en la posición basal en el linaje que conduce a M71d, compartiendo una conexión genética materna con las poblaciones actuales de Asia sudoriental continental.
- Qihe-3 ()奇) (11,747–11,356 cal BP; Cueva Qihe 奇, Zhangping, Fujian) es un individuo paleolítico superior del interior montañoso de Fujian, situado a unos 100 km al norte de la actual ciudad de Zhangzhou. Consistente con otro retraso Homo sapiens, Qihe-3 exhibió una larga cabeza, gran capacidad craneal, cara estrecha alta, forma nasal amplia y baja y exhibe características de las poblaciones norte y sur de la China neolítico. Qihe-3 se puede modelar como una mezcla de ancestro que se encuentra en Neolithic oriental asiáticos costeros (p. ej., Boshan en Neolithic Shandong y Liangdao en Neolithic Fujian), así como otra línea de Asia oriental profundamente divergentes.
- Qihe-2, un espécimen más reciente de una capa diferente del mismo sitio que data de 8,428-8,359 cal BP, fue secuenciado y encontrado que está estrechamente relacionado con la edad de hierro Taiwán y Austronesios actuales.
- Liangdao-2 (~7.600 BP; Liangdao, Fujian), se encontró que tenía mayormente ancestro relacionado con Qihe-3 (82%–90%), así como un porcentaje menor de ascendencia norte de Asia oriental (10%–18%) que puede estar asociado con Neolithic Shandong y otros sitios del este del norte asiático. Como resultado, hay más continuidad genética de lo temprano a lo tardío Neolítico en Fujian que en Guangxi.
- Personas arqueas de 9.000 a 6.000 BP
- Dushan ()独iado) (8,974–8,593 cal BP; Linfeng Town 林逢, Tiandong County, Guangxi) es un individuo masculino que puede ser modelado como una mezcla de ancestro relacionado con Longlin (17%) y ancestro relacionado con Qihe (83%). Esto sugiere una migración masiva de antiguas poblaciones Fujian en Guangxi, donde se mezclaron con los habitantes indígenas en lugar de reemplazarlos por completo.
- Baojianshan ()Alternativa) (8,335–6,400 cal BP; Baojianshan Cave (Baojianshan Cave A), Longzhou County, Chongzuo City, Guangxi) se puede modelar como una mezcla de 72,3% de ancestro relacionado con Dushan y 27.7% de ancestro relacionado con Hoabinhian.
BaBanQinCen y GaoHuaHua fueron también dos poblaciones antiguas de Guangxi, de hace 1500 y 500 años respectivamente, que contribuyeron a la composición genética de los grupos actuales Kra-Dai y Hmong-Mien de la región. BaBanQinCen puede describirse como una mezcla de ascendencia relacionada con Dushan (5%–64%), asiáticos del norte de Asia Oriental (19%–40%) y asiáticos del sur de Asia Oriental (5%–72%). Se observan patrones similares en los grupos Kra-Dai actuales. GaoHuaHua puede describirse como una mezcla de ascendencia del norte de Asia Oriental (Boshan, 34%) y ascendencia relacionada con Dushan (66%). Los zhuang y los dong del condado de Congjiang en Guizhou, China, también se agrupan con estas antiguas poblaciones de Guangxi.
Mientras tanto, las poblaciones de Fujian del Neolítico Tardío, representadas por Xitoucun y Tanshishan, se modelan mejor como una mezcla de ascendencia relacionada con Dushan (35/54%), ascendencia del norte de Asia Oriental (44/34%), ascendencia relacionada con Qihe3 (17/8%) y ascendencia relacionada con la Periferia del Indo (4/3%).
Huang et al. (2022) asocian la ascendencia relacionada con Dushan y Baojianshan con los primeros agricultores neolíticos del Sudeste Asiático Continental (MSEA), es decir, agricultores del Neolítico tardío que se expandieron desde el sur de China hacia el MSEA. Entre las poblaciones actuales, encontraron que:- Los oradores de Khmuic (Austroasiático) en el sudeste continental de Asia, como el Mal (Htin) y Mlabri, en su mayoría llevan el primer ancestro relacionado con el agricultor MSEA (69,2–75,2%).
- Los altavoces Hmongic Occidental (Longlin Miao, Xilin Miao y Hmong) tienen un poco más de la primera ascendencia relacionada con el agricultor MSEA (32,3–35,0%) que la ascendencia neol Fujian Austronesiana (23,7–26,0%).
- Se encontró que Hlai, Maonan y Guangxi Zhuang tenían más ascendencia neolítica relacionada con Fujian Austronesiana (40,7–53,9%, con Hlai llevando más al 46,1–53,9%) que la primera ascendencia relacionada con el agricultor MSEA (24,9–33,1%).
- Al igual que las poblaciones de habla Kra-Dai, los chinos del sur de Han en Fujian y Guangdong llevan más retraso en la ascendencia neolítica relacionada con la autóstica Fujian (35.0–40,3%) que la primera ascendencia relacionada con los agricultores de MSEA (21,8–23,6%).
Estudios genéticos de Shaanxi y Liaoning
Paternal lineages

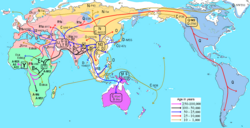

"La mayor parte del trabajo para construir un dique y drenar un área consiste en cavar una zanja y abonar la tierra para construir un dique de tierra. Para construir la alcantarilla y la compuerta de marea, se puede usar una canoa vieja y desgastada como tubería... y prácticamente cualquier piedra plana de buen tamaño que se consiga. Si se cuenta con el doble de personas para construir el dique, se puede construir el doble de metros lineales de dique. Duplicar el perímetro de un dique cuadrado resulta en cuadruplicar el área dentro del dique."El excedente agrícola habría permitido una rápida expansión demográfica, lo que proporcionaría más mano de obra para el riego. Para el momento del establecimiento de las dinastías Xia y Shang, la población estimada era de aproximadamente 13 millones de personas.Estudios de restos de ADN de las llanuras centrales de China de hace 3000 años muestran una estrecha afinidad entre esa población y la de la etnia Han del Norte actual, tanto en el ADN-Y como en el ADNmt. Tanto la etnia Han del Norte como la del Sur muestran una estructura genética de ADN-Y similar.
El haplogrupo O2-M122 del cromosoma Y es un marcador de ADN común en los chinos Han, tal como apareció en China en tiempos prehistóricos. Se encuentra en más del 50% de los varones chinos, con frecuencias que tienden a ser altas hacia el este del país (30/101 = 29,7% Han de Guangxi Pinghua, 13/40 = 32,5% Han de Guangdong, 11/30 = 36,7% Han de Lanzhou, 26/60 = 43,3% Han de Yunnan, 251/565 = 44,4% Han de Zhaotong, 15/32 = 46,9% Han de Yili, 23/49 = 46,9% Han de Lanzhou, 32/65 = 49,2% Han del sur de China, 18/35 = 51,4% Han de Meixian, 22/42 = 52,4% Han del norte, 43/82 = 52,4% Han del norte, 18/34 = 52,9% Han de Chengdu, 154/280 = 55,0% Han del Sur, 27/49 = 55,1% Han del Norte, 73/129 = 56,6% Han del Norte de China, 49/84 = 58,3% Han de Taiwán, 35/60 = 58,3% Han de Taiwán, 99/167 = 59,3% Han del Este de China, 33/55 = 60,0% Han de Fujian, 157/258 = 60,9 % Han de Taiwán, 13/21 = 61,9 % Han de Taiwán, 189/305 = 62,0 % Han de Zibo, 23/35 = 65,7 % Han de Harbin, 29/44 = 65,9 % Han del Norte, 23/34 = 67,6 % Hakka de Taiwán, 35/51 = 68,6% Pekín Han).
Durante la dinastía Zhou, o incluso antes, los pueblos con el haplogrupo Q-M120 también contribuyeron a la etnogénesis del pueblo chino Han. Se presupone que este haplogrupo está extendido en la estepa euroasiática y el norte de Asia, ya que se encuentra entre los cimerios de Moldavia y los nativos de la Edad de Bronce de Khövsgöl. Sin embargo, actualmente está prácticamente ausente en estas regiones, excepto en Asia Oriental. En la China moderna, el haplogrupo Q-M120 se encuentra en las regiones septentrional y oriental.Los chinos Han se distinguen genéticamente de los japoneses Yamato y los coreanos, e internamente, los diferentes subgrupos chinos Han son genéticamente más cercanos entre sí que cualquiera de ellos a los coreanos y japoneses. Sin embargo, algunos chinos Han del sur, como los Han de Guangxi, son genéticamente más cercanos a los vietnamitas y dai que los Han del norte. Pero mientras tanto, cuando se compara la genética europea, los chinos Han, los del sudeste asiático, los japoneses y los coreanos son más cercanos entre sí que los europeos y los asiáticos del sur. La investigación genealógica ha indicado perfiles genéticos extremadamente similares con una variación total de menos del 1% en el espectro entre estos tres grupos. Algunos chinos Han del sur y del norte son más cercanos entre sí y muestran las diferencias más pequeñas cuando se comparan con otros asiáticos. Los vietnamitas Kinh de Ciudad Ho Chi Minh están cerca de la minoría étnica Dai de Xishuangbanna y los chinos Han de Guangdong. Los coreanos también son relativamente cercanos a los chinos Han del norte. Los japoneses son más distantes genéticamente de los coreanos que los coreanos de los chinos Han del norte. Sin embargo, los mongoles buriatos y los mongoles qinghai están más distanciados entre sí que los japoneses y los coreanos, a pesar de ser ambos mongoles. Las comparaciones entre el SNP del cromosoma Y y el ADNmt de los chinos han del norte modernos y las muestras antiguas de Hengbei de 3000 años de antigüedad de las llanuras centrales de China muestran que son extremadamente similares entre sí y muestran una continuidad entre los antiguos chinos de Hengbei y los actuales chinos han del norte, mientras que los chinos han del sur eran diferentes de los habitantes de Hengbei. Esto demostró que ya hace 3000 años se había formado la estructura genética actual de los chinos han del norte.linajes maternas
Climate history
Patrones de liquidación
Véase también
- Peopling of Southeast Asia
- Lista de sitios paleolíticos en China
- Lista de sitios neolíticos en China
- Lista de Bronce Sitios de edad en China
- Lista de primeros asentamientos humanos
- Han Chinese § DNA and genetics analysis
Referencias
- ^ Seguin-Orlando, Andaine; Korneliussen, Thorfinn S.; Sikora, Martin; Malaspinas, Anna-Sapfo; Manica, Andrea; Moltke, Ida; Albrechtsenovich, Anders; Ko, Amy; Margaryan, Ashot; Moiseyev, Vyacheslav; Goebel, Ted; Westaway, Michael; Lambert, David; Khar "La estructura genómica en europeos data por lo menos 36,200 años". Ciencia. 346 (6213): 1113 –1118. Bibcode:2014Sci...346.1113S. doi:10.1126/science.aaaa0114. PMID 25378462.
- ^ Vallini, Leonardo; Zampieri, Carlo; Shoaee, Mohamed Javad; Bortolini, Eugenio; Marciani, Giulia; Aneli, Serena; Pievani, Telmo; Benazzi, Stefano; Barausse, Alberto; Mezzavilla, Massimo; Petraglia, Michael D.; Pagani, Luca (25 de marzo de 2024). "La meseta persa sirvió como centro de Homo sapiens después de la dispersión principal de África". Nature Communications. 15 (1): 1882 Bibcode:2024NatCo..15.1882V. doi:10.1038/s41467-024-461-7. PMC 10963722. PMID 38528002.
- ^ a b Aoki, Kenichi; Takahata, Naoyuki; Oota, Hiroki; Wakano, Joe Yuichiro; Feldman, Marcus W. (30 agosto 2023). "Las enfermedades infecciosas pueden haber arrestado el avance hacia el sur de las microbladas en el Alto Palaeolítica Asia Oriental". Proceedings of the Royal Society B: Biological Sciences. 290 (2005). doi:10.1098/rspb.2023.1262. PMC 10465978. PMID 37644833.
Una sola migración importante de humanos modernos en los continentes de Asia y Sahul fue fuertemente apoyada por estudios anteriores utilizando ADN mitocondrial, la porción no recombinante de cromosomas Y y datos de SNP autosómicos [42–45]. Ancestral Los antiguos indios del sur sin relación euroasiática occidental, asiáticos orientales, onge (cazadores andamaneses) y los papúes se derivan en un corto tiempo evolutivo de la dispersión hacia el este de una población de fuera de África [46,47]. [...] El consorcio Pan-Asian SNP [44] investigó la diversidad de haplotipos dentro de las poblaciones asiáticas actuales y encontró una fuerte correlación con la latitud, con la diversidad disminuyendo de sur a norte. La correlación sigue existiendo cuando sólo se consideran las poblaciones de Asia sudoriental y Asia oriental, y puede atribuirse a un efecto fundador de serie [50]. Estas observaciones son coherentes con la opinión de que poco después de la migración única hacia el este de los seres humanos modernos, los asiáticos orientales se dividieron en el Asia meridional y se dispersaron hacia el norte por todo el continente.
- ^ Moreno-Mayar, J. Víctor; Potter, Ben A.; Vinner, Lasse; Steinrücken, Matthias; Rasmussen, Simon; Terhorst, Jonathan; Kamm, John A.; Albrechtsen, Anders; Malaspinas, Anna-Sapfo; Sikora, Martin; Reuther, Joshua D.; Irish, Joel D.; Malhi, Lupando. "El genoma de Alaska del pleistoceno terminal revela la primera población fundadora de los nativos americanos" (PDF). Naturaleza. 553 (7687): 203 –207. Bibcode:2018Natur.553..203M. doi:10.1038/nature25173. PMID 293294.
- ^ Yang, Melinda A. (6 de enero de 2022). "Una historia genética de migración, diversificación y admixtura en Asia". Genética y Genómica de Población Humana: 1 –32. doi:10.47248/hpgg2202010001.
- ^ Adhikari, Kaustubh; Fuentes-Guajardo, Macarena; Quinto-Sánchez, Mirsha; Mendoza-Revilla, Javier; Camilo Chacón-Duque, Juan; Acuña-Alonzo, Victor; Jaramillo, Claudia; Arias, William; Lozano, Rodrigo Barquera; Pérez, Gastón Macín; Gómez-Valdés, Jorge; Villamil-Rabit "Un análisis de asociación en todo el genoma implica DCHS2, RUNX2, GLI3, PAX1 y EDAR en la variación facial humana". Nature Communications. 7 (1): 11616. Bibcode:2016NatCo...7116A. doi:10.1038/ncomms116. PMC 4874031. PMID 27193062.
- ^ a b Huang, Xiufeng; Xia, Zi-Yang; Bin, Xiaoyun; He, Guanglin; Guo, Jianxin; Adnan, Atif; Yin, Lianfei; Huang, Youyi; Zhao, Jing; Yang, Yidong; Ma, Fuwei; Li, Yingxiang; Hu, Rong; Yang, Tianle; Wei, Chun "Pesas genómicas en la historia demográfica de los chinos del sur". Fronteras en Ecología y Evolución. 10. doi:10.3389/fevo.2022.853391.
- ^ Yang, Melinda A. (6 de enero de 2022). "Una historia genética de migración, diversificación y admixtura en Asia". Genética y Genómica de Población Humana: 1 –32. doi:10.47248/hpgg2202010001.
... Por el contrario, los asiáticos de Oriente y Sudeste y otros isleños del Pacífico (por ejemplo, los hablantes de Austronesia) están estrechamente relacionados entre sí [9,15,16] y aquí denotados como pertenecientes a un linaje de Asia Oriental y Sudoriental (ESEA) (Box 2). ...el linaje ESEA diferenciado en por lo menos tres diferentes antepasados: Ancestro de Tianyuan que se puede encontrar hace 40.000-33.000 años en el norte de Asia oriental, ancestro encontrado hoy en las poblaciones actuales de Asia oriental, Asia sudoriental y Siberia, pero cuyos orígenes son desconocidos, y ancestro de Hòabìnhian encontrado hace 8.000-4,000 años en el sudeste asiático, pero cuyos orígenes en el Paleolítico Superior son desconocidos.
- ^ a b c Ma, Hao; Hu, Jizhong; Wang, Rui; Qiao, Yu; Yang, Panming; Qiao, Xiaoyang; Pu, Youwei; Zhou, Yawei; Xu, Yu; Chen, Haodong; Wang, Chuan-Chao; Yang, Zhaohui (abril 2025). "Subestructura genómica en los agricultores de la cuenca del río Amarillo durante el período de transición Yangshao-Longshan". Journal of Archaeological Science. 176: 106178. Bibcode:2025JArSc.176j6178M. doi:10.1016/j.jas.2025.106178.
- ^ Gao, Shizhu; Cui, Yinqiu (2023). "Los genomas científicos revelan el origen y la historia evolutiva de las poblaciones chinas". Frontiers in Earth Science. 10. Bibcode:2023FrEaS..1059196G. doi:10.3389/feart.2022.1059196.
- ^ Wu, Xiyan; Ding, Baoxu; Nie, Linyi; et al. (2024). "Excepciones genómicas sobre la compleja historia demográfica y los fenómenos de engendramiento durante la dinastía Zhou en las llanuras centrales de China". Fronteras en Microbiología. 15. doi:10.3389/fmicb.2024.1471740. PMC 11427373. PMID 39345259.
- ^ Wang, Chuan-Chao; Yeh, Hui-Yuan; Popov, Alexander N.; Zhang, Hu-Qin; Matsumura, Hirofumi; Sirak, Kendra; Cheronet, Olivia; Kovalev, Alexey; Rohland, Nadin; Kim, Alexander M.; Mallick, Swapan; Bernardos, Rebecca; Tumen, Dashtseveg; Liu, Yi-Chang; Liu, Jiun-Yu; Mah, Mateo; Wang, Ke; Zhanga, Zhanie; "Excepciones genómicas sobre la formación de poblaciones humanas en Asia Oriental". Naturaleza. 591 (7850): 413 –419. Bibcode:2021Natur.591..413W. doi:10.1038/s41586-021-03336-2. PMC 7993749. PMID 33618348.
- ^ a b c d e f h i j Bennett, Andrew E.; Liu, Yichen; Fu, Qiaomei (2025). Reconstruyendo la historia de la población humana de Asia oriental a través de la antigua genómica. doi:10.1017/9781009246675. ISBN 978-1-009-24667-5.
- ^ a b c d Su, Haoran; Wang, Mengge; Li, Xiangping; Duan, Shuhan; Sun, Qiuxia; Sun, Yuntao; Wang, Zhiyong; Yang, Qingxin; Huang, Yuguo; Zhong, Jie; Chen, Jing; Jiang, Xiucheng; Ma, Jinyue; Yang, Ting; Liu, Yunhui; Luo, Lintao; Liu, Yan; Yang, Junbao; Chen, Gang; Liu, Chao; Cai, Yan; Él, Guanglin (18 de junio de 2024). "Population genetic admixture and evolutionary history in the Shandong Peninsula inferred from integrative modern and former genomic resources". BMC Genomics. 25 1): 611. doi:10.1186/s12864-024-10514-9. PMC 11184692. PMID 38890579.
- ^ Huang, Xiufeng; Xia, Zi-Yang; Bin, Xiaoyun; et al. (2020). "Las visiones genómicas en la historia demográfica del sur de China". biorxiv. doi:10.1101/2020.11.08.373225.
- ^ Wang, Mengge; Él, Guanglin; Zou, Xing; et al. (2021). "Nuevas ideas de la discriminación combinada de los alelos y haplotipos compartidos del genoma moderno y científico: Reconstrucción de historia demográfica diferenciada de las personas Tai-Kadai y Sinitic en el sur de China". biorxiv.
- ^ Liu, Xiaoxi; Koyama, Satoshi; Tomizuka, Kohei; Takata, Sadaaki; Ishikawa, Yuki; Ito, Shuji; Kosugi, Shunichi; Suzuki, Kunihiko; Hikino, Keiko; Koido, Masaru; Koike, Yoshinao; Horikoshi, Momochioz "Decoding triancestral origins, archaic introgression, and natural selection in the Japanese population by whole-genome sequencing". Avances científicos. 10 (16): eadi8419. Bibcode:2024SciA...10I8419L. doi:10.1126/sciadv.adi8419. PMC 11023554. PMID 38630824.
- ^ Zhou, Jingbin; Zhang, Xianpeng; Li, Xin; Sui, Jie (2022). "La estructura genética y la historia demográfica de las personas del norte de Han en la provincia de Liaoning se inferían de datos de matriz de todo el genoma". Fronteras en Ecología y Evolución. 10. doi:10.3389/fevo.2022.1014024.
- ^ a b c d Wang, Tianyi; Wang, Wei; Xie, Guangmao; Li, Zhen; Fan, Xuechun (2021). "La historia de la población humana en la encrucijada de Asia oriental y sudoriental desde hace 11.000 años". Celular. 184 (14). Elsevier BV: 3829–3841.e21. doi:10.1016/j.cell.2021.05.018. PMID 34171307.
- ^ Curnoe, Darren; Ji, Xueping; Taçon, Paul S.C.; Yaozheng, Ge (2015). "Posibles firmas de la híbridación de Hominin desde el Holoceno temprano del suroeste de China". Scientific Reports. 5 (12408) – vía Naturaleza.
- ^ Xiujie, Wu (2014). "El cráneo humano neolítico temprano de la Cueva Qihe, Zhangping, Fujian". Acta Antropologica Sinica. 33 (4) – vía SciEngine.
- ^ Wang, Chuan-Chao; Yeh, Hui-Yuan; Popov, Alexander N.; Zhang, Hu-Qin; Matsumura, Hirofumi; Sirak, Kendra; Cheronet, Olivia; Kovalev, Alexey; Rohland, Nadin; Kim, Alexander M.; Mallick, Swapan; Bernardos, Rebecca; Tumen, Dashtseveg; Liu, Yi-Chang; Liu, Jiun-Yu; Mah, Mateo; Wang, Ke; Zhanga, Zhanie; "Excepciones genómicas sobre la formación de poblaciones humanas en Asia Oriental". Naturaleza. 591 (7850): 413 –419. Bibcode:2021Natur.591..413W. doi:10.1038/s41586-021-03336-2. PMC 7993749. PMID 33618348.
- ^ Wang, Jiawen; Wu, Jun; Sun, Qiuxia; Wu, Qian; Li, Youjing; Duan, Shuhan; Yang, Lin; Wu, Wenxin; Wang, Zheng; Liu, Yan; Tang, Renkuan; Yang, Junbao; Wang, Chuanchao; Liu, Chao; Xu, Jianwei; Wang, Mengge; He, Guanglin (12 de junio de 2023). "Extensiva mezcla genética entre personas de habla Tai-Kadai y sus vecinos en la región nororiental de la meseta de Yungui inferida de variaciones de todo el genoma". BMC Genomics. 24 (1): 317. doi:10.1186/s12864-023-09412-3. PMC 10259048. PMID 37308851.
- ^ Wang, Jiawen; Wu, Jun; Sun, Qiuxia; Wu, Qian; Li, Youjing; Duan, Shuhan; Yang, Lin; Wu, Wenxin; Wang, Zheng; Liu, Yan; Tang, Renkuan; Yang, Junbao; Wang, Chuanchao; Liu, Chao; Xu, Jianwei; Wang, Mengge; He, Guanglin (12 de junio de 2023). "Extensiva mezcla genética entre personas de habla Tai-Kadai y sus vecinos en la región nororiental de la meseta de Yungui inferida de variaciones de todo el genoma". BMC Genomics. 24 1). doi:10.1186/s12864-023-09412-3. PMC 10259048.
- ^ a b c d Él, Guang-Lin; Wang, Meng-Ge; Li, Ying-Xiang; Zou, Xing; Yeh, Hui-Yuan; Tang, Ren-Kuan; Yang, Xiao-Min; Wang, Zheng; Guo, Jian-Xin; Luo, Ting; Zhao, Jing; Sun, Jin; Hu, Rong; Wei, Lan-Hai; Chen, Gang; Hou, Yi-Ping; Wang, Chuan-Chao (Julio 2022). "Fine-scale north-to-south genetic admixture profile in Shaanxi Han Chinese revealed by genome-wide demographic history reconstruction". Journal of Systematics and Evolution. 60 4): 955–972. doi:10.1111/jse.12715.
- ^ Chiang, Charleston W.K.; Mangul, Serghei; Robles, Christopher; Sankararaman, Sriram (2018). "Un mapa integral de la variación genética en el grupo étnico más grande del mundo —Han Chinese". Biología molecular y evolución. 35 (11): 2736 –2750. doi:10.1093/molbev/msy170. PMC 6693441. PMID 30169787.
- ^ Zhou, Jingbin; Zhang, Xianpeng; Li, Xin; et al. (2022). "La estructura genética y la historia demográfica de las personas del norte de Han en la provincia de Liaoning se inferían de datos de matriz de todo el genoma". Fronteras en Ecología y Evolución. 10. doi:10.3389/fevo.2022.1014024.
- ^ Soares, Pedro A.; Trejaut, Jean A.; Rito, Teresa; Cavadas, Bruno; Hill, Catherine; Eng, Ken Khong; Mormina, Maru; Brandão, Andreia; Fraser, Ross M.; Wang, Tse-Yi; Loo, Jun-Hun; Snell, Christopher; Ko, Tsang-Ming; Amorim, António; Pala, Maria "Resolver la ascendencia de poblaciones de habla Austronesia". Genética Humana. 135 3): 309 –326. doi:10.1007/s00439-015-1620-z. PMC 4757630. PMID 26781090.
- ^ Hallast, Pille; Agdzhoyan, Anastasia; Balanovsky, Oleg; Xue, Yali; Tyler-Smith, Chris (Febrero 2021). "Un origen sudeste asiático para los cromosomas humanos y no africanos actuales". Genética Humana. 140 2): 299 –307. doi:10.1007/s00439-020-02204-9. PMC 7864842. PMID 29217544.
- ^ "Ancestro asiático basado en Estudios de Variación Y-DNA: Parte 1 Origen temprano — raíces de África y emergencia en Asia Oriental". Tutoriales de base genética. Archivado desde el original el 2 de enero de 2012. Retrieved 28 de diciembre 2011.
- ^ Shi H, Zhong H, Peng Y, et al. (2008). "Y cromosoma evidencia de la primera solución humana moderna en Asia oriental y múltiples orígenes de poblaciones tibetanas y japonesas". BMC Biol. 6: 45. doi:10.1186/1741-7007-6-45. PMC 2605740. PMID 18959782.
- ^ WANG, Chuan-Chao; YAN, Shi; QIN, Zhen-Dong; LU, Yan; DING, Qi-Liang; WEI, Lan-Hai; LI, Shi-Lin; YANG, Ya-Jun; JIN, Li; LI, Hui (mayo de 2013). "Late expansión neolítica de los antiguos chinos revelada por Y cromosoma haplogroup O3a1c-002611". Journal of Systematics and Evolution. 51 3): 280 –286. Bibcode:2013JSyEv.51..280W. doi:10.1111/j.1759-6831.2012.00244.x.
- ^ Chang-Qun, Duan; Xue-Chun, Gan; Wang, Jeanny; Chien, Paul K. (1998). "Relocación de Centros de Civilización en la China Antigua: Factores Ambientales". Ambio. 27 (7): 572 –575. JSTOR 4314793.
- ^ a b Zhao, Yong-Bin; Zhang, Ye; Zhang, Quan-Chao; Li, Hong-Jie; Cui, Ying-Qiu; Xu, Zhi; Jin, Li; Zhou, Hui; Zhu, Hong (4 de mayo de 2015). "Ancient DNA Reveals that the Genetic Structure of the Northern Han Chinese was Shaped Prior to 3,000 Years Ago". PLOS ONE. 10 (5): e0125676. código:2015PLoSO..1025676Z. doi:10.1371/journal.pone.0125676. PMC 4418768. PMID 25938511.
- ^ Gan RJ, Pan SL, Mustavich LF, Qin ZD, Cai XY, Qian J, Liu CW, Peng JH, Li SL, Xu JS, Jin L, Li H (2008). "La población de Pinghua es una excepción a la estructura genética coherente de Han Chino". Journal of Human Genetics. 53 4): 303 –13. doi:10.1007/s10038-008-0250-x. PMID 18270655.
- ^ a b c Hammer MF, Karafet TM, Park H, Omoto K, Harihara S, Stoneking M, Horai S (2006). "Los orígenes finales de los japoneses: terreno común para el cazador-recolector y los cromosomas Y agricultores". Journal of Human Genetics. 51 1): 47 –58. doi:10.1007/s10038-005-0322-0. PMID 16328082.
- ^ a b c d e Xue Y, Zerjal T, Bao W, Zhu S, Shu Q, Xu J, Du R, Fu S, Li P, Hurles ME, Yang H, Tyler-Smith C (abril de 2006). "Male demography in East Asia: a north-south contrast in human population expansion times". Genética. 172 4): 2431–9. doi:10.1534/genetics.105.054270. PMC 1456369. PMID 16489223.
- ^ a b Kim SH, Kim KC, Shin DJ, Jin HJ, Kwak KD, Han MS, Song JM, Kim W, Kim W (abril 2011). "High frequencies of Y-chromosome haplogroup O2b-SRY465 lineages in Korea: a genetic perspective on the peopling of Korea". Genética Investigadora. 2 1): 10. doi:10.1186/2041-2223-2-10. PMC 3087676. PMID 21463511.
 El texto fue copiado de esta fuente, que está disponible bajo una licencia Creative Commons Attribution 2.0 (CC BY 2.0).
El texto fue copiado de esta fuente, que está disponible bajo una licencia Creative Commons Attribution 2.0 (CC BY 2.0). - ^ a b Yin C, Su K, He Z, Zhai D, Guo K, Chen X, Jin L, Li S (Julio 2020). "Reconstrucción Genética y Análisis Forense de las Poblaciones Chinas Shandong y Yunnan Han por Co-Analyzing Y Chromosomal STRs y SNPs". Genes. 11 (7): 743. doi:10.3390/genes11070743. PMC 7397191. PMID 32635262.
- ^ Yao, Hongbin; Wang, Mengge; Zou, Xing; Li, Yingxiang; Yang, Xiaomin; Li, Ailin; Yeh, Hui-Yuan; Wang, Peixin; Wang, Zheng; Bai, Jingya; Guo, Jianxin; Chen, Jinwen; Ding, Xiao; Zhang, Yan; Lin, Baoquan; Wang, Chuan-Chao; He, Guanglin (Mayo 2021). "Nuevas ideas sobre la historia a gran escala de la mezcla occidental-oriental de la población china noroccidental en el Corredor Hexi a través del legado genético de todo el genoma". Genética molecular y genómica. 296 3): 631 –651. doi:10.1007/s00438-021-01767-0. PMID 33650010.
- ^ Yao H, Wang M, Zou X, Li Y, Yang X, Li A, Yeh HY, Wang P, Wang Z, Bai J, Guo J, Chen J, Ding X, Zhang Y, Lin B, Wang CC, He G (Mayo 2021). "Nuevas ideas sobre la historia a gran escala de la mezcla occidental-oriental de la población china del noroeste en el Corredor Hexi a través del legado genético de todo el genoma". Genética molecular y genómica. 296 3): 631 –651. doi:10.1007/s00438-021-01767-0. PMID 33650010.
- ^ a b c Yan S, Wang CC, Li H, Li SL, Jin L (septiembre de 2011). "Un árbol actualizado de Haplogroup O y posiciones filogenéticas revisadas de mutaciones P164 y PK4". European Journal of Human Genetics. 19 (9): 1013 –5. doi:10.1038/ejhg.2011.64. PMC 3179364. PMID 21505448.
- ^ Katoh T, Munkhbat B, Tounai K, Mano S, Ando H, Oyungerel G, et al. (febrero de 2005). "Características genéticas de grupos étnicos mongoles reveladas por el análisis cromosómico Y". Gene. 346: 63–70. doi:10.1016/j.gene.2004.10.023. PMID 15716011.
- ^ a b Su B, Xiao J, Underhill P, Deka R, Zhang W, Akey J, Huang W, Shen D, Lu D, Luo J, Chu J, Tan J, Shen P, Davis R, Cavalli-Sforza L, Chakraborty R, Xiong M, Du R, Oefner P, Chen Z, Jin L (diciembre de 1999). "Y-Cromosome evidence for a northward migration of modern humans into Eastern Asia during the last Ice Age". American Journal of Human Genetics. 65 (6): 1718 –24. doi:10.1086/302680. PMC 12883. PMID 10577926.
- ^ a b Tajima A, Hayami M, Tokunaga K, Juji T, Matsuo M, Marzuki S, Omoto K, Horai S (2004). "Los orígenes genéticos de los Ainu se derivan de análisis combinados de ADN de linajes materno-paternales". Journal of Human Genetics. 49 4): 187 –93. doi:10.1007/s10038-004-0131-x. PMID 14997363.
- ^ a b c d Trejaut JA, Poloni ES, Yen JC, Lai YH, Loo JH, Lee CL, He CL, Lin M (junio de 2014). "Taiwan Y-chromosomal DNA variación y su relación con Island Southeast Asia". BMC Genetics. 15: 77. doi:10.1186/1471-2156-15-77. PMC 4083334. PMID 24965575.
- ^ Yan S, Wang CC, Zheng HX, Wang W, Qin ZD, Wei LH y otros (2014). "Y cromosomas del 40% chino descienden de tres super-abuelos neolíticos". PLOS ONE. 9 (8): e105691. arXiv:1310.3897. Bibcode:2014PLoSO...9j5691Y. doi:10.1371/journal.pone.0105691. PMC 4149484. PMID 25170956.
- ^ Hurles ME, Sykes BC, Jobling MA, Forster P (Mayo 2005). "El origen dual de la malgache en la Isla Asia Sudoriental y África Oriental: evidencia de linajes materno-paternales". American Journal of Human Genetics. 76 5): 894–901. doi:10.1086/430051. PMC 1199379. PMID 15793703.
- ^ Sun, Na; Ma, Peng-Cheng; Yan, Shi; et al. (2019). "Phylogeography of Y-chromosome haplogroup Q1a1a-M120, a paternal lineage connecting populations in Siberia and East Asia". Annals of Human Biology. 46 3): 261 –266. doi:10.1080/03014460.2019.1632930. PMID 31208219.
- ^ Wang, Yuchen; Lu, Dongsheng; Chung, Yeun-Jun; Xu, Shuhua (diciembre de 2018). "La estructura genética, la divergencia y la mezcla de las poblaciones chinas, japonesas y coreanas de Han". Hereditas. 155 1): 19. doi:10.1186/s41065-018-0057-5. PMC 5889524. PMID 29636655.
- ^ Yanan Cao; Lin Li; Min Xu; Zhimin Feng; Xiaohui Sun; Jieli Lu; Yu Xu; Peina Du; Tiange Wang; Ruying Hu; Zhen Ye; Lixin Shi; Xulei Tang; Li Yan (30 de abril de 2020). "El análisis de ChinaMAP de profundas secuencias de genoma entero en 10.588 individuos". Cell Research. 30 (9): 717–731. doi:10.1038/s41422-020-0322-9. PMC 7609296. PMID 32355288.
- ^ Wang, Yuchen; Lu, Dongsheng; Chung, Yeun-Jun; Xu, Shuhua (6 de abril de 2018). "La estructura genética, la divergencia y la mezcla de las poblaciones chinas, japonesas y coreanas de Han". Hereditas. 155 (19): 19. doi:10.1186/s41065-018-0057-5. PMC 5889524. PMID 29636655.
- ^ Wen B, Li H, Lu D, Song X, Zhang F, He Y, et al. (septiembre 2004). "La evidencia genética apoya la difusión de la cultura Han". Naturaleza. 431 (7006): 302 –305. Bibcode:2004Natur.431..302W. doi:10.1038/nature02878. PMID 15372031.
- ^ Zhao YB, Zhang Y, Zhang QC, Li HJ, Cui YQ, Xu Z, Jin L, Zhou H, Zhu H (2015). "Ancient DNA revela que la estructura genética del norte de Han Chino se formó antes de hace 3.000 años". PLOS ONE. 10 (5): e0125676. código:2015PLoSO..1025676Z. doi:10.1371/journal.pone.0125676. PMC 4418768. PMID 25938511.
- ^ Yao YG, Kong QP, Bandelt HJ, Kivisild T, Zhang YP (marzo de 2002). "Diferenciación pilogeográfica del ADN mitocondrial en Han Chino". American Journal of Human Genetics. 70 3): 635 –51. doi:10.1086/338999. PMC 384943. PMID 11836649.
- ^ Kivisild T, Tolk HV, Parik J, Wang Y, Papiha SS, Bandelt HJ, Villems R (octubre de 2002). "Las extremidades emergentes y las ramitas del árbol de MtDNA de Asia Oriental". Biología molecular y evolución. 19 (10): 1737 –51. doi:10.1093/oxfordjournals.molbev.a003996. PMID 12270900.
- ^ Yao YG, Kong QP, Man XY, Bandelt HJ, Zhang YP (febrero de 2003). "Reconstruyendo la historia evolutiva de China: una cueva sobre las inferencias extraídas del ADN antiguo". Biología molecular y evolución. 20 2): 214–9. doi:10.1093/molbev/msg026. PMID 12598688.
- ^ Kong QP, Sun C, Wang HW, Zhao M, Wang WZ, Zhong L, Hao XD, Pan H, Wang SY, Cheng YT, Zhu CL, Wu SF, Liu LN, Jin JQ, Yao YG, Zhang YP (enero de 2011). "La proyección de mtDNA de gran escala revela una sorprendente complejidad matrilineal en Asia oriental y sus implicaciones para el peopling de la región". Biología molecular y evolución. 28 1): 513 –22. doi:10.1093/molbev/msq219. PMID 20713468.
- ^ Zhang Chi; Hsiao-Chun Hung (Fall 2008). "El Neolítico del Sur de China-Origin, Desarrollo y Dispersal". Asian Perspectives. 47 2): 299 –329. doi:10.1353/asi.0.0004. Hdl:10125/17291.
- ^ Shelach, Gideon (diciembre de 2000). "Las culturas neolíticas más antiguas del noreste de China: descubrimientos recientes y nuevas perspectivas en el comienzo de la agricultura". Journal of World Prehistory. 14 4): 363 –413. doi:10.1023/A:1011124209079.
- ^ Zhang Chi; Hsiao-chun Hun (marzo de 2010). "El surgimiento de la agricultura en el sur de China". Antigüedad. 84 (323): 11–25. doi:10.1017/S0003598X00099737.
- ^ Higham, C.F.W.; Xie Guangmao; Lin Qiang (junio de 2011). "La prehistoria de una zona de fricción: primeros agricultores y cazadores-recolectores en el sudeste asiático". Antigüedad. 85 (328): 529 –543. doi:10.1017/S0003598X00067922.
- ^ Aldenderfer, Mark; Zhang Yinong (marzo de 2004). "La prehistoria de la meseta tibetana al siglo VII d.C.: Perspectivas e Investigación de China y Occidente Desde 1950". Journal of World Prehistory. 18 1): 1 –55. doi:10.1023/B:JOWO.0000038657.79035.9e.