Piruvato descarboxilasa
La piruvato descarboxilasa es una enzima (EC 4.1.1.1) que cataliza la descarboxilación del ácido pirúvico a acetaldehído. También se le llama 2-oxoácido carboxilasa, alfa-cetoácido carboxilasa y descarboxilasa pirúvica. En condiciones anaeróbicas, esta enzima participa en el proceso de fermentación que se produce en las levaduras, especialmente del género Saccharomyces, para producir etanol por fermentación. También está presente en algunas especies de peces (incluidos los peces de colores y la carpa), donde les permite realizar la fermentación del etanol (junto con la fermentación del ácido láctico) cuando el oxígeno es escaso. La piruvato descarboxilasa inicia este proceso convirtiendo el piruvato en acetaldehído y dióxido de carbono. La piruvato descarboxilasa depende de los cofactores pirofosfato de tiamina (TPP) y magnesio. Esta enzima no debe confundirse con la enzima piruvato deshidrogenasa no relacionada, una oxidorreductasa (EC 1.2.4.1), que cataliza la descarboxilación oxidativa del piruvato a acetil-CoA.
Estructura
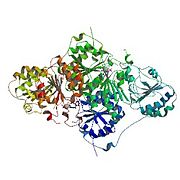
La piruvato descarboxilasa se presenta como un dímero de dímeros con dos sitios activos compartidos entre los monómeros de cada dímero. La enzima contiene una estructura beta-alfa-beta, que produce láminas beta paralelas. Contiene 563 subunidades de residuos en cada dímero; la enzima tiene fuertes atracciones entre monómeros, pero los dímeros interactúan libremente para formar un tetrámero suelto.
- Diagrama de caricatura de pyruvate decarboxylase monómero con TPP adjunto.
- Caricatura diagrama de pyruvate decarboxylase tetramer.
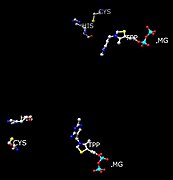
- Sitio activo de decarboxilasa de pyruvate con aminoácidos seleccionados: Glu-51, Glu-477, Asp-444 y Asp-28. También se muestran cofactores TPP y Mg2+.
- Posiciones de Sus residuos y Cis respecto al sitio activo (TPP y Mg) que participan en cambios de conformación cuando interactúan con sustrato de pyruvate.
Residuos del sitio activo
Cada sitio activo tiene 20 residuos de aminoácidos, incluido el ácido Glu-477, que interactúa con el anillo TPP, y Glu-51, que participa en la unión del cofactor. Estos glutamatos también estabilizan el ílido de TPP, actuando como donantes de protones. El entorno no polar alrededor de este Glu 477 no es polar, lo que contribuye a una pKa más alta de lo normal (la pKa normal de Glu y Asp es de alrededor de 4,6 en las proteínas pequeñas).
Los residuos lipófilos Ile-476, Ile-480 y Pro-26 contribuyen a la no polaridad del área alrededor de Glu-477. El único otro residuo cargado negativamente además de la coenzima TPP es el Asp-28, que también ayuda a aumentar el pKa de Glu-477. Por lo tanto, el entorno de la enzima debe permitir que la protonación del grupo gamma-carboxilo de Glu-477 esté alrededor de pH 6.
El anillo de aminopirimidina en TPP actúa como base, una vez en su forma imina, para extraer el protón C2 de TPP y formar el iluro nucleófilo. Esto debe ocurrir porque la enzima no tiene cadenas laterales básicas presentes para desprotonar el TPP C2. Una mutación en el sitio activo que involucra a estos Glu puede resultar en la ineficiencia o inactividad de la enzima. Esta inactividad se ha demostrado en experimentos en los que el N1' y/o faltan grupos 4'-amino. En el análisis de RMN, se ha determinado que cuando el TPP se une a la enzima junto con la piruvamida análoga del subestado, la tasa de formación de ílidos es mayor que la tasa normal de la enzima. Además, la tasa de mutación de Glu 51 a Gln reduce esta tasa significativamente.
Los residuos Asp-444 y Asp-28 se unen al Mg2+. Dos Cys-221 (a más de 20 Ångstroms de cada sitio) y His-92 desencadenan un cambio conformacional, que inhibe o activa la enzima dependiendo de la disponibilidad del sustrato. Si el sustrato unido al sitio activo es piruvato, la enzima se activa mediante un cambio conformacional en este sitio regulador. El cambio conformacional implica una adición 1,2 nucleofílica. Esta reacción, la formación de un tiocetal, transforma la enzima de su estado inactivo a activo.
La inhibición del sitio se realiza mediante una clase XC6H4CH=CHCOCOOH de inhibidores/análogos de sustrato, así como mediante el producto de la descarboxilación de dichos compuestos. como cinamaldehídos. Otros sitios nucleofílicos potenciales para el inhibidor incluyen Cys-152, Asp-28, His-114, His-115 y Gln-477.
La velocidad catalítica normal de la piruvato descarboxilasa es kcat = 10 s−1. Sin embargo, la velocidad de la enzima con una mutación Glu-51 a Gln es 1,7 s−1.
Grupo protésico TPP
El cofactor TPP es el grupo protésico de la enzima. El centro CH ubicado entre los átomos de azufre y nitrógeno en el anillo de tiazol es ácido. Tras la desprotonación, genera un iluro y adquiere carga negativa como carbanión. Este puede reaccionar como nucleófilo en el carbono cetónico del ácido pirúvico. Durante la descarboxilación del piruvato, el TPP estabiliza los intermediarios carbaniones como electrófilos mediante enlaces no covalentes. Específicamente, el nitrógeno piridílico N1' y el grupo 4'-amino de TPP son esenciales para la función catalítica del complejo enzima-TPP.
Mecanismo
La enzima divide el piruvato en dióxido de carbono y acetaldehído. La reacción se produce por ataque del carbono nucleofílico del tiazol al grupo ceto. El intermedio pierde dióxido de carbono, dando un enol, en un paso irreversible. Posteriormente se libera acetaldehído libre y se regenera el TPP.
Levadura
En la levadura, la piruvato descarboxilasa actúa de forma independiente durante la fermentación anaeróbica y libera el fragmento de 2 carbonos como acetaldehído más dióxido de carbono. La piruvato descarboxilasa crea los medios de eliminación de CO2, que la célula disipa. La enzima también sirve para crear etanol, que se utiliza como antibiótico para eliminar organismos competidores. La enzima es necesaria para ayudar en la descarboxilación de los alfa-cetoácidos porque se produce una acumulación de carga negativa en el átomo de carbono carbonilo en el estado de transición; por lo tanto, la enzima proporciona el entorno adecuado para que se encuentren el TPP y el alfa-cetoácido (piruvato).