Parvoviridae
parvovirus son una familia de virus animales que constituyen la familia Parvoviridae. Tienen genomas lineales de ADN monocatenario (ssDNA) que normalmente contienen dos genes que codifican una proteína iniciadora de la replicación, llamada NS1, y la proteína de la que está hecha la cápside viral. La porción de codificación del genoma está flanqueada por telómeros en cada extremo que forman bucles de horquilla que son importantes durante la replicación. Los viriones de parvovirus son pequeños en comparación con la mayoría de los virus, de 23 a 28 nanómetros de diámetro, y contienen el genoma encerrado en una cápside icosaédrica que tiene una superficie rugosa.
Los parvovirus ingresan a una célula huésped por endocitosis, viajando al núcleo donde esperan hasta que la célula ingrese a su etapa de replicación. En ese momento, el genoma no está recubierto y la parte codificante se replica. El ARN mensajero viral (ARNm) luego se transcribe y traduce, lo que da como resultado que NS1 inicie la replicación. Durante la replicación, las horquillas se despliegan, se replican y se repliegan repetidamente para cambiar la dirección de la replicación y progresar de un lado a otro a lo largo del genoma en un proceso llamado replicación de horquilla rodante que produce una molécula que contiene numerosas copias del genoma. Los genomas de ssDNA de la progenie se escinden de este concatémero y se empaquetan en cápsides. Los viriones maduros abandonan la célula por exocitosis o lisis.
Se cree que los parvovirus descienden de virus ssDNA que tienen genomas circulares que forman un bucle porque estos virus codifican una proteína iniciadora de replicación que está relacionada con NS1 y tienen un mecanismo de replicación similar. Otro grupo de virus llamado bidnavirus parece descender de los parvovirus. Dentro de la familia se reconocen tres subfamilias, 26 géneros y 126 especies. Parvoviridae es la única familia del orden Piccovirales, que es el único orden de la clase Quintoviricetes. Esta clase se asigna al filo Cossaviricota, que también incluye virus del papiloma, poliomavirus y bidnavirus.
Una variedad de enfermedades en animales son causadas por parvovirus. En particular, el parvovirus canino y el parvovirus felino causan enfermedades graves en perros y gatos, respectivamente. En cerdos, el parvovirus porcino es una de las principales causas de infertilidad. Los parvovirus humanos son menos graves, los dos más notables son el parvovirus B19, que causa una variedad de enfermedades, incluida la quinta enfermedad en los niños, y el bocavirus humano 1, que es una causa común de enfermedad aguda de las vías respiratorias, especialmente en niños pequeños. En medicina, los virus adenoasociados recombinantes (AAV) se han convertido en un vector importante para llevar genes al núcleo celular durante la terapia génica.
Los parvovirus animales se descubrieron por primera vez en la década de 1960, incluido el diminuto virus de ratones, que se utiliza con frecuencia para estudiar la replicación de parvovirus. También se descubrieron muchos AAV durante este período de tiempo y la investigación sobre ellos a lo largo del tiempo ha revelado su beneficio como forma de medicamento. El primer parvovirus humano patógeno que se descubrió fue el parvovirus B19 en 1974, que se asoció con varias enfermedades a lo largo de la década de 1980. Los parvovirus se clasificaron por primera vez como el género Parvovirus en 1971, pero se elevaron a la categoría de familia en 1975. Toman su nombre de la palabra latina parvum, que significa "pequeño". 39; o 'pequeño', en referencia al pequeño tamaño de los viriones del virus.
Genoma
Los parvovirus tienen genomas lineales de ADN monocatenario (ADNss) que tienen entre 4 y 6 kilobases (kb) de longitud. El genoma del parvovirus normalmente contiene dos genes, denominados gen NS/rep y gen VP/cap. El gen NS codifica la proteína no estructural (NS) NS1, que es la proteína iniciadora de la replicación, y el gen VP codifica la proteína viral (VP) de la que está hecha la cápside viral. NS1 contiene un dominio de endonucleasa de la superfamilia HUH cerca de su extremo N-terminal, que contiene actividad de unión específica del sitio y actividad de corte específica del sitio, y un dominio de helicasa de la superfamilia 3 (SF3) hacia el extremo C-terminal. La mayoría de los parvovirus contienen un dominio de activación transcripcional cerca del extremo C que aumenta la transcripción de los promotores virales, así como marcos de lectura abiertos alternos o superpuestos que codifican una pequeña cantidad de proteínas de apoyo involucradas en diferentes aspectos del ciclo de vida viral.
La parte codificante del genoma está flanqueada en cada extremo por secuencias terminales de aproximadamente 116 a 550 nucleótidos (nt) de longitud que consisten en palíndromos imperfectos plegados en estructuras de bucles de horquilla. Estos bucles de horquilla contienen la mayor parte de la información que actúa en cis requerida para la replicación y el empaquetamiento del ADN y actúan como bisagras durante la replicación para cambiar la dirección de la replicación. Cuando el genoma se convierte en formas de doble cadena, se crean sitios de origen de replicación que involucran secuencias en y adyacentes a las horquillas.
Las hebras de ADN genómico en viriones maduros pueden ser de sentido positivo o negativo. Esto varía de una especie a otra, ya que algunos tienen preferencia por empaquetar hilos de una polaridad, otros empaquetan proporciones variables y otros empaquetan ambos sentidos en proporciones iguales. Estas preferencias reflejan la eficiencia con la que se sintetizan las cadenas de progenie, que a su vez refleja la eficiencia de los sitios de origen de replicación específicos. El extremo 3′ (generalmente pronunciado "extremo principal tres") de una hebra de sentido negativo, y el extremo 5′ (generalmente pronunciado "extremo principal cinco") de una cadena de sentido positivo, se denomina extremo izquierdo, y el extremo 5′ de la hebra de sentido negativo, y el extremo 3′ de una hebra de sentido positivo, se denomina extremo derecho.
Estructura
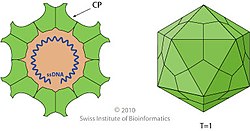
Los viriones de parvovirus tienen entre 23 y 28 nanómetros (nm) de diámetro y consisten en el genoma encerrado dentro de una cápside de forma icosaédrica con una superficie rugosa. La cápside está compuesta por 60 cadenas polipeptídicas estructuralmente equivalentes derivadas del extremo C-terminal de la secuencia de una proteína VP, que se entrelazan extensamente para formar un icosaedro con 60 unidades triangulares superficiales asimétricas. Estas unidades tienen simetría radial de 3 pliegues en dos vértices y simetría radial de 5 pliegues en uno, con simetría radial de 2 pliegues en la línea opuesta al vértice de 5 pliegues y una pared de pliegue circular de 2/5 que rodea el punto del 5 -doblar vértice. Existen veinte vértices de 3 pliegues, treinta líneas de 2 pliegues y doce vértices de 5 pliegues por cápside, este último correspondiente a los 12 vértices del icosaedro.
Las características típicas de la superficie de la cápside incluyen depresiones en cada eje de 2 pliegues, protuberancias elevadas que rodean los ejes de 3 pliegues y proyecciones cilíndricas elevadas hechas de cinco barriles beta rodeados por depresiones en forma de cañón en los ejes de 5 pliegues. Cada uno de estos cilindros contiene potencialmente una abertura para conectar el exterior de la cápside con el interior, que media la entrada y salida del genoma. Alrededor de 20 nucleótidos del extremo 5' del genoma pueden permanecer expuestos fuera de la cápside que llevan una copia de NS1 unida al extremo 5', lo que es el resultado de cómo se sintetiza y empaqueta el genoma.
Diferentes tamaños de la proteína VP se expresan para diferentes parvovirus, los más pequeños, VP2-5, se expresan con mayor frecuencia que los de gran tamaño, VP1. Los VP más pequeños comparten un extremo C común con diferentes longitudes del extremo N debido al truncamiento. Para VP1, el extremo N se extiende para contener regiones importantes en el ciclo de replicación y se incorpora a la cápside, típicamente de 5 a 10 por cápside, con el extremo C común responsable de ensamblar las cápsidas.
Cada monómero de VP contiene una estructura de barril beta central llamada motivo de rollo de gelatina de ocho hebras dispuestas en dos láminas beta antiparalelas adyacentes, denominadas CHEF y BIDG después de las hebras individuales; esta última forma la superficie interior de la cápside. Las hebras beta individuales están conectadas por bucles que tienen longitud, secuencia y conformación variables, y la mayoría de estos bucles se extienden hacia la superficie exterior, dando a los parvovirus su superficie rugosa única. Los parvovirus relacionados comparten sus topologías de superficie y los pliegues de la proteína VP en mayor medida que sus identidades de secuencia, por lo que la estructura de la cápside y la proteína de la cápside son indicadores útiles de la filogenia.
Ciclo de vida
Los parvovirus ingresan a las células por endocitosis, utilizando una variedad de receptores celulares para unirse a la célula huésped. En los endosomas, muchos parvovirus experimentan un cambio en la conformación, de modo que el dominio de la fosfolipasa A2 (PLA2) en los terminales N de VP1 queda expuesto para que el virión pueda penetrar las membranas de la bicapa lipídica. El tráfico intracelular de viriones varía, pero los viriones finalmente llegan al núcleo, dentro del cual el genoma no está revestido de la cápside. Con base en estudios del virus diminuto de ratones (MVM), el genoma se expulsa de la cápside en una dirección de 3′ a 5′ desde una de las aberturas de la cápside, dejando el extremo 5′ del ADN unido al cápside
Los parvovirus carecen de la capacidad de inducir a las células a su etapa de replicación del ADN, llamada fase S, por lo que deben esperar en el núcleo hasta que la célula huésped entre en la fase S por sí sola. Esto hace que las poblaciones de células que se dividen rápidamente, como las células fetales, sean un entorno excelente para los parvovirus. Los virus adenoasociados (AAV) dependen de virus auxiliares, que pueden ser un adenovirus o un herpesvirus, ya que la coinfección altera el entorno celular para permitir la replicación. En ausencia de coinfección, el genoma de AAV se integra en el genoma de la célula huésped hasta que se produce la coinfección. Las células infectadas que entran en la fase S se ven obligadas a sintetizar ADN viral y no pueden salir de la fase S. Los parvovirus establecen focos de replicación en el núcleo que crecen progresivamente a medida que avanza la infección.
Una vez que una célula entra en la fase S y el genoma no está recubierto, una polimerasa de ADN huésped usa el extremo 3′ de la horquilla 3′ como cebador para sintetizar una hebra de ADN complementaria para la parte codificante del genoma, que es conectado al extremo 5′ de la horquilla de 5′. El ARN mensajero (ARNm) que codifica NS1 es luego transcrito del genoma por la ADN polimerasa, protegido y poliadenilado, y traducido por los ribosomas del huésped para sintetizar NS1. Si las proteínas están codificadas en múltiples marcos colineales, entonces se pueden usar cortes y empalmes alternativos, iniciación de traducción subóptima o escaneo con fugas para traducir diferentes productos génicos.
Los parvovirus replican su genoma a través de la replicación en horquilla rodante, una forma unidireccional de replicación de ADN con desplazamiento de cadena que es iniciada por NS1. La replicación comienza una vez que NS1 se une y hace una muesca en un sitio de origen de replicación en la molécula de ADN dúplex al final de una horquilla. El mellado libera el extremo 3′ de la hebra mellada como un hidroxilo libre (-OH) para cebar la síntesis de ADN y el NS1 permanece unido al extremo 5′. La muesca hace que la horquilla adyacente se despliegue en una forma lineal extendida. En el 3′-OH, se establece una horquilla de replicación utilizando la actividad helicasa de NS1 y el telómero extendido se replica mediante la ADN polimerasa. Luego, las dos cadenas de telómeros se repliegan sobre sí mismas a sus configuraciones originales, lo que reposiciona la horquilla de replicación para cambiar las plantillas a la otra cadena y moverse en la dirección opuesta hacia el otro extremo del genoma.
Los parvovirus varían en si los extremos son similares o iguales, llamados parvovirus homoteloméricos, o diferentes, llamados parvovirus heteroteloméricos. En general, los parvovirus homoteloméricos, como AAV y B19, replican ambos extremos de su genoma a través del proceso antes mencionado, llamado resolución terminal, y sus secuencias de horquilla están contenidas dentro de repeticiones terminales más grandes (invertidas). Los virus heteroteloméricos, como el virus del minuto de los ratones (MVM), replican un extremo por resolución terminal y el otro extremo a través de un proceso asimétrico llamado resolución de unión para que se pueda copiar la orientación correcta del telómero.
Durante la resolución de la unión asimétrica, los telómeros dúplex de forma extendida se repliegan sobre sí mismos en una forma cruciforme. NS1 corta un sitio de origen de replicación en la hebra inferior del brazo derecho del cruciforme, lo que lleva a que el brazo inferior del cruciforme se despliegue en su forma lineal extendida. Una horquilla de replicación establecida en el sitio de la muesca baja por el antebrazo extendido para copiar la secuencia del antebrazo. Luego, las dos hebras del brazo inferior se vuelven a plegar para cambiar la posición de la horquilla de replicación y regresar hacia el otro extremo, desplazando la hebra superior en el proceso.
El patrón de ida y vuelta, de extremo a extremo, de la replicación en horquilla rodante produce un concatémero que contiene múltiples copias del genoma. NS1 hace muescas periódicamente en esta molécula y, a través de una combinación de resolución terminal y resolución de unión, las hebras individuales del genoma se eliminan del concatémero. Los genomas extirpados pueden reciclarse para rondas adicionales de replicación o empaquetarse en cápsides de progenie. La traducción de ARNm que contiene proteínas VP conduce a la acumulación de proteínas de la cápside en el núcleo que se ensamblan en estas cápsidas vacías.
Los genomas se encapsulan en uno de los vértices de la cápside a través de un portal, posiblemente el opuesto al portal utilizado para expulsar el genoma. Una vez que se han construido los viriones completos, pueden exportarse desde el núcleo al exterior de la célula antes de la desintegración del núcleo. La interrupción del entorno de la célula huésped también puede ocurrir más adelante en la infección. Esto da como resultado la lisis celular mediante necrosis o apoptosis, que libera viriones al exterior de la célula.
Evolución
Se cree que los parvovirus descienden de los virus ssDNA que tienen un genoma circular que forma un bucle y que se replican a través de la replicación en círculo rodante, que es similar a la replicación en horquilla rodante. Estos virus ssDNA circulares codifican una proteína iniciadora de la replicación que está relacionada y posee muchas de las mismas características que la proteína iniciadora de la replicación de los parvovirus, como el dominio de endonucleasa HUH y el dominio de helicasa SF3. En contraste con estas otras proteínas iniciadoras de la replicación, NS1 muestra solo rastros vestigiales de poder realizar la ligadura, que es una parte clave de la replicación del círculo rodante. La familia Bidnaviridae, que también son virus ssDNA lineales, parece descender de un parvovirus que tenía su genoma integrado en el genoma de un polinton, un tipo de transposón de ADN relacionado con los virus en el reino varidnaviria.
Según el análisis filogenético de la helicasa SF3, los parvovirus se dividen en dos ramas al principio de su historia evolutiva, una de las cuales contiene virus asignados a la subfamilia Hamaparvovirinae. La otra rama se dividió en dos sublinajes que constituyen las otras dos subfamilias, Densovirinae y Parvovirinae. Los parvovirus del linaje Hamaparvovirinae son probablemente todos heteroteloméricos, Densovirinae son exclusivamente homoteloméricos y Parvovirinae varía. Las secuencias de telómeros tienen una complejidad y diversidad significativas, lo que sugiere que muchas especies las han cooptado para realizar funciones adicionales. También se considera que los parvovirus tienen altas tasas de mutaciones y recombinaciones genéticas.
Clasificación
Los parvovirus constituyen la familia Parvoviridae. La familia es la única familia del orden Piccovirales, que es el único orden de la clase Quintoviricetes. La clase Quintoviricetes pertenece al filo Cossaviricota, que también incluye papilomavirus, poliomavirus y bidnavirus. Cossaviricota se incluye en el reino Shotokuvirae, que se asigna al reino Monodnaviria. Parvoviridae pertenece al Grupo II: virus ssDNA en el sistema de clasificación de Baltimore, que agrupa a los virus según su forma de síntesis de ARNm. Dentro de Parvoviridae, se reconocen tres subfamilias, 26 géneros y 126 especies a partir de 2020 (-virinae denota subfamilia y -virus denota género):
- Densovirinae (11 géneros, 21 especies)
- Aquambidensovirus (3 especies)
- Blattambidensovirus (1 especie)
- Diciambidensovirus (1 especie)
- Hemiambidensovirus (2 especies)
- Iteradensovirus (5 especies)
- Miniambidensovirus (1 especie)
- Muscodensovirus (1 especie)
- Pefuambidensovirus (1 especie)
- Protoambidensovirus (2 especies)
- Scindoambidensovirus (3 especies)
- Tetuambidensovirus (1 especie)
- Hamaparvovirinae (5 géneros, 21 especies)
- Brevihamaparvovirus (2 especies)
- Chaphamaparvovirus (16 especies)
- Hepanhamaparvovirus (1 especie)
- Ichthamaparvovirus (1 especie)
- Penstylhamaparvovirus (1 especie)
- Parvovirinae (10 géneros, 84 especies)
- Amdoparvovirus (5 especies)
- Artiparvovirus (1 especie)
- Aveparvovirus (3 especies)
- Bocaparvovirus (28 especies)
- Copiparvovirus (7 especies)
- Dependoparvovirus (11 especies)
- Erythroparvovirus (7 especies)
- Loriparvovirus (1 especie)
- Protoparvovirus (15 especies)
- Tetraparvovirus (6 especies)
Los parvovirus se asignan a la misma especie si comparten al menos el 85 % de sus identidades de secuencia de proteínas. Las especies se agrupan en un género según la filogenia de los dominios helicasa NS1 y SF3, así como la similitud de la identidad y la cobertura de la secuencia NS1. Si no se cumplen estos criterios, aún se pueden establecer los géneros siempre que se admita la ascendencia común. Las tres subfamilias se distinguen en función de la filogenia del dominio helicasa SF3, que corresponde al rango de huéspedes: los virus de Densovirinae infectan a los invertebrados, los virus de Hamaparvovirinae infectan a los invertebrados y vertebrados, y los virus en Parvovirinae infectan a los vertebrados.
Enfermedad
En los seres humanos, los parvovirus más destacados que causan enfermedades son el parvovirus B19 y el bocavirus humano 1. La infección por B19 suele ser asintomática, pero puede manifestarse de diversas formas, incluida la quinta enfermedad con su erupción característica en niños, anemia persistente en personas inmunodeprimidas y en personas que tienen hemoglobinopatías subyacentes, crisis aplásicas transitorias, hidropesía fetal en mujeres embarazadas y artropatía. El bocavirus humano 1 es una causa común de infección aguda del tracto respiratorio, especialmente en niños pequeños, siendo las sibilancias un síntoma común. Otros parvovirus asociados con diferentes enfermedades en humanos incluyen el parvovirus humano 4 y el bufavirus humano, aunque la forma en que estos virus causan la enfermedad no está clara.
Los virus que infectan a los carnívoros del género Protoparvovirus, a diferencia de los parvovirus humanos, son más peligrosos para la vida. El parvovirus canino causa una enfermedad grave en los perros, siendo el síntoma más común la enteritis hemorrágica, con una tasa de mortalidad de hasta el 70 % en los cachorros, pero por lo general menos del 1 % en los adultos. El parvovirus felino, un virus estrechamente relacionado, también causa una enfermedad grave en los gatos junto con panleucopenia. En los cerdos, el parvovirus porcino es una de las principales causas de infertilidad, ya que la infección conduce con frecuencia a la muerte del feto.
Uso en medicina
Los virus adenoasociados se han convertido en un vector importante para la terapia génica destinada a tratar enfermedades genéticas, como las causadas por una sola mutación. El AAV recombinante (rAAV) contiene una cápside viral pero carece de un genoma viral completo. En cambio, el ácido nucleico típico empaquetado en la cápside contiene una región promotora, el gen de interés y una región terminadora, todo contenido dentro de dos repeticiones terminales invertidas derivadas del genoma viral. rAAV actúa esencialmente como un contenedor que puede atravesar la membrana celular y entregar su carga de ácido nucleico al núcleo.
Historia
Los parvovirus se descubrieron relativamente tarde en comparación con otras familias de virus prominentes, posiblemente debido a su pequeño tamaño. A fines de la década de 1950 y 1960, se descubrió una variedad de parvovirus animales, incluido el virus diminuto de ratones, que desde entonces se ha utilizado ampliamente para estudiar la replicación en horquilla. También se descubrieron muchos AAV durante este período de tiempo y la investigación sobre ellos condujo a su primer uso en terapia génica en la década de 1980. Con el tiempo, las mejoras en aspectos como el diseño de vectores hicieron que determinados productos de terapia génica de AAV alcanzaran eficacia clínica en 2008 y fueran aprobados en los años siguientes.
En 1974, Yvonne Cossart, et al. descubrieron el primer parvovirus humano patógeno. Cuando se analizó el antígeno de superficie del virus de la hepatitis B, una muestra de suero arrojó resultados anómalos y, con microscopía electrónica, se demostró que contenía un virus parecido a los parvovirus animales. Este virus se denominó B19 después de la codificación de la muestra de suero, número 19 en el panel B. Posteriormente, el Comité Internacional de Taxonomía de Virus (ICTV) reconoció a B19 como una especie en 1985, y a lo largo de la década de 1980 se asoció cada vez más con varios enfermedades.
En el primer informe del ICTV de 1971, los parvovirus se agruparon en el género Parvovirus. Fueron elevados al rango de familia en 1975 y permanecieron sin asignar a taxones superiores hasta 2019, cuando fueron asignados a taxones superiores hasta el reino de rango más alto. La familia se reorganizó en 2019, partiendo de la "tradicional" distinción invertebrado-vertebrado entre Densovirinae y Parvovirinae y, en cambio, distinguir las subfamilias en función de la filogenia helicasa, lo que lleva al establecimiento de una nueva subfamilia, Hamaparvovirinae.
Etimología
Los parvovirus toman su nombre del latín parvus o parvum, que significa pequeño o diminuto, en referencia al pequeño tamaño de los viriones de parvovirus en comparación con la mayoría de los otros virus. En el nombre de la familia Parvoviridae, -viridae es el sufijo utilizado para las familias de virus. El orden Piccovirales toma la primera parte de su nombre de la palabra italiana piccolo, que significa pequeño, y la segunda parte es el sufijo utilizado para virus pedidos. La clase Quintoviricetes toma la primera parte de su nombre de la palabra gallega quinto, que significa quinto, en referencia a la quinta enfermedad (eritema infeccioso) causada por parvovirus B19 y viricetes, el sufijo utilizado para las clases de virus.
Contenido relacionado
Economía de la biodiversidad
Lista de familias de anuros
Ansiolítico