Partícula de ARN que reconoce señales
El ARN de partícula de reconocimiento de señales (también conocido como ARN 7SL, 6S, ffs o 4.5S) es parte del complejo ribonucleoproteico de partícula de reconocimiento de señales (SRP). La SRP reconoce el péptido señal y se une al ribosoma, deteniendo la síntesis de proteínas. El receptor de SRP es una proteína que está incrustada en una membrana y que contiene un poro transmembrana. Cuando el complejo SRP-ribosoma se une al receptor de SRP, la SRP libera el ribosoma y se aleja. El ribosoma reanuda la síntesis de proteínas, pero ahora la proteína se mueve a través del poro transmembrana del receptor de SRP.
De esta manera, la SRP dirige el movimiento de las proteínas dentro de la célula para unirse a un poro transmembrana que permite que la proteína cruce la membrana hasta donde se la necesita. Los componentes de ARN y proteína de este complejo están altamente conservados, pero varían entre los diferentes reinos de la vida.
La familia común SINE Alu probablemente se originó a partir de un gen de ARN 7SL después de la eliminación de una secuencia central.
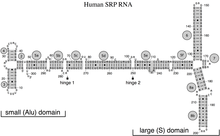
El SRP eucariota consta de un ARN 7S de 300 nucleótidos y seis proteínas: SRP 72, 68, 54, 19, 14 y 9. El SRP arqueal consta de un ARN 7S y homólogos de las proteínas eucariotas SRP19 y SRP54. Los ARN 7S eucariotas y arqueales tienen estructuras secundarias muy similares.
En la mayoría de las bacterias, el SRP consiste en una molécula de ARN (4.5S) y la proteína Ffh (un homólogo de la proteína eucariota SRP54). Algunas bacterias Gram-positivas (p. ej., Bacillus subtilis) tienen un ARN SRP más largo, similar al eucariota, que incluye un dominio Alu.
En eucariotas y arqueas, ocho elementos helicoidales se pliegan en los dominios Alu y S, separados por una región de enlace larga. Se cree que el dominio Alu media la función de retardo de la elongación de la cadena peptídica del SRP. La hélice universalmente conservada que interactúa con el dominio M de SRP54 media el reconocimiento de la secuencia señal. Se cree que el complejo SRP19-hélice 6 está involucrado en el ensamblaje de SRP y estabiliza la hélice 8 para SRP54. Los humanos tienen tres genes de ARN SRP funcionales, convenientemente denominados RN7SL1, RN7SL2 y RN7SL3. Se sabe que el genoma humano en particular contiene una gran cantidad de secuencias relacionadas con el ARN SRP, incluidas las repeticiones Alu.
Discovery
El ARN SRP se detectó por primera vez en partículas de virus de ARN oncogénico (ocorna) aviar y murino. Posteriormente, se descubrió que el ARN SRP era un componente estable de células HeLa no infectadas, donde se asociaba con fracciones de membrana y polisoma. En 1980, los biólogos celulares purificaron a partir del páncreas canino una proteína de reconocimiento de señal 11S (afortunadamente también abreviada como SRP) que promovía la translocación de proteínas secretoras a través de la membrana del retículo endoplasmático. Luego se descubrió que SRP contenía un componente de ARN. La comparación de los genes de ARN SRP de diferentes especies reveló que la hélice 8 del ARN SRP estaba altamente conservada en todos los dominios de la vida. Las regiones cercanas a los extremos 5′ y 3′ del ARN SRP de mamíferos son similares a la familia dominante Alu de secuencias repetitivas medias del genoma humano. Actualmente se sabe que el ADN Alu se originó a partir del ARN SRP por escisión del fragmento central específico del ARN SRP (S), seguida de transcripción inversa e integración en múltiples sitios de los cromosomas humanos. También se han identificado ARN SRP en algunos orgánulos, por ejemplo, en los SRP plastídicos de muchos organismos fotosintéticos y en la región espaciadora transcrita interna del ribosoma nuclear de varios hongos ectomicorrízicos.
Transcripción y procesamiento
Los ARN SRP eucariotas se transcriben a partir del ADN por la ARN polimerasa III (Pol III). La ARN polimerasa III también transcribe los genes del ARN ribosómico 5S, ARNt, ARN 7SK y ARN espliceosómico U6. Los promotores de los genes del ARN SRP humano incluyen elementos ubicados aguas abajo del sitio de inicio de la transcripción. Los promotores del ARN SRP vegetal contienen un elemento estimulador aguas arriba (USE) y una caja TATA. Los genes del ARN SRP de levadura tienen una caja TATA y secuencias promotoras intragénicas adicionales (denominadas bloques A y B) que desempeñan un papel en la regulación de la transcripción del gen SRP por la Pol III. En las bacterias, los genes se organizan en operones y se transcriben por la ARN polimerasa. El extremo 5' del ARN SRP pequeño (4,5S) de muchas bacterias es cortado por la ARNasa P. Los extremos del ARN SRP de Bacillus subtilis son procesados por la ARNasa III. Hasta el momento, no se han observado intrones de ARN SRP.
Función
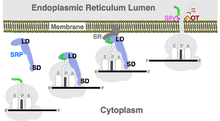
Translocación co-translacional
El ARN SRP es una parte integral del dominio pequeño y del dominio grande del SRP. La función del dominio pequeño es retrasar la traducción de la proteína hasta que el SRP unido al ribosoma tenga la oportunidad de asociarse con el receptor SRP residente en la membrana (SR). Dentro del dominio grande, el ARN SRP del SRP cargado con péptido señal promueve la hidrólisis de dos moléculas de guanosina trifosfato (GTP). Esta reacción libera el SRP del receptor SRP y del ribosoma, lo que permite que la traducción continúe y que la proteína ingrese al translocón. La proteína atraviesa la membrana de manera co-traduccional (durante la traducción) y entra en otro compartimento celular o en el espacio extracelular. En los eucariotas, el objetivo es la membrana del retículo endoplasmático (RE). En Archaea, el SRP entrega proteínas a la membrana plasmática. En las bacterias, el SRP incorpora principalmente proteínas en la membrana interna.
Transporte posterior a la traducción
El SRP también participa en la clasificación de proteínas una vez que se ha completado su síntesis (clasificación de proteínas postraduccional). En los eucariotas, las proteínas ancladas a la cola que poseen una secuencia de inserción hidrofóbica en su extremo C son enviadas al retículo endoplasmático (RE) por el SRP. De manera similar, el SRP ayuda postraduccionalmente en la importación de proteínas codificadas en el núcleo a la membrana tilacoide de los cloroplastos.
Estructura
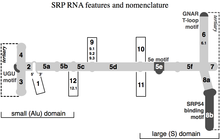
En 2005, una nomenclatura para todos los ARN SRP propuso un sistema de numeración de 12 hélices. Las secciones de la hélice se nombran con un sufijo de letra minúscula (p. ej., 5a). Las inserciones, o "ramas" de la hélice, se numeran con puntos (p. ej., 9.1 y 12.1).
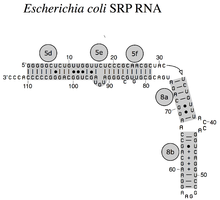
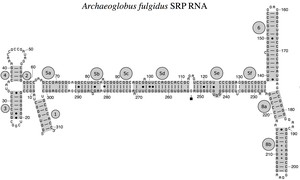
El ARN SRP abarca un amplio espectro filogenético en cuanto a tamaño y número de sus características estructurales (consulte los ejemplos de estructura secundaria del ARN SRP, a continuación). Los ARN SRP funcionales más pequeños se han encontrado en micoplasmas y especies relacionadas. El ARN SRP de Escherichia coli (también llamado ARN 4.5S) está compuesto por 114 residuos de nucleótidos y forma un tallo-bucle de ARN. La bacteria grampositiva Bacillus subtilis codifica un ARN SRP 6S más grande que se parece a los homólogos Archaeal pero carece de la hélice 6 del ARN SRP. Los ARN SRP Archaeal poseen las hélices 1 a 8, carecen de la hélice 7 y se caracterizan por una estructura terciaria que involucra los bucles apicales de la hélice 3 y la hélice 4. Los ARN SRP eucariotas carecen de la hélice 1 y contienen una hélice 7 de tamaño variable. Algunos ARN SRP de protozoos tienen hélices 3 y 4 reducidas. Los ARN SRP de ascomicetos tienen un dominio pequeño totalmente reducido y carecen de las hélices 3 y 4. Los ARN SRP más grandes conocidos hasta la fecha se encuentran en las levaduras (Saccharomycetes) que adquirieron las hélices 9 a 12 como inserciones en la hélice 5, así como una hélice 7 extendida. Las plantas con semillas expresan numerosos ARN SRP altamente divergentes.
Motifs
Se han identificado cuatro características conservadas (motivos) (mostrados en la Figura en gris oscuro): (1) el motivo de unión SRP54, (2) el motivo tetraloop Helix 6 GNAR, (3) el motivo 5e y (4) el motivo UGU(NR).
SRP54 binding
El bucle asimétrico entre las secciones helicoidales 8a y 8b y la sección 8b con pares de bases adyacentes son una propiedad destacada de cada ARN SRP. La sección helicoidal 8b contiene pares de bases que no son de Watson-Crick, lo que contribuye a la formación de un surco menor aplanado en el ARN adecuado para la unión de la proteína SRP54 (llamada Ffh en las bacterias). El bucle apical de la hélice 8 contiene cuatro, cinco o seis residuos, según la especie. Tiene una guanosina altamente conservada como el primer residuo del bucle y una adenosina como el último. Esta característica es necesaria para la interacción con el tercer residuo de adenosina del motivo tetraloop GNAR de la hélice 6.
Helix 6 GNAR tetraloop
Los ARN SRP de eucariotas y arqueas tienen un tetraloop GNAR (N es para cualquier nucleótido, R es para una purina) en la hélice 6. Su residuo de adenosina conservado es importante para la unión de la proteína SRP19. Esta adenosina realiza una interacción terciaria con otro residuo de adenosina ubicado en el bucle apical de la hélice 8.
5e
Los 11 nucleótidos del motivo 5e forman cuatro pares de bases que están interrumpidos por un bucle de tres nucleótidos. En los eucariotas, el primer nucleótido del bucle es una adenosina, necesaria para la unión de la proteína SRP72.
UGU(NR)
El motivo UGU(NR) conecta las hélices 3 y 4 en el dominio SRP pequeño (Alu). Los ARN SRP fúngicos que carecen de las hélices 3 y 4 contienen el motivo dentro del bucle de la hélice 2. Es importante en la unión del heterodímero de la proteína SRP9/14 como parte de un giro en U del ARN.
Secundaria
terciario
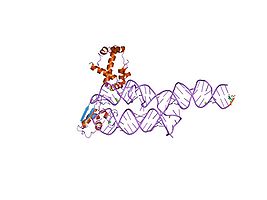
Se han utilizado la cristalografía de rayos X, la resonancia magnética nuclear (RMN) y la criomicroscopía electrónica (crio-EM) para determinar la estructura molecular de porciones de los ARN SRP de varias especies. Las estructuras PDB disponibles muestran la molécula de ARN libre o unida a una o más proteínas SRP.
Proteínas vinculantes
Una o más proteínas SRP se unen al ARN SRP para ensamblar el SRP funcional. Las proteínas SRP se nombran de acuerdo con su masa molecular aproximada medida en kilodalton. La mayoría de las SRP bacterianas están compuestas de ARN SRP y SRP54 (también llamado Ffh por "cincuenta y cuatrohomolog"). El SRP arqueal contiene las proteínas SRP54 y SRP19. En eucariotas, el ARN SRP se combina con las proteínas SRP importadas SRP9/14, SRP19 y SRP68/72 en una región del nucléolo. Este pre-SRP se transporta al citosol donde se une a la proteína SRP54. Las estructuras moleculares de las proteínas SRP9/14, SRP19 o SRP54 libres o unidas al ARN SRP se conocen con alta resolución.
SRP9 y SRP14
SRP9 y SRP14 están estructuralmente relacionados y forman el heterodímero SRP9/14 que se une al ARN SRP del dominio pequeño (Alu). El SRP de levadura carece de SRP9 y contiene la proteína de unión estructuralmente relacionada SRP21. El SRP14 de levadura forma homodímeros en cristal y no se une a Alu. SRP9/14 está ausente en el SRP del tripanosoma que, en cambio, posee una molécula similar al ARNt.
SRP19
La SRP19 se encuentra en la SRP de eucariotas y arqueas. Su función principal es preparar el ARN de la SRP para la unión de SRP54, SRP68 y SRP72 mediante la disposición adecuada de las hélices 6 y 8 del ARN de la SRP. La SRP de levadura contiene Sec65p, un homólogo más grande de la SRP19.
SRP54
La proteína SRP54 (denominada Ffh en las bacterias) es un componente esencial de cada SRP. Está compuesta por tres dominios funcionales: el dominio N-terminal (N), el dominio GTPasa (G) y el dominio rico en metionina (M).
SRP68 y SRP72
Las proteínas SRP68 y SRP72 son constituyentes estructuralmente no relacionados del dominio grande del SRP eucariota. Forman un heterodímero SRP68/72 estable. Se ha demostrado que aproximadamente un tercio de la proteína SRP68 humana se une al ARN del SRP. Una región relativamente pequeña ubicada cerca del extremo C de SRP72 se une al motivo 5e del ARN del SRP.
Referencias
- ^ a b c GRCh38: Ensembl release 89: ENSG00000276168 – Ensembl, May 2017
- ^ "Human PubMed Referencia:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b Ullu E, Tschudi C (1984). "Las secuencias de alu se procesan genes de ARN 7SL". Naturaleza. 312 (5990): 171–172. Bibcode:1984Natur.312..171U. doi:10.1038/312171a0. PMID 6209580. S2CID 4328237.
- ^ a b Rosenblad MA, Larsen N, Samuelsson T, Zwieb C (2009). "Kinship in the SRP RNA family". RNA Biology. 6 5): 508 –516. doi:10.4161/rna.6.5.9753. PMID 19838050.
- ^ a b c d Regalia M, Rosenblad MA, Samuelsson T (agosto de 2002). "Predicción de genes RNA de la partícula de reconocimiento de señal". Nucleic Acids Research. 30 (15): 3368–3377. doi:10.1093/nar/gkf468. PMC 137091. PMID 12140321.
- ^ a b c Wild K, Weichenrieder O, Strub K, Sinning I, Cusack S (febrero de 2002). "Hacia la estructura de la partícula de reconocimiento de señales mamíferas". Opinión actual en Biología Estructural. 12 1): 72–81. doi:10.1016/S0959-440X(02)00292-0. PMID 11839493.
- ^ a b c Batey RT, Rambo RP, Lucast L, Rha B, Doudna JA (Febrero 2000). "La estructura de cristal del núcleo ribonucleoproteína de la partícula de reconocimiento de señal". Ciencia. 287 (5456): 1232–1239. Bibcode:2000Sci...287.1232B. doi:10.1126/science.287.5456.1232. PMID 10678824.
- ^ Batey RT, Sagar MB, Doudna JA (marzo de 2001). "Análisis estructural y energético del reconocimiento del ARN por una proteína universalmente conservada de la partícula de reconocimiento de señales". Journal of Molecular Biology. 307 1): 229 –246. doi:10.1006/jmbi.2000.4454. PMID 11243816.
- ^ Bishop JM, Levinson WE, Sullivan D, Fanshier L, Quintrell N, Jackson J (diciembre de 1970). "Los ARN de peso molecular bajo del virus del sarcoma Rous. II. El 7 S ARN". Virología. 42 4): 927 –937. doi:10.1016/0042-6822(70)90341-7. PMID 4321311.
- ^ Walker TA, Pace NR, Erikson RL, Erikson E, Behr F (septiembre de 1974). "El ARN 7S común a los oncornavirus y las células normales se asocia con los poliribólicos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 71 (9): 3390 –3394. Bibcode:1974PNAS...71.3390W. doi:10.1073/pnas.71.9.3390. PMC 433778. PMID 4530311.
- ^ Zieve G, Penman S (mayo de 1976). "Pequeñas especies de ARN de la célula HeLa: metabolismo y localización subcelular". Celular. 8 1): 19 –31. doi:10.1016/0092-8674(76)90181-1. PMID 954090. S2CID 26928799.
- ^ Walter P, Ibrahimi I, Blobel G (noviembre de 1981). "Translocación de proteínas a través del reticulum endoplasmático. I. La proteína de reconocimiento de signos (SRP) se une a los polímeros mezclados con el vitro sintetizador de proteína secretor". The Journal of Cell Biology. 91 (2 Pt 1): 545 –550. doi:10.1083/jcb.91.2.545. PMC 2111968. PMID 7309795.
- ^ Walter P, Blobel G (octubre de 1982). "La partícula de reconocimiento de signos contiene un ARN de 7S esencial para la translocación de proteínas en el reticulum endoplasmático". Naturaleza. 299 (5885): 691 –698. Bibcode:1982Natur.299..691W. doi:10.1038/299691a0. PMID 6181418. S2CID 4237513.
- ^ Larsen N, Zwieb C (enero de 1991). "SRP-RNA secuencia alineación y estructura secundaria". Nucleic Acids Research. 19 2): 209 –215. doi:10.1093/nar/19.2.209. PMC 333582. PMID 1707519.
- ^ Ullu E, Murphy S, Melli M (mayo de 1982). "El ARN Humano 7SL consiste en una secuencia de 140 nucleótidos medianos-repetitivos insertados en una secuencia alu". Celular. 29 1): 195–202. doi:10.1016/0092-8674(82)90103-9. PMID 6179628. S2CID 12709599.
- ^ Rosenblad MA, Samuelsson T (noviembre 2004). "Identificación de los genes de ARN de las partículas de reconocimiento de señales de cloroplast". Fisiología celular. 45 (11): 1633–1639. doi:10.1093/pcp/pch185. PMID 15574839.
- ^ Alm Rosenblad M, Martin MP, Tedersoo T, Ryberg MR, Larsson E, Wurzbacher C, Abarenkov K, Nilsson RH (2016). "Detección de partículas de reconocimiento de señal (SRP) ARNs en el espaciamiento interno ribosomal nuclear transcribido 1 (ITS1) de tres linajes de hongos ectomycorrhizal". MycoKeys (13): 21 –33. doi:10.3897/mycokeys.13.8579. hdl:10261/163935.
- ^ Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (diciembre de 2007). "La transcripción RNA de polimerasa III en expansión". Tendencias en la genética. 23 (12): 614 –622. doi:10.1016/j.tig.2007.09.001. Hdl:11381/1706964. PMID 17977614.
- ^ Dieci G, Giuliodori S, Catellani M, Percudani R, Ottonello S (marzo de 2002). "La adaptación del promotor intragénico y el reciclaje de polimerasa III RNA facilitado en la transcripción de SCR1, el gen de ARN 7SL de Saccharomyces cerevisiae". El Diario de Química Biológica. 277 (9): 6903 –6914. doi:10.1074/jbc.M105036200. PMID 11741971.
- ^ Bothwell, AL; Garber, RL; Altman, S (10 de diciembre de 1976). "Secuencia de nicleotida y procesamiento in vitro de una molécula precursora a Escherichia coli 4.5 S RNA". El Diario de Química Biológica. 251 (23): 7709 –16. doi:10.1016/S0021-9258(17)32909-5. PMID 794064.
- ^ Shan SO, Walter P (febrero de 2005). "Proteína co-translacional dirigida por la partícula de reconocimiento de señales". Cartas FEBS. 579 4): 921 –926. Bibcode:2005FEBSL.579..921S. doi:10.1016/j.febslet.2004.11.049. PMID 15680975. S2CID 46046514.
- ^ Zwieb C, Eichler J (marzo de 2002). "Conseguir el objetivo: la partícula de reconocimiento de señales arqueales". Archaea. 1 1): 27 –34. doi:10.1155/2002/729649. PMC 2685543. PMID 15803656.
- ^ Ulbrandt ND, Newitt JA, Bernstein HD (enero de 1997). "La partícula de reconocimiento de señales E. coli es necesaria para la inserción de un subconjunto de proteínas de membrana interna". Celular. 88 2): 187 –196. doi:10.1016/S0092-8674(00)81839-5. PMID 9008159. S2CID 15246619.
- ^ Abell BM, Pool MR, Schlenker O, Sinning I, High S (Julio 2004). "La partícula de reconocimiento de señales media la segmentación post-translacional en eucariotas". El Diario EMBO. 23 (14): 2755–2764. doi:10.1038/sj.emboj.7600281. PMC 514945. PMID 15229647.
- ^ Schuenemann D, Gupta S, Persello-Cartieaux F, Klimyuk VI, Jones JD, Nussaume L, Hoffman NE (agosto de 1998). "Una nueva partícula de reconocimiento de señal apunta a las proteínas que da luz a las membranas tilakoideas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 95 (17): 10312–10316. Bibcode:1998PNAS...9510312S. doi:10.1073/pnas.95.17.10312. PMC 21505. PMID 9707644.
- ^ Zwieb C, van Nues RW, Rosenblad MA, Brown JD, Samuelsson T (enero de 2005). "Una nomenclatura para todos los ARN de las partículas de reconocimiento de señales". ARN. 11 1): 7 –13. doi:10.1261/rna.7203605. PMC 1370686. PMID 15611297.
- ^ Zwieb C (agosto de 1992). "Recognition of a tetranucleotide loop of signal recognition particle RNA by protein SRP19". El Diario de Química Biológica. 267 (22): 15650–15656. doi:10.1016/S0021-9258(19)49585-9. PMID 1379233.
- ^ a b Hainzl T, Huang S, Sauer-Eriksson AE (junio de 2002). "Fuente del complejo SRP19 RNA y implicaciones para el montaje de partículas de reconocimiento de señales". Naturaleza. 417 (6890): 767 –771. Bibcode:2002Natur.417..767H. doi:10.1038/nature00768. PMID 12050674. S2CID 2509475.
- ^ a b Iakhiaeva E, Wower J, Wower IK, Zwieb C (junio de 2008). "El motivo 5e de la partícula de reconocimiento de señales eucariotas RNA contiene una adenosina conservada para la unión de SRP72". ARN. 14 (6): 1143 –1153. doi:10.1261/rna.979508. PMC 2390789. PMID 18441046.
- ^ a b Weichenrieder O, Wild K, Strub K, Cusack S (noviembre de 2000). "Strutura y montaje del dominio Alu de la partícula de reconocimiento de señales mamíferas". Naturaleza. 408 (6809): 167–173. Bibcode:2000Natur.408..167W. doi:10.1038/35041507. PMID 11089964. S2CID 4427070.
- ^ a b Kuglstatter A, Oubridge C, Nagai K (2002). "Cambios estructurales inducidos del ARN 7SL durante la asamblea de partículas humanas de reconocimiento de señales". Nat Struct Biol. 9 (10): 740 –744. doi:10.1038/nsb843. PMID 12244299. S2CID 9543041.
- ^ Walter P, Blobel G (septiembre de 1983). "Disasambly and reconstitution of signal recognition part". Celular. 34 2): 525 –533. doi:10.1016/0092-8674(83)90385-9. PMID 6413076. S2CID 17907778.
- ^ Politz JC, Yarovoi S, Kilroy SM, Gowda K, Zwieb C, Pederson T (enero 2000). "Compuestos de partículas de reconocimiento de señales en el núcleo". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 97 1): 55–60. Bibcode:2000PNAS...97...55P. doi:10.1073/pnas.97.1.55. PMC 26615. PMID 10618370.
- ^ Brooks MA, Ravelli RB, McCarthy AA, Strub K, Cusack S (mayo de 2009). "Structure de SRP14 de la partícula de reconocimiento de señal de señales de Schizosaccharomyces". Acta Crystallographica Section D. 65 (Pt 5): 421 –433. doi:10.1107/S0907444909005484. PMID 19390147.
- ^ Liu L, Ben-Shlomo H, Xu YX, Stern MZ, Goncharov I, Zhang Y, Michaeli S (mayo de 2003). "La partícula de reconocimiento de señal tripanosomatida consiste en dos moléculas de ARN, un homólogo de ARN 7SL y una nueva molécula tRNA". El Diario de Química Biológica. 278 (20): 18271–18280. doi:10.1074/jbc.M209215200. PMID 12606550.
- ^ Hann BC, Stirling CJ, Walter P (abril de 1992). "SEC65 gene product is a subunit of the leeast signal recognition part required for its integrity". Naturaleza. 356 (6369): 532–533. Bibcode:1992Natur.356..532H. doi:10.1038/356532a0. PMID 1313947. S2CID 4287636.
- ^ Römisch K, Webb J, Herz J, Prehn S, Frank R, Vingron M, Dobberstein B (agosto de 1989). "Homología de la proteína 54K de partículas de reconocimiento de señales, proteína de acoplamiento y dos proteínas E. coli con dominios de unión GTP putative" (PDF). Naturaleza. 340 (6233): 478 –482. Código: 1989Natur.340..478R. doi:10.1038/340478a0. PMID 2502717. S2CID 4343347.
- ^ Bernstein HD, Poritz MA, Strub K, Hoben PJ, Brenner S, Walter P (agosto de 1989). "Modelo para el reconocimiento de secuencias de señales de secuencia aminoácida de 54K subunidad de partículas de reconocimiento de señales". Naturaleza. 340 (6233): 482 –486. Código: 1989Natur.340..482B. doi:10.1038/340482a0. PMID 2502718. S2CID 619959.
- ^ Iakhiaeva E, Bhuiyan SH, Yin J, Zwieb C (junio de 2006). "Proteína SRP68 de partículas de reconocimiento de señales humanas: identificación de los dominios vinculantes RNA y SRP72". Protein Science. 15 (6): 1290 –1302. doi:10.1110/ps.051861406. PMC 2242529. PMID 16672232.
- ^ Iakhiaeva E, Yin J, Zwieb C (enero de 2005). "Identificación de un dominio vinculante de ARN en el SRP72 humano". Journal of Molecular Biology. 345 4): 659 –666. doi:10.1016/j.jmb.2004.10.087. PMID 15588816.
Más lectura
- Kuglstatter A, Oubridge C, Nagai K (octubre de 2002). "Cambios estructurales inducidos del ARN 7SL durante la asamblea de partículas humanas de reconocimiento de señales". Naturaleza Biología estructural. 9 (10): 740 –744. doi:10.1038/nsb843. PMID 12244299. S2CID 9543041.
- Wang T, Tian C, Zhang W, Luo K, Sarkis PT, Yu L, Liu B, Yu Y, Yu XF (diciembre de 2007). "7SL RNA media virion packaging of the antiviral cytidine deaminase APOBEC3G". Journal of Virology. 81 (23): 13112–13124. doi:10.1128/JVI.00892-07. PMC 2169093. PMID 17881443.
- Ullu E, Weiner AM (diciembre de 1984). "Génes humanos y pseudogenos para el componente de ARN 7SL de partículas de reconocimiento de señales". El Diario EMBO. 3 (13): 3303 –3310. doi:10.1002/j.1460-2075.1984.tb02294.x. PMC 557853. PMID 6084597.
- Englert M, Felis M, Junker V, Beier H (diciembre de 2004). "Novelar elementos de control intríngeno e intrínseco para la transcripción dependiente de la polimerasa RNA del ARN humano". Biochimie. 86 (12): 867 –874. doi:10.1016/j.biochi.2004.10.012. PMID 15667936.
- Oubridge C, Kuglstatter A, Jovine L, Nagai K (junio de 2002). "Crystal structure of SRP19 in complex with the S domain of SRP RNA and its implication for the assembly of the signal recognition part". Celda molecular. 9 (6): 1251 –1261. doi:10.1016/S1097-2765(02)00530-0. PMID 12086622.
- Wang T, Tian C, Zhang W, Sarkis PT, Yu XF (enero de 2008). "La interacción con ARN 7SL pero no con ARN genómico VIH-1 o cuerpos P es necesaria para el embalaje de virión APOBEC3F". Journal of Molecular Biology. 375 4): 1098 –1112. doi:10.1016/j.jmb.2007.11.017. PMID 18067920.
Enlaces externos
- The SRP Database (SRPDB): Alineación de ARNs de SRP y proteínas asociadas, estructuras secundarias de RNA de SRP y modelos 3-D
- Entrada Rfam para la partícula de reconocimiento de señal tipo Metazoan RNA
- Entrada Rfam para la partícula Bacterial de pequeño reconocimiento de señal RNA
- Entrada Rfam para la partícula de reconocimiento de señales bacterianas RNA
- Entrada Rfam para la partícula de reconocimiento de señal fúngica RNA
- Entrada Rfam para la partícula de reconocimiento de señales de plantas RNA
- Entrada Rfam para la partícula de reconocimiento de señales Protozoan RNA
- Entrada Rfam para la partícula de reconocimiento de señal arqueal RNA
- Dnatube Señal Recognition Particle Movie