Trakehner
Trakehner es una raza de caballo de sangre caliente ligera, desarrollada originalmente en la yeguada estatal de Prusia Oriental en la ciudad de Trakehnen, de... (leer más)
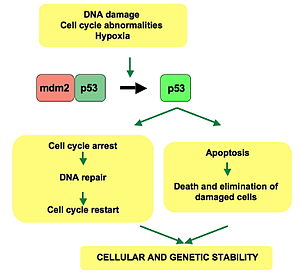
p53, también conocida como proteína tumoral P53, antígeno tumoral celular p53 (nombre UniProt) o proteína relacionada con la transformación 53 (TRP53) es una proteína reguladora que a menudo está mutada en los cánceres humanos. Las proteínas p53 (originalmente se pensaba que eran una sola proteína y, a menudo, se las denominaba así) son cruciales en los vertebrados, donde previenen la formación de cáncer. Como tal, p53 ha sido descrito como "el guardián del genoma" debido a su papel en la conservación de la estabilidad al prevenir la mutación del genoma. Por lo tanto, TP53 se clasifica como un gen supresor de tumores.
El nombre p53 se le dio en 1979 para describir la masa molecular aparente; El análisis SDS-PAGE indica que es una proteína de 53 kilodalton (kDa). Sin embargo, la masa real de la proteína p53 de longitud completa (p53α) basada en la suma de las masas de los residuos de aminoácidos es sólo de 43,7 kDa. Esta diferencia se debe a la gran cantidad de residuos de prolina en la proteína, que ralentizan su migración en SDS-PAGE, haciéndola parecer más pesada de lo que realmente es. Además de la proteína de longitud completa, el gen TP53 humano codifica al menos 15 isoformas de proteínas, cuyo tamaño oscila entre 3,5 y 43,7 kDa. Todas estas proteínas p53 se denominan isoformas p53. El gen TP53 es el gen mutado con mayor frecuencia (>50 %) en el cáncer humano, lo que indica que el gen TP53 desempeña un papel crucial en la prevención de la formación del cáncer. El gen TP53 codifica proteínas que se unen al ADN y regulan la expresión génica para prevenir mutaciones del genoma.
En humanos, el gen TP53 se encuentra en el brazo corto del cromosoma 17 (17p13.1). El gen abarca 20 kb, con un exón 1 no codificante y un primer intrón muy largo de 10 kb. La secuencia de codificación contiene cinco regiones que muestran un alto grado de conservación en los vertebrados, predominantemente en los exones 2, 5, 6, 7 y 8, pero las secuencias encontradas en los invertebrados muestran solo un parecido lejano con el TP53 de los mamíferos. Se han identificado ortólogos TP53 en la mayoría de los mamíferos para los que se dispone de datos completos del genoma.
En los humanos, un polimorfismo común involucra la sustitución de una arginina por una prolina en la posición del codón 72 del exón 4. Muchos estudios han investigado un vínculo genético entre esta variación y la susceptibilidad al cáncer; sin embargo, los resultados han sido controvertidos. Por ejemplo, un metanálisis de 2009 no pudo mostrar un vínculo con el cáncer de cuello uterino. Un estudio de 2011 encontró que la mutación de prolina TP53 tenía un efecto profundo en el riesgo de cáncer de páncreas entre los hombres. Un estudio de mujeres árabes encontró que la homocigosidad de prolina en el codón 72 TP53 está asociada con un menor riesgo de cáncer de mama. Un estudio sugirió que los polimorfismos del codón 72 TP53, MDM2 SNP309 y A2164G pueden asociarse colectivamente con la susceptibilidad al cáncer no orofaríngeo y que MDM2 SNP309 en combinación con el codón 72 TP53 puede acelerar el desarrollo de cáncer no orofaríngeo en mujeres. Un estudio de 2011 encontró que el polimorfismo del codón 72 TP53 estaba asociado con un mayor riesgo de cáncer de pulmón.
Los metaanálisis de 2011 no encontraron asociaciones significativas entre los polimorfismos del codón 72 de TP53 y el riesgo de cáncer colorrectal y de endometrio. Un estudio de 2011 de una cohorte de nacimiento brasileña encontró una asociación entre la arginina no mutante TP53 y las personas sin antecedentes familiares de cáncer. Otro estudio de 2011 encontró que el genotipo homocigoto p53 (Pro/Pro) estaba asociado con un riesgo significativamente mayor de carcinoma de células renales.
p53 tiene siete dominios:
Las mutaciones que desactivan p53 en el cáncer suelen ocurrir en el DBD. La mayoría de estas mutaciones destruyen la capacidad de la proteína para unirse a sus secuencias de ADN objetivo y, por lo tanto, evita la activación transcripcional de estos genes. Como tal, las mutaciones en DBD son mutaciones recesivas de pérdida de función. Las moléculas de p53 con mutaciones en la OD se dimerizan con p53 de tipo salvaje y evitan que activen la transcripción. Por lo tanto, las mutaciones de OD tienen un efecto negativo dominante sobre la función de p53.
La p53 de tipo salvaje es una proteína lábil que comprende regiones plegadas y no estructuradas que funcionan de forma sinérgica.
p53 juega un papel en la regulación o progresión a través del ciclo celular, la apoptosis y la estabilidad genómica a través de varios mecanismos:
Codificación WAF1/CIP1 para p21 y cientos de otros genes posteriores. p21 (WAF1) se une a los complejos G1-S/CDK (CDK4/CDK6, CDK2 y CDK1) (moléculas importantes para la transición G1/S en el ciclo celular) inhibiendo su actividad.
Cuando p21(WAF1) forma complejo con CDK2, la célula no puede continuar a la siguiente etapa de división celular. Una p53 mutante ya no se unirá al ADN de manera efectiva y, como consecuencia, la proteína p21 no estará disponible para actuar como la 'señal de parada'. para la división celular. Los estudios de células madre embrionarias humanas (hESC) comúnmente describen el eje p53-p21 no funcional de la vía del punto de control G1/S con relevancia posterior para la regulación del ciclo celular y la respuesta al daño del ADN (DDR). Es importante destacar que el ARNm de p21 está claramente presente y regulado al alza después de la DDR en hESC, pero la proteína p21 no es detectable. En este tipo de células, p53 activa numerosos microARN (como miR-302a, miR-302b, miR-302c y miR-302d) que inhiben directamente la expresión de p21 en hESC.
La proteína p21 se une directamente a los complejos ciclina-CDK que impulsan el ciclo celular e inhiben su actividad quinasa, lo que provoca la detención del ciclo celular para permitir que se lleve a cabo la reparación. p21 también puede mediar en la detención del crecimiento asociada con la diferenciación y una detención del crecimiento más permanente asociada con la senescencia celular. El gen p21 contiene varios elementos de respuesta p53 que median la unión directa de la proteína p53, lo que da como resultado la activación transcripcional del gen que codifica la proteína p21.
Las vías p53 y RB1 están vinculadas a través de p14ARF, lo que plantea la posibilidad de que las vías se regulen entre sí.
La luz ultravioleta puede estimular la expresión de p53, lo que también provoca daños en el ADN. En este caso, p53 puede iniciar eventos que conducen al bronceado.
Los niveles de p53 juegan un papel importante en el mantenimiento de las células madre durante el desarrollo y el resto de la vida humana.
En las células madre embrionarias humanas (hESC), p53 se mantiene en niveles inactivos bajos. Esto se debe a que la activación de p53 conduce a una rápida diferenciación de hESC. Los estudios han demostrado que la eliminación de p53 retrasa la diferenciación y que agregar p53 provoca una diferenciación espontánea, lo que muestra cómo p53 promueve la diferenciación de hESC y desempeña un papel clave en el ciclo celular como regulador de la diferenciación. Cuando p53 se estabiliza y activa en hESC, aumenta p21 para establecer un G1 más largo. Esto generalmente conduce a la abolición de la entrada en la fase S, lo que detiene el ciclo celular en G1 y conduce a la diferenciación. Sin embargo, el trabajo en células madre embrionarias de ratón ha demostrado recientemente que la expresión de P53 no conduce necesariamente a la diferenciación. p53 también activa miR-34a y miR-145, que luego reprimen los factores de pluripotencia de hESC, lo que instiga aún más la diferenciación.
En las células madre adultas, la regulación de p53 es importante para el mantenimiento de la troncalidad en los nichos de células madre adultas. Las señales mecánicas como la hipoxia afectan los niveles de p53 en estas células de nicho a través de los factores inducibles por hipoxia, HIF-1α y HIF-2α. Mientras que HIF-1α estabiliza p53, HIF-2α la suprime. La supresión de p53 desempeña funciones importantes en el fenotipo de las células madre cancerosas, las células madre pluripotentes inducidas y otras funciones y comportamientos de las células madre, como la formación de blastema. Se ha demostrado que las células con niveles reducidos de p53 se reprograman en células madre con una eficiencia mucho mayor que las células normales. Los artículos sugieren que la falta de detención del ciclo celular y la apoptosis da a más células la oportunidad de ser reprogramadas. También se demostró que los niveles reducidos de p53 son un aspecto crucial de la formación de blastema en las patas de las salamandras. La regulación de p53 es muy importante al actuar como una barrera entre las células madre y un estado de células madre diferenciadas, así como una barrera entre las células madre funcionales y las cancerosas.
Además de los efectos celulares y moleculares mencionados anteriormente, p53 tiene un efecto anticancerígeno a nivel tisular que actúa inhibiendo la angiogénesis. A medida que los tumores crecen, necesitan reclutar nuevos vasos sanguíneos para abastecerlos, y p53 inhibe eso al (i) interferir con los reguladores de la hipoxia tumoral que también afectan la angiogénesis, como HIF1 y HIF2, (ii) inhibir la producción de factores promotores de la angiogénesis, y (iii) aumentar directamente la producción de inhibidores de la angiogénesis, como arresten.
Se ha demostrado que p53 al regular el factor inhibidor de la leucemia facilita la implantación en el ratón y posiblemente en la reproducción humana.
p53 actúa como un sensor de estrés celular. Normalmente se mantiene en niveles bajos al estar constantemente marcada para su degradación por la proteína MDM2 de ubiquitina ligasa E3. p53 se activa en respuesta a innumerables factores estresantes, incluido el daño en el ADN (inducido por UV, IR o agentes químicos como el peróxido de hidrógeno), estrés oxidativo, choque osmótico, agotamiento de ribonucleótidos y expresión oncogénica desregulada. Esta activación está marcada por dos grandes eventos. Primero, la vida media de la proteína p53 aumenta drásticamente, lo que lleva a una rápida acumulación de p53 en las células estresadas. En segundo lugar, un cambio conformacional obliga a p53 a activarse como regulador de la transcripción en estas células. El evento crítico que conduce a la activación de p53 es la fosforilación de su dominio N-terminal. El dominio de activación transcripcional N-terminal contiene una gran cantidad de sitios de fosforilación y puede considerarse como el objetivo principal para las proteínas quinasas que transducen señales de estrés.
Las proteínas quinasas que se sabe que se dirigen a este dominio de activación transcripcional de p53 se pueden dividir aproximadamente en dos grupos. Un primer grupo de proteínas quinasas pertenece a la familia MAPK (JNK1-3, ERK1-2, p38 MAPK), que se sabe que responde a varios tipos de estrés, como daño de membrana, estrés oxidativo, choque osmótico, choque térmico, etc. Un segundo grupo de proteínas quinasas (ATR, ATM, CHK1 y CHK2, DNA-PK, CAK, TP53RK) está implicado en el punto de control de integridad del genoma, una cascada molecular que detecta y responde a varias formas de daño en el ADN causado por estrés genotóxico. Los oncogenes también estimulan la activación de p53, mediada por la proteína p14ARF.
En células no estresadas, los niveles de p53 se mantienen bajos a través de una degradación continua de p53. Una proteína llamada Mdm2 (también llamada HDM2 en humanos), se une a p53, impidiendo su acción y la transporta desde el núcleo hasta el citosol. Mdm2 también actúa como una ligasa de ubiquitina y une covalentemente la ubiquitina a p53 y, por lo tanto, marca a p53 para su degradación por el proteasoma. Sin embargo, la ubiquitilación de p53 es reversible. En la activación de p53, Mdm2 también se activa, configurando un circuito de retroalimentación. Los niveles de p53 pueden mostrar oscilaciones (o pulsos repetidos) en respuesta a ciertos estreses, y estos pulsos pueden ser importantes para determinar si las células sobreviven al estrés o mueren.
MI-63 se une a MDM2, reactivando p53 en situaciones donde la función de p53 se ha inhibido.
Una proteasa específica de ubiquitina, USP7 (o HAUSP), puede escindir la ubiquitina de p53, protegiéndola así de la degradación dependiente del proteasoma a través de la vía de la ubiquitina ligasa. Este es un medio por el cual p53 se estabiliza en respuesta a ataques oncogénicos. También se ha demostrado que USP42 desubiquitina p53 y puede ser necesario para la capacidad de p53 para responder al estrés.
Investigaciones recientes han demostrado que HAUSP se localiza principalmente en el núcleo, aunque una fracción se puede encontrar en el citoplasma y las mitocondrias. La sobreexpresión de HAUSP da como resultado la estabilización de p53. Sin embargo, el agotamiento de HAUSP no da como resultado una disminución en los niveles de p53, sino que aumenta los niveles de p53 debido al hecho de que HAUSP se une y desubiquitina a Mdm2. Se ha demostrado que HAUSP es un mejor compañero de unión a Mdm2 que a p53 en células no estresadas.
Sin embargo, se ha demostrado que USP10 está ubicado en el citoplasma de células no estresadas y desubiquitina p53 citoplasmático, revirtiendo la ubiquitinación de Mdm2. Después del daño en el ADN, USP10 se transloca al núcleo y contribuye a la estabilidad de p53. Además, USP10 no interactúa con Mdm2.
La fosforilación del extremo N-terminal de p53 por parte de las proteínas quinasas mencionadas anteriormente interrumpe la unión de Mdm2. Otras proteínas, como Pin1, luego se reclutan para p53 e inducen un cambio conformacional en p53, lo que evita aún más la unión de Mdm2. La fosforilación también permite la unión de coactivadores transcripcionales, como p300 y PCAF, que luego acetilan el extremo carboxi-terminal de p53, exponiendo el dominio de unión al ADN de p53, lo que le permite activar o reprimir genes específicos. Las enzimas desacetilasas, como Sirt1 y Sirt7, pueden desacetilar p53, lo que lleva a una inhibición de la apoptosis. Algunos oncogenes también pueden estimular la transcripción de proteínas que se unen a MDM2 e inhiben su actividad.
Si el gen TP53 está dañado, la supresión del tumor se ve gravemente comprometida. Las personas que heredan solo una copia funcional del gen TP53 probablemente desarrollarán tumores en la edad adulta temprana, un trastorno conocido como síndrome de Li-Fraumeni.
El gen TP53 también puede ser modificado por mutágenos (químicos, radiación o virus), lo que aumenta la probabilidad de división celular descontrolada. Más del 50 por ciento de los tumores humanos contienen una mutación o eliminación del gen TP53. La pérdida de p53 crea una inestabilidad genómica que, en la mayoría de los casos, da como resultado un fenotipo de aneuploidía.
Aumentar la cantidad de p53 puede parecer una solución para el tratamiento de tumores o la prevención de su propagación. Esto, sin embargo, no es un método utilizable de tratamiento, ya que puede causar envejecimiento prematuro. La restauración de la función endógena normal de p53 es prometedora. La investigación ha demostrado que esta restauración puede conducir a la regresión de ciertas células cancerosas sin dañar otras células en el proceso. Las formas en que se produce la regresión tumoral dependen principalmente del tipo de tumor. Por ejemplo, la restauración de la función p53 endógena en los linfomas puede inducir la apoptosis, mientras que el crecimiento celular puede reducirse a niveles normales. Por lo tanto, la reactivación farmacológica de p53 se presenta como una opción viable de tratamiento del cáncer. La primera terapia génica comercial, Gendicine, fue aprobada en China en 2003 para el tratamiento del carcinoma de células escamosas de cabeza y cuello. Entrega una copia funcional del gen p53 utilizando un adenovirus diseñado.
Ciertos patógenos también pueden afectar a la proteína p53 que expresa el gen TP53. Un ejemplo de ello, el virus del papiloma humano (VPH), codifica una proteína, E6, que se une a la proteína p53 y la inactiva. Este mecanismo, en sinergia con la inactivación del regulador del ciclo celular pRb por la proteína E7 del VPH, permite la división celular repetida que se manifiesta clínicamente como verrugas. Ciertos tipos de VPH, en particular los tipos 16 y 18, también pueden conducir a la progresión de una verruga benigna a una displasia cervical de bajo o alto grado, que son formas reversibles de lesiones precancerosas. La infección persistente del cuello uterino a lo largo de los años puede causar cambios irreversibles que conducen a un carcinoma in situ y, finalmente, a un cáncer de cuello uterino invasivo. Esto se debe a los efectos de los genes del VPH, en particular los que codifican E6 y E7, que son las dos oncoproteínas virales que se retienen y expresan preferentemente en los cánceres de cuello uterino mediante la integración del ADN viral en el genoma del huésped.
La proteína p53 se produce y degrada continuamente en las células de las personas sanas, lo que da como resultado una oscilación amortiguada (consulte un modelo estocástico de este proceso en). La degradación de la proteína p53 está asociada con la unión de MDM2. En un bucle de retroalimentación negativa, la proteína p53 induce al propio MDM2. Las proteínas mutantes p53 a menudo no logran inducir MDM2, lo que hace que p53 se acumule en niveles muy altos. Además, la propia proteína p53 mutante puede inhibir los niveles normales de proteína p53. En algunos casos, se ha demostrado que las mutaciones sin sentido únicas en p53 alteran la estabilidad y la función de p53.
Se ha demostrado que la supresión de p53 en células de cáncer de mama humano conduce a una mayor expresión del gen del receptor de quimiocinas CXCR5 y activa la migración celular en respuesta a la quimiocina CXCL13.
Un estudio encontró que las proteínas p53 y Myc eran clave para la supervivencia de las células de la leucemia mieloide crónica (LMC). Dirigirse a las proteínas p53 y Myc con fármacos dio resultados positivos en ratones con LMC.
Esta imagen muestra diferentes patrones de expresión p53 en cánceres endometriales en inmunohistoquímica cromogénica, de los cuales todos excepto el tipo salvaje son variamente denominados anormales/aberrant/mutación-tipo y son fuertemente predictivos de una mutación TP53 subyacente:
|
La mayoría de las mutaciones de p53 se detectan mediante secuenciación de ADN. Sin embargo, se sabe que las mutaciones únicas de sentido erróneo pueden tener un amplio espectro desde efectos funcionales bastante leves hasta muy graves.
El amplio espectro de fenotipos de cáncer debido a mutaciones en el gen TP53 también está respaldado por el hecho de que diferentes isoformas de proteínas p53 tienen diferentes mecanismos celulares para la prevención contra el cáncer. Las mutaciones en TP53 pueden dar lugar a diferentes isoformas, impidiendo su funcionalidad general en diferentes mecanismos celulares y, por lo tanto, extendiendo el fenotipo del cáncer de leve a grave. Estudios recientes muestran que las isoformas de p53 se expresan diferencialmente en diferentes tejidos humanos, y las mutaciones de pérdida o ganancia de función dentro de las isoformas pueden causar cáncer específico de tejido o proporcionar potencial de células madre cancerosas en diferentes tejidos. La mutación TP53 también afecta el metabolismo energético y aumenta la glucólisis en las células de cáncer de mama.
La dinámica de las proteínas p53, junto con su antagonista Mdm2, indican que los niveles de p53, en unidades de concentración, oscilan en función del tiempo. Este "amortiguado" la oscilación se documenta clínicamente y se modela matemáticamente. Los modelos matemáticos también indican que la concentración de p53 oscila mucho más rápido una vez que los teratógenos, como las roturas de doble cadena (DSB) o la radiación UV, se introducen en el sistema. Esto respalda y modela la comprensión actual de la dinámica de p53, donde el daño del ADN induce la activación de p53 (consulte la regulación de p53 para obtener más información). Los modelos actuales también pueden ser útiles para modelar las mutaciones en las isoformas de p53 y sus efectos en la oscilación de p53, promoviendo así el descubrimiento de fármacos farmacológicos específicos de tejido de novo.
p53 fue identificado en 1979 por Lionel Crawford, David P. Lane, Arnold Levine y Lloyd Old, que trabajaban en el Imperial Cancer Research Fund (Reino Unido), la Universidad de Princeton/UMDNJ (Instituto del Cáncer de Nueva Jersey) y el Memorial Sloan-Kettering Centro del Cáncer, respectivamente. Se había planteado la hipótesis de que existía antes como objetivo del virus SV40, una cepa que inducía el desarrollo de tumores. El gen TP53 del ratón fue clonado por primera vez por Peter Chumakov de la Academia de Ciencias de la URSS en 1982, y de forma independiente en 1983 por Moshe Oren en colaboración con David Givol (Instituto de Ciencias Weizmann). El gen TP53 humano se clonó en 1984 y el clon completo en 1985.
Inicialmente se supuso que era un oncogén debido al uso de ADNc mutado después de la purificación del ARNm de células tumorales. Su papel como gen supresor de tumores fue revelado en 1989 por Bert Vogelstein en la Escuela de Medicina Johns Hopkins y Arnold Levine en la Universidad de Princeton. p53 pasó a ser identificado como un factor de transcripción por Guillermina Lozano, que trabajaba en el MD Anderson Cancer Center.
Warren Maltzman, del Instituto Waksman de la Universidad de Rutgers, demostró por primera vez que TP53 respondía al daño del ADN en forma de radiación ultravioleta. En una serie de publicaciones de 1991 a 1992, Michael Kastan, de la Universidad Johns Hopkins, informó que TP53 era una parte fundamental de una vía de transducción de señales que ayudaba a las células a responder al daño del ADN.
En 1993, p53 fue votada como molécula del año por la revista Science.
Al igual que el 95 % de los genes humanos, TP53 codifica más de una proteína. Se descubrieron varias isoformas en 2005, y hasta ahora se han identificado 12 isoformas de p53 humanas (p53α, p53β, p53γ, ∆40p53α, ∆40p53β, ∆40p53γ, ∆133p53α, ∆1333p53β, ∆1333p53γ, ∆160p53x, ∆161610p5x,). Además, las isoformas de p53 se expresan de manera dependiente del tejido y p53α nunca se expresa sola.
Las proteínas de la isoforma p53 de longitud completa se pueden subdividir en diferentes dominios proteicos. A partir del extremo N, primero están los dominios de transactivación amino-terminal (TAD 1, TAD 2), que son necesarios para inducir un subconjunto de genes diana de p53. Este dominio va seguido del dominio rico en prolina (PXXP), por lo que se repite el motivo PXXP (P es una prolina y X puede ser cualquier aminoácido). Se requiere, entre otros, para la apoptosis mediada por p53. Algunas isoformas carecen del dominio rico en prolina, como Δ133p53β,γ y Δ160p53α,β,γ; por lo tanto, algunas isoformas de p53 no median la apoptosis, lo que enfatiza las funciones diversificadoras del gen TP53. Luego está el dominio de unión al ADN (DBD), que permite a las proteínas secuenciar la unión específica. El dominio carboxilo terminal completa la proteína. Incluye la señal de localización nuclear (NLS), la señal de exportación nuclear (NES) y el dominio de oligomerización (OD). El NLS y NES son responsables de la regulación subcelular de p53. A través de la OD, p53 puede formar un tetrámero y luego unirse al ADN. Entre las isoformas, pueden faltar algunos dominios, pero todos ellos comparten la mayor parte del dominio de unión al ADN altamente conservado.
Las isoformas se forman por diferentes mecanismos. Las isoformas beta y gamma se generan por corte y empalme múltiple del intrón 9, lo que conduce a un extremo C diferente. Además, el uso de un promotor interno en el intrón 4 provoca las isoformas ∆133 y ∆160, que carecen del dominio TAD y de una parte del DBD. Además, la iniciación alternativa de la traducción en el codón 40 o 160 lleva las isoformas ∆40p53 y ∆160p53.
Debido a la naturaleza isofórmica de las proteínas p53, ha habido varias fuentes de evidencia que muestran que las mutaciones dentro del gen TP53 que dan lugar a isoformas mutadas son agentes causantes de varios fenotipos de cáncer, desde leves a graves., debido a una mutación única en el gen TP53 (consulte la sección Análisis experimental de las mutaciones de p53 para obtener más detalles).
Se ha demostrado que p53 interactúa con:
Trakehner es una raza de caballo de sangre caliente ligera, desarrollada originalmente en la yeguada estatal de Prusia Oriental en la ciudad de Trakehnen, de... (leer más)
enfermedad de Charcot-Marie-Tooth es una neuropatía sensitiva y motora hereditaria del sistema nervioso periférico caracterizada por la pérdida progresiva... (leer más)
El pez de las cavernas del norte o pez ciego del norte se encuentra en cuevas en Kentucky y el sur de Indiana.. Está catalogado como una especie amenazada en... (leer más)