Motivo de activación basado en tirosina del inmunorreceptor
Un motivo de activación basado en tirosina de un inmunorreceptor (ITAM) es una secuencia conservada de cuatro aminoácidos que se repite dos veces en las colas citoplasmáticas de los receptores no catalíticos fosforilados en tirosina, proteínas de la superficie celular que se encuentran principalmente en las células inmunitarias. Su función principal es ser un componente integral para la iniciación de una variedad de vías de señalización y, posteriormente, la activación de las células inmunitarias, aunque se han descrito diferentes funciones, por ejemplo, la maduración de los osteoclastos.
Estructura
El motivo contiene una tirosina separada de una leucina o isoleucina por otros dos aminoácidos, lo que da la firma YxxL/I. Dos de estas firmas suelen estar separadas por entre 6 y 8 aminoácidos en la cola citoplasmática de la molécula (YxxL/Ix(6-8)YxxL/I). Sin embargo, en varias fuentes, esta secuencia de consenso difiere, principalmente en el número de aminoácidos entre firmas individuales. Aparte de los ITAM que tienen la estructura descrita anteriormente, también existe una variedad de proteínas que contienen motivos similares a los ITAM, que tienen una estructura y función muy similares (por ejemplo, en la proteína Dectin-1).
Función
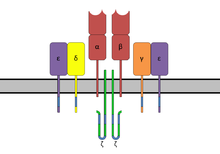
Los ITAM son importantes para la transducción de señales, principalmente en las células inmunes. Se encuentran en las colas citoplasmáticas de los receptores no catalíticos fosforilados en tirosina, como las cadenas CD3 y ζ del complejo receptor de células T, las cadenas CD79-alfa y -beta del complejo receptor de células B y ciertos receptores Fc. Los residuos de tirosina dentro de estos motivos se fosforilan por las quinasas de la familia Src después de la interacción de las moléculas del receptor con sus ligandos. Los ITAM fosforilados sirven como sitios de acoplamiento para otras proteínas que contienen un dominio SH2, generalmente dos dominios en tándem, induciendo una cascada de señalización mediada por las quinasas de la familia Syk (que son las proteínas primarias que se unen a los ITAM fosforilados), es decir, Syk o ZAP-70, lo que resulta principalmente en la activación de una célula determinada. Paradójicamente, en algunos casos, los ITAM y los motivos similares a ITAM no tienen un efecto activador, sino más bien inhibidor. Los mecanismos exactos de este fenómeno aún no se han dilucidado.
Otros receptores no catalíticos fosforilados en tirosina tienen un motivo inhibidor conservado (ITIM) que, cuando se fosforila, produce la inhibición de la vía de señalización a través del reclutamiento de fosfatasas, a saber, SHP-1, SHP-2 y SHIP1. Esto sirve no solo para la inhibición y regulación de las vías de señalización relacionadas con la señalización basada en ITAM, sino también para la terminación de la señalización.
Variaciones genéticas
En las bases de datos de variación genética humana se han catalogado mutaciones genéticas humanas raras que, según se informa, pueden dar lugar a la creación o eliminación de ITIM e ITAM.
Ejemplos
Los ejemplos que se muestran a continuación enumeran tanto proteínas que contienen el ITAM como proteínas que utilizan la señalización basada en ITAM con la ayuda de proteínas asociadas que contienen el motivo.
CD3γ, CD3δ, CD3ε, TYROBP (DAP12), FcαRI, FcγRI, FcγRII, FcγRIII, Dectin-1, CLEC-1, CD28, CD72
Referencias
- ^ a b c Abbas AK, Lichtman AH (2009), Inmunología básica: Funciones y Trastornos del Sistema Inmunitario (3 ed.), Philadelphia, PA: Saunders, ISBN 978-1-4160-4688-2
- ^ Humphrey, Mary Beth; Daws, Michael R.; Spusta, Steve C.; Niemi, Eréne C.; Torchia, James A.; Lanier, Lewis L.; Seaman, William E.; Nakamura, Mary C. (Febrero de 2006). "TREM2, un receptor asociado con DAP12, regula la diferenciación y función osteoclasto" (PDF). Journal of Bone and Mineral Research. 21 2): 237 –245. doi:10.1359/JBMR.051016. ISSN 0884-0431. PMID 16418779. S2CID 34957715.
- ^ Paloneva, Juha; Mandelin, Jami; Kiialainen, Anna; Böhling, Tom; Prudlo, Johannes; Hakola, Panu; Haltia, Matti; Konttinen, Yrjö T.; Peltonen, Leena (2003-08-18). "DAP12/TREM2 Resultados de Deficiencia en Diferenciación de Osteoclasto y Características Osteoporóticas con discapacidad". Journal of Experimental Medicine. 198 4): 669 –675. doi:10.1084/jem.20030027. ISSN 0022-1007. PMC 2194176. PMID 12925681.
- ^ Rogers, Neil C.; Slack, Emma C.; Edwards, Alexander D.; Nolte, Martijn A.; Schulz, Oliver; Schweighoffer, Edina; Williams, David L.; Gordon, Siamon; Tybulewicz, Victor L.; Brown, Gordon D.; Reis e Sousa, Caetano (abril de 2005). "La inducción de citoquinas dependientes de Syk por Dectin-1 revela un nuevo camino de reconocimiento de patrones para lectinas tipo C". Inmunity. 22 4): 507 –517. doi:10.1016/j.immuni.2005.03.004. ISSN 1074-7613. PMID 15845454.
- ^ Underhill, David M.; Rossnagle, Eddie; Lowell, Clifford A.; Simmons, Randi M. (2005-10-01). "Dectin-1 activa Syk tyrosine kinase en un subconjunto dinámico de macrófagos para la producción reactiva de oxígeno". Sangre. 106 (7): 2543 –2550. doi:10.1182/blood-2005-03-1239. ISSN 0006-4971. PMC 1895265. PMID 15956283.
- ^ Suzuki-Inoue, Katsue; Fuller, Gemma L. J.; García, Angel; Eble, Johannes A.; Pöhlmann, Stefan; Inoue, Osamu; Gartner, T. Kent; Hughan, Sascha C.; Pearce, Andrew C.; Laing, Gavin D.; Theakston, R. David G. (2006-01-15). "Un nuevo mecanismo dependiente de Syk de activación de plaquetas por el receptor de lectina tipo C CLEC-2". Sangre. 107 2): 542 –549. doi:10.1182/blood-2005-05-1994. ISSN 0006-4971. PMID 16174766. S2CID 168505.
- ^ a b Dushek O, Goyette J, van der Merwe PA (noviembre de 2012). "Receptores no calíticos de tirosina-fosforilados". Reseñas inmunológicas. 250 1): 258–76. doi:10.1111/imr.12008. PMID 23046135. S2CID 1549902.
- ^ Pasquier, Benoit; Launay, Pierre; Kanamaru, Yutaka; Moura, Ivan C.; Pfirsch, Séverine; Ruffié, Claude; Hénin, Dominique; Benhamou, Marc; Pretolani, Marina; Blank, Ulrich; Monteiro, Renato C. (enero 2005). "Identificación de Fcalpha RI como receptor inhibidor que controla la inflamación: doble papel de FcRgamma ITAM". Inmunity. 22 1): 31–42. doi:10.1016/j.immuni.2004.11.017. ISSN 1074-7613. PMID 15664157.
- ^ O’Neill, Shannon K.; Getahun, Andrew; Gauld, Stephen B.; Merrell, Kevin T.; Tamir, Idan; Smith, Mia J.; Dal Porto, Joseph M.; Li, Quan-Zhen; Cambier, John C. (2011-11-23). "Monofosforilación de motivos CD79a y b ITAM inicia una cascada de señalización inhibitoria mediada por fosfatasa SHIP-1 requerida para la anergia celular B". Inmunity. 35 5): 746 –756. doi:10.1016/j.immuni.2011.10.011. ISSN 1074-7613. PMC 3232011. PMID 22078222.
- ^ Pfirsch-Maisonnas, Séverine; Aloulou, Meryem; Xu, Ting; Claver, Julien; Kanamaru, Yutaka; Tiwari, Meetu; Launay, Pierre; Monteiro, Renato C.; Blank, Ulrich (2011-04-19). "Inhibidor ITAM Trampas de señalización Receptores de activación con el SHP-1 de fosfatasa para formar Clusters Polarizados "Inhibisome". Ciencia Signaling. 4 (169): ra24. doi:10.1126/scisignal.2001309. ISSN 1945-0877. PMID 21505186. S2CID 206670699.
- ^ Long, Eric O. (agosto de 2008). "La señalización negativa por los receptores inhibidores: el paradigma de la célula NK". Reseñas inmunológicas. 224: 70–84. doi:10.1111/j.1600-065X.2008.00660.x. ISSN 1600-065X. PMC 2587243. PMID 18759921.
- ^ Kane, Barry A.; Bryant, Katherine J.; McNeil, H. Patrick; Tedla, Nicodemo T. (2014). "Terminación de la activación inmunitaria: un componente esencial de respuestas sanas de inmunes". Journal of Innate Immunity. 6 (6): 727 –738. doi:10.1159/000363449. ISSN 1662-811X. PMC 6741560. PMID 25033984.
- ^ Ligeti, E.; Csépányi-Kömi, R.; Hunyady, L. (abril de 2012). "Mecanismos fisiológicos de terminación de señal en sistemas biológicos". Acta Physiologica. 204 4): 469 –478. doi:10.1111/j.1748-1716.2012.02414.x. ISSN 1748-1716. PMID 22260256. S2CID 13455093.
- ^ Auton A, Brooks LD, Durbin RM, Garrison EP, Kang HM, Korbel JO, et al. (octubre de 2015). "Una referencia global para la variación genética humana". Naturaleza. 526 (7571): 68–74. Bibcode:2015Natur.526...68T. doi:10.1038/nature15393. PMC 4750478. PMID 26432245.
- ^ Sherry ST, Ward MH, Kholodov M, Baker J, Phan L, Smigielski EM, Sirotkin K (enero de 2001). "dbSNP: la base de datos NCBI de variación genética". Nucleic Acids Research. 29 1): 308–11. doi:10.1093/nar/29.1.308. PMC 29783. PMID 11125122.
- ^ Cummings BB, Karczewski KJ, Kosmicki JA, Seaby EG, Watts NA, Singer-Berk M, et al. (May 2020). "La anotación del conocimiento de la expresión transcript mejora la interpretación de la variante rara". Naturaleza. 581 (7809): 452 –458. Bibcode:2020Natur.581..452C. doi:10.1038/s41586-020-2329-2. PMC 7334198. PMID 32461655.
- ^ Ulaganathan VK (mayo 2020). "TraPS-VarI: Identificar variantes genéticas alterando los motivos de señalización basados en fosfotyrosina". Scientific Reports. 10 (1): 8453. Código:2020 NatSR..10.8453U. doi:10.1038/s41598-020-65146-2. PMC 7242328. PMID 32439998.