Marcador de tamaño de peso molecular

Un marcador de tamaño de peso molecular, también conocido como escalera de proteínas, escalera de ADN o escalera de ARN, es un conjunto de estándares que se utilizan para identificar el tamaño aproximado de una molécula que se procesa en un gel durante la electroforesis, utilizando el principio de que el peso molecular es inversamente proporcional a la velocidad de migración a través de una matriz de gel. Por lo tanto, cuando se utilizan en la electroforesis en gel, los marcadores proporcionan efectivamente una escala logarítmica con la que se puede estimar el tamaño de los demás fragmentos (siempre que se conozcan los tamaños de los fragmentos del marcador).
Se encuentran disponibles en el mercado marcadores de proteínas, ADN y ARN con tamaños y concentraciones de fragmentos predeterminados. Estos se pueden analizar en geles de agarosa o poliacrilamida. Los marcadores se cargan en carriles adyacentes a los carriles de muestra antes del comienzo del análisis.
Marcadores de ADN

Desarrollo
Aunque se ha mantenido el concepto de marcadores de peso molecular, las técnicas de desarrollo han variado a lo largo de los años. Las nuevas invenciones de marcadores de peso molecular se distribuyen en kits específicos para el tipo de marcador.
Uno de los primeros problemas en el desarrollo de marcadores fue lograr una alta resolución en toda la longitud del marcador. Según las condiciones de funcionamiento de la electroforesis en gel, los fragmentos pueden haberse comprimido, lo que altera la claridad. Para abordar este problema, en 1990 se desarrolló un kit para el análisis Southern Blot, que proporcionó el primer marcador que combinaba ADN objetivo y ADN sonda. Esta técnica aprovechaba el espaciado logarítmico y podía utilizarse para identificar bandas objetivo con una longitud de 20.000 nucleótidos.
Diseño
Existen dos métodos comunes para construir un marcador de tamaño de peso molecular de ADN. Uno de estos métodos emplea la técnica de ligadura parcial. La ligadura de ADN es el proceso por el cual se conectan entre sí fragmentos de ADN lineales mediante enlaces covalentes; más específicamente, estos enlaces son enlaces fosfodiéster. Aquí, se liga parcialmente un fragmento de ADN dúplex de 100 pb. La consecuencia de esto es que se formarán dímeros de 200 pb, trímeros de 300 pb, tetrámeros de 400 pb, pentámeros de 500 pb, etc. Además, quedará una porción del dsADN de 100 pb. Como resultado, se crea en el gel una "escalera" de ADN compuesta por fragmentos de ADN de masa molecular conocida.
El segundo método emplea enzimas de restricción y una secuencia de ADN reconocida. El ADN es digerido por una enzima de restricción particular, lo que da como resultado fragmentos de ADN de masas moleculares variables. Una de las ventajas de este método es que se puede crear más marcador con facilidad simplemente digiriendo más ADN conocido. Por otro lado, el tamaño de los fragmentos de ADN se basa en los sitios donde corta la enzima de restricción. Esto hace que sea más difícil controlar el tamaño de los fragmentos en el marcador.
Más recientemente, los laboratorios están empleando otro método para construir marcadores de tamaño de peso molecular de ADN. Esta estrategia implica el uso de la reacción en cadena de la polimerasa (PCR). Esto se logra de una o dos maneras: 1) se amplifica un objetivo de ADN al mismo tiempo mediante conjuntos de cebadores, o 2) se amplifican diferentes objetivos de ADN de forma independiente mediante cebadores particulares.
Efectos de las condiciones de gel
Al igual que con las muestras experimentales, las condiciones del gel pueden afectar el tamaño del marcador de peso molecular que corre junto a ellas. Factores como el tampón, la carga/voltaje y la concentración del gel pueden afectar la movilidad o la apariencia del marcador/escalera/estándar. Estos elementos deben tenerse en cuenta al seleccionar un marcador y al analizar los resultados finales en un gel.

- Buffers
- Los amortiguadores actúan a 1) establecer pH, y 2) proporcionar iones para apoyar la conductividad. En la electroforesis del ADN, el TAE (Tris-acetate-EDTA) y TBE (Tris-borate-EDTA) son los buffers habituales de elección. TBE buffer es preferido para pequeñas piezas de ADN, mientras que TAE es más adecuado para fragmentos mayores de 1500 pares base. En términos de capacidad de amortiguación, TAE es menor en comparación con TBE; esto generalmente resulta en una movilidad más lenta del ADN. TBE también es capaz de una mejor resolución.
- Cabe señalar que el agua no puede servir de sustituto a uno de estos buffers, ya que el ADN no migrará a lo largo del gel. Además, el uso de agua en lugar de amortiguador dará lugar a la fusión de gel.
- Carga/Voltaje
- En términos de tensión, el rango recomendado es entre 4 y 10 V/cm (es decir, voltios/cm). Los geles de agarose generalmente se ejecutan a un voltaje de 5 V/cm. La unidad de distancia, cm, se refiere a la distancia entre los electrodos (es decir, el ánodo y la cátodo) y no la longitud del gel mismo.
- Los voltajes demasiado por debajo o por encima de esta gama afectarán la movilidad y la resolución de las bandas. Baja tensión disminuirá la movilidad y hará que las bandas se amplíen. Por otro lado, los voltajes altos disminuirán la resolución de las bandas. Esto se debe en gran medida al hecho de que los voltajes demasiado altos pueden causar que el gel se recaliente, e incluso se derrita.
- Concentración
- La concentración de agarosa debe tenerse en cuenta al seleccionar un marcador. El porcentaje de gel afecta la migración del ADN. En general, cuanto mayor sea la concentración de gel, más lento será la tasa a la que el ADN pasará por el gel. Esto se suma al papel que juega el peso molecular en la migración de un marcador de ADN o muestra, es decir, que cuanto más alto sea el peso molecular, más lento migrará el ADN.
- La concentración de gel también afecta la capacidad de visualizar las bandas que se ejecutan en el gel. Las bandas más pequeñas se resuelven mejor en un mayor porcentaje de gel, mientras que el aumento de las bandas de peso molecular se visualiza más fácilmente en un menor porcentaje de gel.
Marcadores de proteínas
Desarrollo
Anteriormente, los marcadores proteicos se habían desarrollado utilizando una variedad de proteínas completas. El desarrollo de un kit que incluía un marcador de tamaño de peso molecular basado en fragmentos de proteínas comenzó en 1993. Este marcador proteico, compuesto por 49 secuencias de aminoácidos diferentes, incluía proteínas multidominio y permitía el análisis de proteínas escindidas en diferentes sitios.
Las mejoras técnicas actuales en los marcadores proteicos implican el uso del desarrollo automático. El primer marcador proteico de peso regular desarrollado automáticamente se inventó en 2012.
Diseño
Al igual que los marcadores de ADN, estos marcadores suelen estar compuestos de proteínas purificadas cuyas masas moleculares ya se conocen. La lista que aparece a continuación describe algunas de las proteínas, así como la masa molecular, que se utilizan habitualmente al construir un marcador proteico.

| Proteína | Masa molecular (kDa) |
| Beta-galactosidase | 120 |
| Phosphorylase B | 94 |
| Bovine Serum Albumin (BSA) | 67 |
| Ovalbumin | 43 |
| Turkey Albumin | 40 |
| Carbonic Anhydrase | 30 |
| Soybean Trypsin Inhibitor | 20.1 |
| a-Lactalbumin | 14.4 |
| Lysozyme | 14 |
Elegir el marcador de proteínas correcto
Los marcadores de tamaño de peso molecular se pueden dividir en dos categorías: marcadores de peso molecular y marcadores de escalera molecular. Los marcadores están teñidos o no teñidos y, según las circunstancias, uno puede ser más apropiado que otro. Los marcadores de tamaño de peso molecular también se pueden alterar bioquímicamente. La conjugación con biotina es la más común. Los marcadores de tamaño de peso molecular se utilizan con mayor frecuencia en la electroforesis en gel de poliacrilamida con SDS y en la transferencia Western. Con todos los diferentes tipos y usos de los marcadores de tamaño de peso molecular, es importante elegir el estándar de proteína adecuado. Además del uso más común, como una forma de calcular el peso molecular de las muestras, otros usos incluyen permitir evidencia visual de la migración de proteínas y la eficiencia de transferencia y, a veces, incluso se utilizan como control positivo.
- Marcador MW vs Escaleras Protein
- Un marcador de peso molecular es un tipo de estándar de proteínas. Pueden prestarse o no detenerse antes de cargar; dependiendo del tipo de experimento uno puede ser más ventajoso. En cualquier caso, normalmente se ejecutan en el carril exterior de un gel, mientras que la muestra se carga en los carriles medios. Los marcadores moleculares son diferentes de las escaleras de proteína en que se componen de una mezcla de proteínas nativas cuyas especificaciones están bien categorizadas pero no corresponden a números enteros. Generalmente estos son mucho más baratos, pero el análisis sólo permite un valor aproximado de las proteínas separadas de la electroforesis.
- Una escalera de proteína es otro tipo de estándar de proteínas. Casi siempre están manchados. Las escaleras de proteína difieren de los marcadores moleculares en que están compuestas de una mezcla de proteínas altamente purificadas cuyas especificaciones son conocidas y corresponden a números enteros. Generalmente las escaleras de proteína se componen de 10-12 proteínas. Al final del experimento, después de que se produzca la migración de tamaño, una sola banda representará el tamaño de cada proteína contenida en la escalera. Los marcadores son uniformemente espaciados, y el análisis de tamaño usando estos marcadores permite un valor preciso de la proteína de interés. En algunos casos, como método de confirmación molecular, los marcadores MW se ejecutan con escaleras de proteína para la verificación.
- Marcadores prescritos y no contaminados
- Los marcadores de proteína pueden ser no asegurados o prestados, pero ambos tienen sus beneficios y desventajas. La visualización simple de la separación y transferencia de proteínas se hace posible mediante el uso de marcadores prestained. Se utilizan comúnmente tanto en la electroforesis de gel de poliacrilamida SDS como en la hinchazón occidental. En SDS-PAGE permite el monitoreo de la migración de proteínas, ya que las bandas de proteínas se separarán y se pueden ver durante una carrera electroforética. En las manchas occidentales, las normas de proteínas manchadas permiten la transferencia de proteínas de visualización a la membrana. Sin embargo, las determinaciones de tamaño no son tan exactas con estos marcadores (ver sección Recombinant y Natural Marker para mayor explicación).
- Mientras que los marcadores no manchados permiten determinar el tamaño más exacto, no pueden ser vistos mientras el gel está funcionando. Como tal, el gel debe ser manchado para visualizar las bandas.
- Marcadores Recombinantes y Naturales
- Además de marcadores manchados y no manchados, los marcadores de proteínas se pueden pensar en términos de recombinante y natural. Los marcadores recombinantes consisten en proteínas recombinantes que han sido muy purificadas. Estos marcadores están diseñados de tal manera que destacar características particulares. Ejemplos de estas características incluyen etiquetas de afinidad y pesos moleculares que se colocan uniformemente en relación entre sí.
- Los marcadores naturales, como el nombre implica, son una mezcla de proteínas que suceden naturalmente. Los marcadores naturales mantenidos funcionan bien para la visualización de la separación de gel. Sin embargo, estos marcadores tienden a unirse a la mancha de manera covalente en cantidades variables y en varias posiciones. En consecuencia, las bandas resultantes pueden ser más amplias. Esto es especialmente cierto al hacer comparaciones con los marcadores recombinantes prestained. Debido a este efecto, es probable que las determinaciones de peso molecular sean menos precisas con los marcadores naturales prestained.
- Biochemically Altered
- Las normas de proteína también pueden alterarse químicamente. Una alteración común es a través de la biotina de uso. Biotina tiene una afinidad muy alta para la estreptavidina, y por lo tanto, la unión forma un complejo muy fuerte. Para la visualización, una etiqueta de color se adjunta a la estreptavidina.
Efectos de las condiciones de gel
Al igual que con la electroforesis de ADN, al seleccionar un marcador proteico se deben tener en cuenta condiciones como los tampones, la carga/voltaje y la concentración.
- Buffers
- Los amortiguadores pueden afectar la movilidad del marcador y las muestras. El pH del búfer varía con el sistema utilizado y, en consecuencia, cada sistema de búfer tendrá un efecto diferente de la carga de una proteína o proteínas. Además, en el caso de SDS-PAGE, la afinidad vinculante para SDS puede verse afectada por el sistema de amortiguación. Incluso al utilizar el mismo porcentaje y tipo de gel, las mismas proteínas migrarán a diferentes tasas dependiendo del búfer usado.
- Carga/Voltaje
- El voltaje juega un papel en la movilidad de las proteínas en un gel. Las proteínas migrarán más rápido a mayores voltajes. En consecuencia, el tiempo de funcionamiento del gel será más corto. Por el contrario, los voltajes más altos pueden resultar en una mayor difusión de banda. Además, si el voltaje es demasiado alto, la temperatura en la cámara de electroforesis puede convertirse en tal que el gel comienza a fundirse.
- El voltaje al que debe correr un gel depende del tipo del gel. Para algunos geles, el voltaje permanece constante a lo largo de la carrera, mientras que, con otros geles, se permite que el voltaje inicial permanezca constante durante un tiempo determinado antes de que se incremente. Este segundo voltaje se utiliza para un marco de tiempo específico, después de lo cual, también se puede aumentar.
- Concentración
- En términos de porcentaje, los geles utilizados para la electroforesis de proteínas se pueden desglosar en geles de un solo porcentaje y geles gradientes. Los geles de un solo porcentaje también se denominan geles lineales. Para los geles lineales, el porcentaje seleccionado generalmente cae entre el 7,5% y el 20%. Los rangos porcentuales comunes para los geles gradientes son 4-15% y 10-20%. Cada tipo de gel tiene sus propias ventajas. Por ejemplo, los geles lineales son preferidos cuando varias proteínas tienen pesos moleculares similares; una mejor separación entre estas proteínas será mostrada por un gel lineal. Por otro lado, los geles gradientes son una mejor opción cuando las muestras de interés contienen proteínas de pesos moleculares muy diferentes o que cubren una gran variedad de pesos moleculares.
Marcadores de ARN
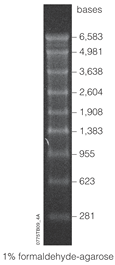
Desarrollo
Las escaleras de ARN compuestas de marcadores de tamaño de peso molecular de ARN se desarrollaron inicialmente utilizando el método del círculo sintético para producir marcadores de diferentes tamaños. Esta técnica fue mejorada por el inventor Eric T. Kool para utilizar vectores de ADN circulares como método para producir marcadores de tamaño de peso molecular de ARN. Conocido como el método del círculo rodante, las mejoras de esta técnica se derivan de su eficiencia en la síntesis de oligonucleótidos de ARN. A partir de la plantilla de ADN circular, se puede producir ARN monocatenario con una longitud que varía de 4 a 1500 pb sin la necesidad de cebadores y reciclando el trifosfato de nucleótidos. El ADN también se puede sintetizar a partir de la plantilla circular, lo que aumenta la versatilidad de esta técnica. En comparación con la transcripción de escurrimiento, el método del círculo sintético produce oligonucleótidos de ARN sin el escurrimiento. En comparación con la PCR, el método del círculo sintético produce oligonucleótidos de ARN sin la necesidad de polimerasa ni de un ciclador térmico. Este método también es rentable en su capacidad para sintetizar grandes cantidades de producto con una tasa de error menor que los sintetizadores automáticos.
Diseño
Los marcadores de ARN consisten en transcripciones de ARN de distintas longitudes que se van incrementando. Por ejemplo, el marcador Lonza de 0,5 a 9 kbp tiene bandas que marcan 0,5, 1, 1,5, 2, 2,5, 3, 4, 5, 6 y 9 pares de kilobases. Los marcadores se disuelven en un tampón de almacenamiento, como EDTA, y pueden tener una vida útil de hasta 2 años cuando se almacenan a -80 °C. Para utilizar el marcador, como para el análisis de transferencia Northern, primero se descongela y luego se tiñe para que sea detectable en una electroforesis en gel. Uno de los colorantes más comunes utilizados para los marcadores es el bromuro de etidio.
El rango de un marcador en particular se refiere a la variedad de bandas que puede mapear. Un rango "alto" se refiere a fragmentos relativamente grandes (medidos en kb), mientras que un rango "bajo" se refiere a marcadores que distinguen entre fragmentos pequeños (medidos en pb). Algunos marcadores pueden incluso describirse como de "rango ultrabajo", pero el marcador de microARN es aún más preciso. Un marcador de microARN se puede utilizar para medir fragmentos de ARN dentro de una docena de nucleótidos, como el marcador de microARN de 17-25 nt.
Uso
En pesos moleculares equivalentes, el ARN migrará más rápido que el ADN. Sin embargo, tanto el ARN como el ADN tienen una pendiente lineal negativa entre su distancia de migración y su peso molecular logarítmico. Es decir, las muestras de menor peso pueden migrar una distancia mayor. Esta relación es un factor a tener en cuenta al elegir marcadores de ARN o ADN como estándar.
Al analizar marcadores de ARN y muestras de ARN en un gel, es importante evitar la contaminación por nucleasas, ya que el ARN es muy sensible a la degradación por ribonucleasas (RNasas) a través de la catálisis. Por lo tanto, se deben tener en cuenta todos los materiales que se utilizarán en el procedimiento. Todo el material de vidrio que entre en contacto con el ARN debe tratarse previamente con dietilpirocarbonato (DEPC) y los materiales plásticos deben ser desechables.
Marcadores de tamaño molecular y SDS-PAGE

Uno de los usos más comunes de los marcadores de tamaño de peso molecular es la electroforesis en gel. El propósito de la electroforesis en gel es separar proteínas según sus propiedades físicas o químicas, que incluyen la carga, el tamaño molecular y el pH.< Cuando se separa en función del tamaño, el método ideal es la electroforesis en gel de poliacrilamida o SDS-PAGE y los marcadores de tamaño de peso molecular son los estándares adecuados para utilizar.
Los geles pueden variar de tamaño. La cantidad de muestras que se van a analizar determinará el tamaño de gel adecuado. Todos los geles se dividen en carriles que recorren el gel en paralelo. Cada carril contendrá una muestra específica. Normalmente, los estándares de tamaño de peso molecular se colocan en un carril exterior. Si un gel tiene una cantidad particularmente alta de carriles, se pueden colocar múltiples escaleras a lo largo del gel para lograr una mayor claridad.
Las proteínas y los estándares se pipetean en el gel en los carriles apropiados. El dodecil sulfato de sodio (SDS) interactúa con las proteínas, las desnaturaliza y les otorga una carga negativa. Dado que todas las proteínas tienen la misma relación carga-masa, la movilidad de las proteínas a través del gel se basará únicamente en el peso molecular. Una vez que se activa el campo eléctrico, se iniciará la migración de proteínas. Una vez completado, se puede utilizar un mecanismo de detección como el Western blotting, que revelará la presencia de bandas. Cada banda representa una proteína específica. La distancia recorrida se basa únicamente en el peso molecular; por lo tanto, el peso molecular de cada proteína se puede determinar comparando la distancia de una proteína desconocida con el estándar de peso molecular conocido.
Diferentes usos de marcadores de tamaño molecular
Existen muchos tipos de marcadores de tamaño de peso molecular y cada uno posee características únicas que permiten su uso en diversas técnicas biológicas. La selección de un marcador de tamaño de peso molecular depende del tipo de marcador (ADN, ARN o proteína) y del rango de longitud que ofrece (p. ej., 1 kb). Antes de seleccionar un marcador de tamaño de peso molecular, es importante familiarizarse con estas características y propiedades. En un caso particular, un tipo puede ser más apropiado que otro. Aunque los marcadores específicos pueden variar entre los protocolos para una técnica determinada, en esta sección se describirán los marcadores generales y sus funciones.
Allozymes
El primer tipo de marcador molecular desarrollado y analizado en electroforesis en gel fueron las alozimas. Estos marcadores se utilizan para detectar variaciones en las proteínas. La palabra "alozima" (también conocida como "aloenzima") proviene de "variantes alélicas de las enzimas". Cuando se analizan en un gel, las proteínas se separan por tamaño y carga. Aunque las alozimas pueden parecer anticuadas en comparación con otros marcadores disponibles, todavía se utilizan hoy en día, principalmente debido a su bajo costo. Una desventaja importante es que, dado que solo hay una cantidad limitada disponible, la especificidad es un problema.
Marcadores basados en ADN (1960s)
Aunque las alozimas pueden detectar variaciones en el ADN, lo hacen mediante un método indirecto y no muy preciso. Los marcadores basados en el ADN se desarrollaron en la década de 1960. Estos marcadores son mucho más eficaces para distinguir entre variantes del ADN. Hoy en día, son los marcadores más utilizados. Los marcadores basados en el ADN funcionan mediante el estudio de los nucleótidos, lo que puede cumplir una variedad de funciones, como detectar diferencias en los nucleótidos o incluso cuantificar el número de mutaciones.
- RFLP
- El polimorfismo de longitud de fragmento de restricción es una técnica utilizada para detectar variaciones en el ADN homologoso. Las endonucleas de restricción específicas se utilizan para digerir el ADN. El marcador molecular RFLP es específico para un solo fragmento. Junto con los marcadores de RFLP aleicos, un marcador de tamaño molecular-peso, en este caso un marcador de ADN, también se incluye en un gel de agarose electorfosed. El marcador de ADN permite calcular el tamaño de los fragmentos de restricción.
- Minisatélites
- Similar a RFLP, esta técnica también utiliza endonucleas de restricción para digerir el ADN genómico. Los minisatélites son secuencias cortas de repeticiones tándem, aproximadamente 10-60 pares base. Los minisatélites se pueden utilizar en la huella de ADN y como reguladores de control de genes.
Marcadores basados en PCR (1980s)
El éxito de los marcadores basados en ADN condujo al desarrollo de la PCR. La PCR (reacción en cadena de la polimerasa) es una técnica de amplificación de ADN que se puede aplicar a varios tipos de fragmentos. Antes de este desarrollo, para amplificar el ADN, era necesario clonarlo o aislarlo. Poco después del descubrimiento de la PCR surgió la idea de utilizar marcadores basados en PCR para la electroforesis en gel. Este tipo de marcadores se basan en cebadores de PCR y se clasifican como polimorfismos de secuencia de ADN.
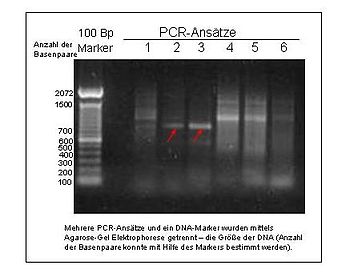
- Microsatélites
- También conocido como SSR (repeticiones de secuencia simple) o STR (repeticiones cortas de tándem), microsatélites difieren de minisatélites en que son más cortos, generalmente 2-6 pares base. Esta propiedad de microsatélites permite un aislamiento fácil. Los microsatélites se utilizan más comúnmente en la genética poblacional. Los microsatélites tienen una tasa de mutación elevada y compleja, que es su principal desventaja.
- AFLP
- El polimorfismo de longitud de fragmento amplificado es una técnica de identificación de ADN basada en PCR. El ADN se digiere primero con endonucleas. Los fragmentos de restricción se ligan juntos. Luego se genera un marcador molecular cuando se seleccionan fragmentos específicos para la amplificación. Los marcadores AFLP se ejecutan junto con un marcador de ADN en un gel. Un marcador común de ADN AFLP es de 30-330bp de largo. Los fragmentos de este marcador se encuentran a intervalos de 10bp para aumentar la precisión.
- RAPD
- El ADN polimorfo amplificado aleatorio es una técnica que se lleva a cabo similar a la AFLP. La diferencia es que los marcadores moleculares se generan al azar. El marcador de tamaño molecular más común para esta técnica es la escalera de ADN de 1kb.
ADN secuencia polimorfismo
Aunque técnicamente hablando, el polimorfismo de secuencias de ADN se ha estado produciendo desde el uso de RFLP en la década de 1960, el análisis ha cambiado significativamente con el paso de los años. El polimorfismo de secuencias de ADN utiliza técnicas más antiguas como RFLP, pero a mayor escala. La secuenciación es mucho más rápida y eficiente. El análisis está automatizado, ya que utiliza una técnica conocida como secuenciación shotgun. Este método de alto rendimiento se utiliza comúnmente en genética de poblaciones.
- SNPs
- Los SNP (polimorfismo de nucleótido único), se utilizan para detectar variaciones en los nucleótidos individuales. La técnica es muy similar a la de RFLP. Los SNP se utilizan con frecuencia para estudios genéticos de población. Después de la amplificación a través de PCR, estos pequeños fragmentos se pueden visualizar usando electroforesis de gel, y de nuevo los marcadores de ADN juegan un papel en la determinación de la longitud del fragmento.
Análisis de polisacáridos por electroforesis de gel de carbohidratos
Los marcadores de carbohidratos se emplean en una técnica conocida como análisis de polisacáridos por electroforesis en gel de carbohidratos (PACE), que es una técnica de separación medible. Permite el análisis de productos de hidrólisis enzimática. Se ha utilizado en aplicaciones como la caracterización de enzimas involucradas en la degradación de hemicelulosa, la determinación de la estructura de polisacáridos de hemicelulosa y el análisis de la escisión enzimática de productos de celulosa.
La PACE depende de la derivatización, que es la conversión de un compuesto químico en un derivado. En este caso, los compuestos de interés son los monosacáridos, oligosacáridos y polisacáridos. Se marcan en sus extremos reductores con una etiqueta fluorescente (es decir, un fluoróforo). Esta derivatización con un fluoróforo permite tanto la separación en un gel en las circunstancias deseadas como la obtención de imágenes de fluorescencia del gel. En este caso, se utiliza un gel de poliacrilamida.
Al igual que con la electroforesis de ADN, ARN y proteínas, los marcadores se analizan junto con las muestras de interés en la electroforesis en gel de carbohidratos. Los marcadores consisten en oligosacáridos de peso molecular conocido. Al igual que las muestras de interés, el marcador también se derivatiza con un fluoróforo (generalmente con ácido 8-aminonaftaleno-1,3,6-trisulfónico (ANTS) o 2-aminoacridona).
Referencias
- ^ Carlson, David P. "Size markers for electrophoretic analysis of DNA". US Patent #5316908A. Google Patentes. Retrieved 30 de octubre 2013.
- ^ Carlson, David P. "Size markers for electrophoretic analysis of DNA". EP Patent #0466404B1. Google Patentes. Retrieved 30 de octubre 2013.
- ^ a b c d e f h i j k l m n o p Blaber, Mike. "Lecture 20: Gel Electrophoresis". BCH5425 Biología molecular y biotecnología.
- ^ Bowen, R (20 de octubre de 1999). "DNA Ligation". Biotecnología e Ingeniería Genética. Retrieved 12 de noviembre 2013.
- ^ a b c Lan, Vo Thi Thuong; Loan, Pham Thi Thanh; Duong, Pham Anh Thuy; Thanh, Le Thi; Ha, Ngo Thi; Thuan, Ta Bich (2012). "Procedimiento de seguimiento para la producción de laboratorio de la escalera de ADN". Journal of Nucleic Acids. 2012: 254630. doi:10.1155/2012/254630. PMC 3306978. PMID 22496965.
- ^ a b c d e f g Bowen, R. (2000). "Agarose Gel Electroforesis de ADN". Hypertexts for Biomedical Sciences – Colorado State University.
- ^ "Tris Borate EDTA and Tris-Acetate-EDTA Buffer (TAE & TBE, pH 8.3)" (PDF). Aniara.
- ^ a b c "Agarose Gel Electrophoresis Tips " Tricks". Life Technologies.
- ^ Hartley, James. "Escalera marcadora de tamaño proteína". US Patent #5449758A. Google Patentes. Retrieved 30 de octubre 2013.
- ^ Cheng, Tian Lu. "El kit y método de marcador de peso molecular de proteína autodesarrollada y con peso regular para preparar el mismo". Patente de los Estados Unidos #20130217133A1. Google Patentes. Retrieved 30 de octubre 2013.
- ^ "Prestained Protein Molecular Weight Marker". ThermoScientific. Retrieved 12 de noviembre 2013.
- ^ a b c d e f Ingelman, Margareta (2004). "Separación y análisis de proteínas". KE7001 Laboratorios de bioquímica. Retrieved 12 de noviembre 2013.
- ^ "Protein Molecular Weight Markers". Ayuda Biotech. 2011. Retrieved 12 de noviembre 2013.
- ^ a b c d e f g h "Protein Molecular Weight Markers Guía de comparación y selección". Retrieved 16 de noviembre 2013.
- ^ a b "Biotinilated Molecular Weight Marker". Retrieved 16 de noviembre 2013.
- ^ a b "Molecular Weight Markers". Retrieved 16 de noviembre 2013.
- ^ a b "Pierce Prestained Protein Molecular Weight Marker". Retrieved 16 de noviembre 2013.
- ^ a b c d e f h i j k l m n "Electroforesis Una Guía para la Electroforesis y Detección de Gel Polyacrylamide" (PDF). Bio-Rad.
- ^ "Protein Molecular Weight Markers Guía de comparación y selección". ThermoScientific. Retrieved 12 de noviembre 2013.
- ^ a b c d e f g h i "Protein Handbook 2013" (PDF). Life Technologies.
- ^ a b Kool, Eric T. "Vídeos de ADN escalables para la síntesis de ARN y ADN". US Patent #6096880A. Google Patentes. Retrieved 27 de noviembre 2013.
- ^ Lonza. "Marcadores ARN 0,5-9 kbp" (PDF). Documento #18123-0807-06. Lonza Rockland Inc. Retrieved 27 de noviembre 2013.
- ^ New England Biolabs. "microRNA Marker". New England Bioloabs. Retrieved 27 de noviembre 2013.
- ^ Wicks, Richard J. (1986). "RNA determinación de peso molecular por electroforesis de gel agarosa usando formaldehído como denaturante: Comparación de rna y dna marcadores de peso moleculares". International Journal of Biochemistry. 18 (3): 277–278. doi:10.1016/0020-711x(86)90118-7. PMID 2937672.
- ^ a b "Catalog #: R0004". Marcador RNA Alto Fácil. Abnova. Retrieved 14 de diciembre 2013.
- ^ "RNA Electroforesis: Introducción". ARN Electroforesis. Thermo Fisher Scientific Inc. Retrieved 14 de diciembre 2013.
- ^ "Molecular Weight Determination of Proteins" (PDF). Retrieved 14 de diciembre 2013.
- ^ a b c d e f g h Schlotterer, Christian. "La evolución de los marcadores moleculares" (PDF). Retrieved 26 de noviembre 2013.
- ^ "Mejoramiento de Populación". Retrieved 30 de octubre 2013.
- ^ Higgins, L. (abril de 2012). "Escalera de ADN para electroforesis Gel". Lewis " Clark College. Retrieved 15 de noviembre 2013.
- ^ a b Mueller, Ulrich (1999). "FLP genotipado y huella dactilar" (PDF). Tendencias en la Ecología " Evolución. 14 (10): 389–394. doi:10.1016/s0169-5347(99)01659-6. PMID 10481200. Retrieved 30 de octubre 2013.
- ^ Invitrogen Corporation (2003). "30-330 bp AFLP® DNA Ladder" (PDF). Manual. Life Technologies Corporation. Retrieved 15 de noviembre 2013.
- ^ Gianniny, Christine; et al. (mayo de 2004). "análisis de ADN de mtDNA de flores de tomate libres de artefactos nucleares de ADN" (PDF). BioTechniques. 36 (5): 772–776. doi:10.2144/04365BM04. PMID 15152595. Retrieved 15 de noviembre 2013.
- ^ Roberts, M. A.; Crawford, D. L. (1o de junio de 2000). "Uso de ADN polimorfo amplificado aleatoriamente como medio de desarrollar sondas de ADN Genus- y Strain-Specific Streptomyces". Microbiología aplicada y ambiental. 66 (6): 2555–2564. doi:10.1128/AEM.66.6.2555-2564.2000. PMC 110581. PMID 10831438.
- ^ McClean, Phillip. "Las clases de marcadores moleculares". Retrieved 30 de octubre 2013.
- ^ a b c d e f g h Kosik, Ondrej; Bromley, Jennifer R.; Busse-Wicher, Marta; Zhang, Zhinong; Dupree, Paul (2012). Estudios de Cleavage enzimático de celulosa con análisis de polisacáridos por electroforesis de gel de carbohidrato (PACE). Métodos en Enzimología. Vol. 510. pp. 51–67. doi:10.1016/B978-0-12-415931-0.00004-5. ISBN 9780124159310. ISSN 0076-6879. PMID 22608721.
- ^ Cammack R, Attwood TK, Campbell PN, Parish HJ, Smith A, Stirling JL, Vella F (2006). "Fluoroforo". Oxford Dictionary of Biochemistry and Molecular Biology (Segunda edición). Oxford University Press.