Mancha del sur
Southern blot es un método utilizado en biología molecular para la detección y cuantificación de una secuencia de ADN específica en muestras de ADN. La transferencia Southern combina la transferencia de fragmentos de ADN separados por electroforesis a una membrana de filtro en un proceso llamado transferencia y la posterior detección de fragmentos por hibridación de sonda.
El método lleva el nombre del biólogo británico Edwin Southern, quien lo publicó por primera vez en 1975. Otros métodos de transferencia (es decir, transferencia occidental, transferencia norte, transferencia oriental, transferencia suroeste) que emplean principios similares, pero usan ARN o proteína, más tarde han sido nombrados en referencia al nombre de Edwin Southern. Como la etiqueta es epónima, Southern está en mayúscula, como es convencional con los nombres propios. Los nombres de otros métodos de transferencia pueden seguir esta convención, por analogía.
Historia
Southern inventó el Southern blot después de combinar tres innovaciones, la primera son las endonucleasas de restricción que fue desarrollada en la Universidad Johns Hopkins por Tom Kelly y Hamilton Smith, esas endonucleasas de restricción se usan para cortar el ADN en una secuencia específica. Esta técnica fue introducida en Southern por Kenneth y Noreen Murray. La segunda innovación es la electroforesis en gel que se usa para separar fragmentos de ADN, que también fue desarrollada en la Universidad Johns Hopkins por Daniel Nathans, Danna y Kathleen en 1971. La tercera innovación son los métodos de transferencia que fue desarrollado por Frederick Sanger, cuando él moléculas de ARN transferidas a papel DEAE. Southern blot se inventó en 1973 pero no se publicó hasta 1975. Aunque se publicó más tarde, la técnica se difundió cuando Southern presentó la técnica de Southern blot a un científico del Laboratorio Cold Spring Harbor llamado Michael Mathews al dibujar esta técnica en un papel.
Método
- Digestión de ADN: Las endonucleas de restricción se utilizan para cortar las cadenas de ADN de alto peso molecular en fragmentos más pequeños. Esto se hace añadiendo la cantidad deseada de ADN que se puede cambiar según la sonda utilizada y la complejidad del ADN, con la enzima de restricción, el amortiguador de enzimas y el agua purificada, incubar la reacción a 37° C durante la noche.
- Electroforesis Gel: Los fragmentos de ADN son entonces electroforescentes en un gel agarose para separarlos por tamaño. Si algunos de los fragmentos de ADN son mayores de 15 kb, entonces antes de hincharse, el gel puede ser tratado con un ácido, como diluir HCl. Esto depurina los fragmentos de ADN, rompiendo el ADN en piezas más pequeñas, permitiendo así una transferencia más eficiente del gel a la membrana.
- Denaturación: Si se utilizan métodos de transferencia de alcalina, el gel de ADN se coloca en una solución alcalina (normalmente que contiene hidróxido de sodio) para desnaturalizar el ADN doble. La desnaturalización en un entorno alcalino puede mejorar la unión de los residuos de timina cargados negativamente de ADN a un grupo de membrana amino cargado positivamente, separandolo en cadenas de ADN individuales para la hibridación posterior a la sonda (ver abajo), y destruye cualquier ARN residual que aún esté presente en el ADN. Sin embargo, la elección del alcalino sobre los métodos de transferencia neutral es a menudo empírica y puede resultar en resultados equivalentes.
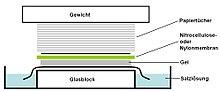
- Blotting: Una hoja de nitrocelulosa (o, alternativamente, nylon) membrana se coloca sobre (o debajo, dependiendo de la dirección de la transferencia) el gel. La presión se aplica uniformemente al gel (ya sea mediante succión, o colocando una pila de toallas de papel y un peso encima de la membrana y el gel), para garantizar un buen contacto e incluso entre el gel y la membrana. Si se transfiere por succión, se utiliza el búfer SSC 20X para asegurar un sello y prevenir el secado del gel. La transferencia de amortiguación por acción capilar de una región de alto potencial de agua a una región de bajo potencial de agua (generalmente filtrar papel y tejidos de papel) se utiliza para mover el ADN del gel a la membrana; interacciones de intercambio de iones unen el ADN a la membrana debido a la carga negativa del ADN y la carga positiva de la membrana.
- Inmovilización: La membrana se hornea luego en un horno vacío o regular a 80 °C durante 2 horas (condiciones estándar; nitrocelulosa o membrana de nylon) o expuesta a la radiación ultravioleta (máquina de nylon) para adjuntar permanentemente el ADN transferido a la membrana.
- Híbridación: La membrana se expone luego a una sonda de hibridación: un solo fragmento de ADN con una secuencia específica cuya presencia en el ADN objetivo debe determinarse. El ADN de la sonda es etiquetado para que pueda ser detectado, generalmente incorporando radioactividad o etiquetando la molécula con un tinte fluorescente o cromogénico. En algunos casos, la sonda de hibridación puede ser hecha de ARN, en lugar de ADN. Para asegurar la especificidad de la unión de la sonda a la muestra de ADN, los métodos más comunes de hibridación utilizan el salmón o el arenque de ADN del esperma para bloquear la superficie de la membrana y el ADN objetivo, formamida deionizada y detergentes como SDS para reducir la unión no específica de la sonda.
- Detección: Después de la hibridación, el exceso de sonda se lava de la membrana (normalmente utilizando el amortiguador SSC), y el patrón de hibridación se visualiza en la película de rayos X por autordiografía en el caso de una sonda radiactiva o fluorescente, o mediante el desarrollo del color en la membrana si se utiliza un método de detección cromogénico.
Interpretación de resultados
La hibridación de la sonda con un fragmento de ADN específico en la membrana del filtro indica que este fragmento contiene una secuencia de ADN que es complementaria a la sonda. El paso de transferencia del ADN del gel de electroforesis a una membrana permite la fácil unión de la sonda de hibridación marcada al ADN fraccionado por tamaño. También permite la fijación de los híbridos diana-sonda, necesarios para el análisis por autorradiografía u otros métodos de detección. Se pueden usar transferencias Southern realizadas con ADN genómico digerido con enzimas de restricción para determinar el número de secuencias (p. ej., copias de genes) en un genoma. Una sonda que se hibrida solo con un único segmento de ADN que no ha sido cortado por la enzima de restricción producirá una sola banda en una transferencia de Southern, mientras que es probable que se observen múltiples bandas cuando la sonda se hibrida con varias secuencias muy similares (p. ej., aquellas que puede ser el resultado de la duplicación de secuencias). Puede usarse la modificación de las condiciones de hibridación (por ejemplo, aumentando la temperatura de hibridación o disminuyendo la concentración de sal) para aumentar la especificidad y disminuir la hibridación de la sonda a secuencias que son menos del 100% similares.
Aplicaciones
- La transferencia de hinchazón del sur se puede utilizar para la clonación basada en la homología sobre la base de secuencia de aminoácidos del producto de proteínas del gen objetivo. Los oligonucleótidos están diseñados para que sean complementarios a la secuencia de destino. Los oligonucleótidos son sintetizados químicamente, radioetiquetados y utilizados para analizar una biblioteca de ADN u otras colecciones de fragmentos de ADN clonados. Las secuencias que hibridizan con la sonda de hibridación se analizan, por ejemplo, para obtener la secuencia de longitud completa del gen objetivo.
- La reorganización cromosómica normal o gen se puede estudiar utilizando esta técnica.
- Se puede utilizar para encontrar secuencias similares en otras especies o en el genoma disminuyendo la especificidad de la hibridación.
- En una mezcla que tiene diferentes tamaños de ADN digerido, se utiliza para identificar el fragmento de restricción de un tamaño específico.
- Es útil identificar cambios que ocurren en genes incluyendo inserciones, reorganizaciones, eliminaciones y mutaciones de puntos que afectan los sitios de restricción.
- Además se utiliza para identificar una región específica que utiliza muchas enzimas de restricción diferentes en una asignación de restricciones. También se utiliza para determinar qué sitio de reconocimiento ha sido alterado debido a un polimorfismo nucleótido único que cambia una enzima de restricción específica.
- La hinchazón del sur también se puede utilizar para identificar sitios metilados en determinados genes. Particularmente útiles son los núcleos de restricción MspI y HpaII, ambos que reconocen y se distancian dentro de la misma secuencia. Sin embargo, HpaII requiere que una C dentro de ese sitio se metilice, mientras que MspI sólo el ADN no metilado en ese sitio. Por lo tanto, cualquier sitio metilado dentro de una secuencia analizada con una sonda particular será arrasado por la primera, pero no la última, enzima.
- Se puede utilizar en identificación personal a través de la huella dactilar, y en el diagnóstico de enfermedades.
Limitaciones de transferencia Southern
La transferencia Southern es una técnica costosa, compleja y que requiere mucho tiempo y consta de varios pasos. Además, esta técnica no se puede utilizar para identificar mutaciones a nivel de pares de bases. Además requiere una gran cantidad de ADN de mayor calidad.
Contenido relacionado
Congestión
Arteritis de células gigantes
Dosis