Gire (bioquímica)
Un giro es un elemento de la estructura secundaria de las proteínas en el que la cadena polipeptídica invierte su dirección general.
Definición
Según una definición, un giro es un motivo estructural en el que los átomos Cα de dos residuos separados por unos pocos enlaces peptídicos (normalmente de 1 a 5) están próximos (menos de 7 Å [0,70 nm]). La proximidad de los átomos Cα terminales suele estar relacionada con la formación de un enlace de hidrógeno entre las cadenas principales de los residuos correspondientes. Este enlace de hidrógeno es la base de la definición original, quizás más conocida, de giro. En muchos casos, pero no en todos, las definiciones de enlace de hidrógeno y de distancia Cα son equivalentes.
Tipos de giros
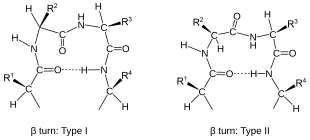
Los giros se clasifican según la separación entre los dos residuos finales:
- En un α-voz los residuos finales están separados por 4 enlaces de péptidos (i → i ± 4).
- En un giro β (la forma más común), por tres bonos (i → i ± 3).
- En un γ-turn, por dos. bonos (i → i ± 2).
- En un δ-turn, por uno bonosi → i ± 1), que es esterilmente improbable.
- En un π-turn, por 5 bonos (i → i ± 5).
| Tipo | φi + 1 | ↑i + 1 | φi + 2 | ↑i + 2 |
|---|---|---|---|---|
| I | ,60° | 30 a 30° | −90° | 0° |
| II | ,60° | 120° | 80° | 0° |
| VIII | ,60° | 30 a 30° | 120 - 120 | 120° |
| I. | 60° | 30° | 90° | 0° |
| II′ | 60° | 120 - 120 | −80° | 0° |
| VIa1 | ,60° | 120° | −90° | 0°* |
| VIa2 | 120 - 120 | 120° | ,60° | 0°* |
| VIb | −135° | 135° | −75° | 160° * |
| IV | de todas las categorías mencionadas | |||
Los giros se clasifican por sus ángulos diedros de la cadena principal (véase el diagrama de Ramachandran). Un giro se puede convertir en su giro inverso (en el que los átomos de la cadena principal tienen quiralidad opuesta) cambiando el signo de sus ángulos diedros. (El giro inverso no es un enantiómero verdadero ya que se mantiene la quiralidad del átomo Cα). Por lo tanto, el giro γ tiene dos formas, una forma clásica con ángulos diedros (φ, ψ) de aproximadamente (75°, −65°) y una forma inversa con ángulos diedros (−75°, 65°). Existen al menos ocho formas del giro beta, que varían en función de si está involucrado un isómero cis de un enlace peptídico y en los ángulos diedros de los dos residuos centrales. Los giros β clásicos e inversos se distinguen con un signo primo, por ejemplo, los giros beta de tipo I y de tipo I′. Si se toma como criterio para los giros un enlace de hidrógeno i → i + 3, las cuatro categorías de Venkatachalam (I, II, II′, I′) son suficientes para describir todos los posibles giros beta. Los cuatro se dan con frecuencia en las proteínas, pero el I es el más común, seguido de II, I′ y II′ en ese orden.
Loops
Un bucle ω es un término general para un bucle más largo, extendido o irregular sin enlaces de hidrógeno internos fijos.
Múltiples giros
En muchos casos, uno o más residuos están involucrados en dos giros parcialmente superpuestos. Por ejemplo, en una secuencia de 5 residuos, tanto los residuos 1 a 4 como los residuos 2 a 5 forman un giro; en tal caso, se habla de un (i, i + 1) doble giro. Los giros múltiples (hasta séptuples) ocurren comúnmente en las proteínas. Las cintas de curvatura beta son un tipo diferente de giro múltiple.
Pepinillos
Una horquilla es un caso especial de giro, en el que la dirección de la cadena principal de la proteína se invierte y los elementos de la estructura secundaria que la flanquean interactúan. Por ejemplo, una horquilla beta conecta dos cadenas β antiparalelas unidas por enlaces de hidrógeno (un nombre bastante confuso, ya que una horquilla β puede contener muchos tipos de giros: α, β, γ, etc.).
Las horquillas beta pueden clasificarse según el número de residuos que forman la espira, es decir, que no forman parte de las hebras β que las flanquean. Si este número es X o Y (según dos definiciones diferentes de láminas β), la horquilla β se define como X:Y.
Los giros beta en los extremos de las horquillas beta tienen una distribución de tipos diferente a los demás; el tipo I′ es el más común, seguido de los tipos II′, I y II.
Enlaces flexibles
A veces se encuentran giros dentro de los enlaces flexibles o bucles que conectan dominios proteicos. Las secuencias de enlaces varían en longitud y suelen ser ricas en aminoácidos polares sin carga. Los enlaces flexibles permiten que los dominios de conexión se tuerzan y roten libremente para reclutar a sus socios de unión a través de la dinámica de los dominios proteicos. También permiten que sus socios de unión induzcan cambios conformacionales a mayor escala mediante alosterio de largo alcance.
Papel en el plegamiento de proteínas
Se han propuesto dos hipótesis sobre el papel de los giros en el plegamiento de proteínas. En una de ellas, los giros desempeñan un papel fundamental en el plegamiento al reunir y permitir o habilitar interacciones entre elementos regulares de la estructura secundaria. Esta hipótesis está respaldada por estudios de mutagénesis que indican un papel fundamental para residuos particulares en los giros de algunas proteínas. Además, los isómeros no nativos de los enlaces peptídicos X−Pro en los giros pueden bloquear por completo el plegamiento conformacional de algunas proteínas. En la hipótesis opuesta, los giros desempeñan un papel pasivo en el plegamiento. Esta hipótesis está respaldada por la escasa conservación de aminoácidos observada en la mayoría de los giros. Los isómeros no nativos de muchos enlaces peptídicos X−Pro en los giros también tienen poco o ningún efecto en el plegamiento.
Métodos de predicción de vuelta de beta
A lo largo de los años se han desarrollado muchos métodos de predicción de giros beta. Recientemente, el grupo del Dr. Raghava desarrolló el método BetaTPred3, que predice un giro beta completo en lugar de residuos individuales que caen en un giro beta. El método también logra una buena precisión y es el primer método que predice los 9 tipos de giros beta. Además de la predicción, este método también se puede utilizar para encontrar el número mínimo de mutaciones necesarias para iniciar o romper un giro beta en una proteína en una ubicación deseada.
Véase también
- Estructura secundaria
- beta turn
Referencias
- ^ Rose, GD; Gierasch, LM; Smith, JA (1985). "Turnas en péptidos y proteínas". Avances en Química Proteína. 37: 1–109. doi:10.1016/s0065-3233(08)60063-7. ISBN 978-0-12-034237-2. PMID 2865874.
- ^ Toniolo, C (1980). "Conformaciones de péptidos intramolecularmente de hidrógeno". CRC Critical Reviews in Biochemistry. 9 (1): 1–44. doi:10.3109/10409238009105471. PMID 6254725.
- ^ Venkatachalam, CM (octubre de 1968). " Criterios estereoquímicos para polipéptidos y proteínas. V. Conformación de un sistema de tres unidades de péptidos vinculadas". Biopolímeros. 6 (10): 1425–36. doi:10.1002/bip.1968.360061006. Hdl:2027.42/37819. PMID 5685102. S2CID 587353535.
- ^ a b Richardson, JS (1981). "La anatomía y taxonomía de la estructura de proteínas". Avances en Química Proteína. 34: 167–339. doi:10.1016/s0065-3233(08)60520-3. ISBN 978-0-12-034234-1. PMID 7020376.
- ^ a b Hutchinson, EG; Thornton, JM (diciembre de 1994). "Un conjunto revisado de potenciales para la formación beta-turn en proteínas". Protein Science. 3 (12): 2207–16. doi:10.1002/pro.5560031206. PMC 2142776. PMID 7756980.
- ^ Venkatachalam, CM (1968). " Criterios estereoquímicos para polipéptidos y proteínas. V. Conformaciones de un sistema de tres unidades de péptidos unidos" (PDF). Biopolímeros. 6 (10): 1425–1436. doi:10.1002/bip.1968.360061006. Hdl:2027.42/37819. PMID 5685102. S2CID 587353535.
- ^ Sibanda, BL; Blundell, TL; Thornton, JM (20 de abril de 1989). "Conformación de beta-hairpins en estructuras de proteínas. Clasificación sistemática con aplicaciones para modelar por homología, fijación de densidad de electrones e ingeniería de proteínas". Journal of Molecular Biology. 206 (4): 759–77. doi:10.1016/0022-2836(89)90583-4. PMID 2500530.
- ^ Dunker AK, Lawson JD, Brown CJ, Williams RM, Romero P, Oh JS, Oldfield CJ, Campen AM, Ratliff CM, Hipps KW, Ausio J, Nissen MS, Reeves R, Kang C, Kissinger CR, Bailey RW, Griswold MD, Chiu W, Garner EC, Obradovic Z (2001). "Proteína intrínsecamente desordenada". Journal of Molecular Graphics " Modelling. 19 (1): 26–59. CiteSeerX 10.1.1.113.556. doi:10.1016/s1093-3263(00)00138-8. PMID 11381529.
- ^ Bu Z, Callaway DJ (2011). "¡Los proteínas se mueven! Dinámica de proteínas y alostería de largo alcance en señalización celular". Estructura de proteínas y enfermedades. Avances en Química Proteína y Biología Estructural. Vol. 83. pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668.
- ^ Compiani M, Capriotti E (Dec 2013). "Metodologías computacionales y teóricas para el plegado de proteínas" (PDF). Bioquímica. 52 (48): 8601–24. doi:10.1021/bi4001529. PMID 24187909. Archivado desde el original (PDF) el 2015-09-04.
Enlaces externos
- BetaTPred3 - Plataforma de Insilico para predecir e iniciar los betavoces en una proteína en la ubicación deseada
- NetTurnP - Predicción de regiones de vuelta de beta en secuencias de proteínas
- BetaTPred - Predicción de Beta Envuelve proteínas usando algoritmos estadísticos
Literatura
Estas referencias están ordenadas por fecha.
- Venkatachalam CM. (1968). " Criterios estereoquímicos para polipéptidos y proteínas. V. Conformación de un sistema de tres unidades de péptidos vinculadas". Biopolímeros. 6 (10): 1425–36. doi:10.1002/bip.1968.360061006. Hdl:2027.42/37819. PMID 5685102.
- Némethy, George; Printz, Morton P. (1972). "El -Turn, una posible Conformación plegada de la cadena de polipéptidos. Comparación con el β-Turn". Macromolecules. 5 (6): 755–758. doi:10.1021/ma60030a017.
- Lewis PN, Momany FA, Scheraga HA (1973). "Reversales de la cadena en proteínas". Biochim Biophys Acta. 303 (2): 211–29. doi:10.1016/0005-2795(73)90350-4. PMID 4351002.
- Toniolo C.; Benedetti, Ettore (1980). "Conformaciones de péptidos intramolecularmente de hidrógeno". CRC Crit Rev Biochem. 9 (1): 1–44. doi:10.3109/10409238009105471. PMID 6254725.
- Richardson JS. (1981). "La anatomía y taxonomía de la estructura de proteínas". Avances en Química Proteína. 34: 167–339. doi:10.1016/S0065-3233(08)60520-3. ISBN 9780120342341. PMID 7020376. Archivado desde el original en 2019-02-10. Retrieved 2009-01-03.
- Rose GD, Gierasch LM, Smith JA (1985). "Turnas en péptidos y proteínas". Avances en Química Proteína. 37: 1–109. doi:10.1016/S0065-3233(08)60063-7. ISBN 9780120342372. PMID 2865874.
- Milner-White EJ, Poet R (1987). "Loops, bulges, turns and hairpins in proteins". Tendencias Biochem Sci. 12: 189–192. doi:10.1016/0968-0004(87)90091-0.
- Wilmot CM, Thornton JM (1988). "Análisis y predicción de los diferentes tipos de vuelta beta en proteínas". J Mol Biol. 203 (1): 221–32. doi:10.1016/0022-2836(88)90103-9. PMID 3184187.
- Sibanda, B.L.; Blundell, T.L.; Thornton, J.M. (1989). "Conformación de β-hairpinas en estructuras de proteínas: Clasificación sistemática con aplicaciones para modelar por homología, fijación de densidad de electrones e ingeniería de proteínas". Journal of Molecular Biology. 206 (4): 759–777. doi:10.1016/0022-2836(89)90583-4. PMID 2500530.
- Milner-White, E (1990). "Situaciones de giros gamma en proteínasSu relación con alfa-helices, hojas beta y sitios de unión ligand". J. Mol. Biol. 216 (2): 385–397. doi:10.1016/S0022-2836(05)80329-8. PMID 2254936.
- Hutchinson, E.G.; Thornton, J.M. (1994). "Un conjunto revisado de potenciales para la formación de β-volución en proteínas". Protein Science. 3 (12): 2207–2216. doi:10.1002/pro.5560031206. PMC 2142776. PMID 7756980.
- Pavone V, Gaeta G, Lombardi A, Nastri F, Maglio O, Isernia C, Saviano M (1996). "Descubriendo estructuras secundarias de proteínas: clasificación y descripción de vueltas alfa aisladas". Biopolímeros. 38 (6): 705–21. doi:10.1002/(SICI)1097-0282(199606)38:6 =705::AID-BIP3 confianza3.0.CO;2-V. PMID 8652792.
- Rajashankar KR, Ramakumar S (1996). "Los giros en proteínas y péptidos: Clasificación, conformación, ocurrencia, hidratación y secuencia". Protein Sci. 5 (5): 932–46. doi:10.1002/pro.5560050515. PMC 2143406. PMID 8732765. Archivado desde el original el 2009-05-24.
- Shapovalov, M; Vucetic, S; Dunbrack RL, Jr (marzo 2019). "Una nueva agrupación y nomenclatura para giros beta derivados de estructuras de proteínas de alta resolución". PLOS Biología computacional. 15 (3): e1006844. Código:2019PLSCB..15E6844S. doi:10.1371/journal.pcbi.1006844. PMC 6424458. PMID 30845191.
