Fi X 174

El bacteriófago phi X 174 (o ΦX174) es un virus de ADN monocatenario (ssDNA) que infecta Escherichia coli. Este virus fue aislado en 1935 por Nicolas Bulgakov en el laboratorio Félix d'Hérelle del Instituto Pasteur, a partir de muestras recogidas en las alcantarillas de París. Su caracterización y el estudio de su mecanismo de replicación se realizaron a partir de la década de 1950. Fue el primer genoma basado en ADN que se secuenció. Este trabajo fue completado por Fred Sanger y su equipo en 1977. En 1962, Walter Fiers y Robert Sinsheimer ya habían demostrado la circularidad física, covalentemente cerrada, del ADN ΦX174. El premio Nobel Arthur Kornberg utilizó ΦX174 como modelo para demostrar por primera vez que el ADN sintetizado en un tubo de ensayo mediante enzimas purificadas podía producir todas las características de un virus natural, marcando el comienzo de la era de la biología sintética. En 1972-1974, Jerard Hurwitz, Sue Wickner y Reed Wickner, con sus colaboradores, identificaron los genes necesarios para producir las enzimas que catalizarían la conversión de la forma monocatenaria del virus a la forma replicativa bicatenaria. En 2003, el grupo de Craig Venter informó que el genoma de ΦX174 fue el primero en ensamblarse completamente in vitro a partir de oligonucleótidos sintetizados. La partícula del virus ΦX174 también se ha ensamblado con éxito in vitro. En 2012, se demostró cómo su genoma altamente superpuesto puede descomprimirse por completo y seguir siendo funcional.
Genoma

Este bacteriófago tiene un genoma de ADN monocatenario circular de sentido [+] de 5.386 nucleótidos. El contenido de GC del genoma es del 44% y el 95% de los nucleótidos pertenecen a genes codificantes. Debido al patrón de bases equilibradas del genoma, se utiliza como ADN de control para los secuenciadores de Illumina.
Genes
ΦX174 codifica 11 genes, nombrados como letras consecutivas del alfabeto en el orden en que fueron descubiertos, con la excepción de A*, que es un codón de inicio alternativo dentro de los genes A grandes. Se cree que sólo los genes A* y K no son esenciales, aunque existen algunas dudas sobre A* porque su codón de inicio podría cambiarse a ATT pero no a cualquier otra secuencia. Ahora se sabe que es probable que el ATT todavía sea capaz de producir proteínas dentro de E. coli y, por lo tanto, este gen puede ser esencial.
La primera mitad del genoma ΦX174 presenta altos niveles de superposición de genes: ocho de los 11 genes se superponen en al menos un nucleótido. Se ha demostrado que estas superposiciones no son esenciales, aunque el fago refactorizado al que se le eliminaron todas las superposiciones de genes tenía una aptitud menor que el tipo salvaje.
El fago ΦX174 se ha utilizado para intentar establecer la ausencia de información genética no descubierta mediante una prueba de "prueba por síntesis" acercarse.
Transcriptoma
En 2020, se generó el transcriptoma de ΦX174. Las características notables del transcriptoma ΦX174 son una serie de hasta cuatro promotores relativamente débiles en serie con hasta cuatro terminadores independientes de Rho (intrínsecos) y un terminador dependiente de Rho.
Proteínas
ΦX174 codifica 11 proteínas.
| Proteína | Copias | Función |
|---|---|---|
| A | — | ADN de RF para iniciar la replicación del círculo de rodamiento; ligados extremos de ADN de phage lineal para formar ADN circular de una sola raya |
| A* | — | Inhibiciones de la replicación de ADN de células anfitrionas; bloquea la superinfección de la phage; no esencial |
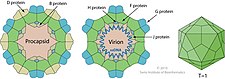
| B | 60 en procapsid | Proteína de andamio interno involucrada en el ensamblaje procapsid |
| C | — | Embalaje de ADN |
| D | 240 en procapsid | Proteína de andamio externo involucrada en el ensamblaje procapsid |
| E | — | Lísis de células anfitrionas |
| F | 60 en virión | proteína capsida mayor |
| G | 60 en virión | Proteína de pico mayor |
| H | 12 en virión | proteína piloto de ADN (o proteína de pico menor) |
| J | 60 en virión | Se une al nuevo ADN de phage de una sola raya; acompaña el ADN de phage en procapsid |
| K | — | Optimiza el tamaño de la explosión; no esencial |
Proteoma
Recientemente se ha informado de la identificación de todas las proteínas ⋅X174 utilizando espectrometría masiva.
Ciclo de infección
La infección comienza cuando la proteína G se une a los lipopolisacáridos en la superficie de la célula bacteriana huésped. La proteína H (o proteína piloto de ADN) dirige el genoma viral a través de la membrana bacteriana de la bacteria E.coli, muy probablemente a través de una hélice de dominio transmembrana N-terminal prevista. Sin embargo, se ha hecho evidente que la proteína H es una proteína multifuncional. Esta es la única proteína de la cápside viral de ΦX174 que carece de estructura cristalina por un par de razones. Tiene un bajo contenido aromático y un alto contenido de glicina, lo que hace que la estructura de la proteína sea muy flexible y, además, los átomos de hidrógeno individuales (el grupo R de las glicinas) son difíciles de detectar en la cristalografía de proteínas. Además, la proteína H induce la lisis del huésped bacteriano en altas concentraciones, ya que la hélice transmembrana N-terminal prevista perfora fácilmente la pared bacteriana. Por bioinformática, esta proteína contiene cuatro dominios en espiral predichos que tienen una homología significativa con factores de transcripción conocidos. Además, se determinó que la proteína H de novo era necesaria para la síntesis óptima de otras proteínas virales. Las mutaciones en la proteína H que impiden la incorporación viral se pueden superar cuando se suministran cantidades excesivas de proteína B, la proteína de andamiaje interno.
El ADN se expulsa a través de un canal hidrófilo en el vértice quíntuple. Se entiende que la proteína H reside en esta área, pero la evidencia experimental no ha verificado su ubicación exacta. Una vez dentro de la bacteria huésped, la replicación del genoma [+] ssDNA continúa a través de un intermediario de ADN de sentido negativo. Esto se hace a medida que el genoma del fago se superenrolla y la estructura secundaria formada por dicho superenrollamiento atrae un complejo de proteína primosómica. Esto se transloca una vez alrededor del genoma y sintetiza un [-]ssDNA del genoma original positivo. [+]Los genomas de ADNss para empaquetar en virus se crean a partir de esto mediante un mecanismo de círculo rodante. Este es el mecanismo por el cual el genoma superenrollado de doble cadena es cortado en la cadena positiva por una proteína A codificada por un virus, atrayendo también una ADN polimerasa bacteriana (DNAP) al sitio de escisión. DNAP utiliza la cadena negativa como plantilla para generar ADN con sentido positivo. A medida que se traslada alrededor del genoma, desplaza la cadena externa de ADN ya sintetizado, que queda inmediatamente recubierta por proteínas SSBP. La proteína A escinde el genoma completo cada vez que reconoce la secuencia de origen.
Como la proteína D es la transcripción genética más abundante, es la mayor cantidad de proteína en la procápsida viral. De manera similar, las transcripciones de genes de F, J y G son más abundantes que las de H, ya que la estequiometría de estas proteínas estructurales es 5:5:5:1. Los primosomas son complejos proteicos que unen o unen la enzima helicasa en la plantilla. Los primosomas proporcionan cebadores de ARN para la síntesis de ADN en las hebras.
Filogenética y diversidad
ΦX174 está estrechamente relacionado con otros microviridos, especialmente el fago NC (por ejemplo, NC1, NC7, NC11, NC16, NC37, NC5, NC41, NC56, NC51, etc.) y más lejanamente relacionado con los fagos similares a G4 e incluso relacionado más lejanamente con el fago tipo α3. Rokyta et al. 2006 presentó un árbol filogenético de sus relaciones.
Usos
Evolución experimental
ΦX174 se ha utilizado como organismo modelo en muchos experimentos de evolución.
Biotecnología
ΦX174 se utiliza regularmente como control positivo en la secuenciación de ADN debido a su tamaño de genoma relativamente pequeño en comparación con otros organismos, su contenido de nucleótidos relativamente equilibrado: aproximadamente 23 % G, 22 % C, 24 % A y 31 % T. , es decir, 45 % G+C y 55 % A+T, consulte la entrada NC_001422.1 para conocer su secuencia de 5.386 nucleótidos de longitud. Los instrumentos de secuenciación de Illumina utilizan ΦX174 como control positivo, y una sola secuenciación de Illumina puede cubrir el genoma de ΦX174 varios millones de veces, lo que lo convierte muy probablemente en el genoma más secuenciado de la historia.
ΦX174 también se utiliza para probar la resistencia del equipo de protección personal a los virus transmitidos por la sangre.
ΦX174 también se ha modificado para permitir la presentación de péptidos (presentación en fagos) de la proteína G de la cápside viral.
Biología sintética
El genoma ΦX174 fue el primer fago clonado en levadura, lo que proporciona un dique seco conveniente para las modificaciones del genoma. ΦX174 también fue el primer genoma que se descomprimió completamente y se eliminaron todas las superposiciones de genes. El efecto de estos cambios resultó en una reducción significativa de la unión al huésped, la desregulación de la expresión de proteínas y la sensibilidad al calor.
Contenido relacionado
ArXiv
Béfalo
Biología estructural
Academias Nacionales de Ciencias, Ingeniería y Medicina
Bill mollison