Estructura primaria de proteínas

Estructura primaria de proteína es la secuencia lineal de aminoácidos en un péptido o proteína. Por convención, la estructura primaria de una proteína se informa comenzando desde el extremo amino-terminal (N) hasta el extremo carboxilo-terminal (C). La biosíntesis de proteínas es más comúnmente realizada por los ribosomas en las células. Los péptidos también se pueden sintetizar en el laboratorio. Las estructuras primarias de proteínas pueden secuenciarse directamente o inferirse de secuencias de ADN.
Formación
Biológica
(feminine)Los aminoácidos se polimerizan a través de enlaces peptídicos para formar una columna vertebral larga, con las diferentes cadenas laterales de aminoácidos sobresaliendo a lo largo de ella. En los sistemas biológicos, las proteínas son producidas durante la traducción por los ribosomas de una célula. Algunos organismos también pueden producir péptidos cortos mediante la síntesis de péptidos no ribosómicos, que a menudo usan aminoácidos distintos del estándar 20, y pueden ciclarse, modificarse y entrecruzarse.
Química
(feminine)Los péptidos se pueden sintetizar químicamente a través de una variedad de métodos de laboratorio. Los métodos químicos suelen sintetizar péptidos en el orden opuesto (comenzando en el extremo C-terminal) a la síntesis biológica de proteínas (comenzando en el extremo N-terminal).
Notación
La secuencia de proteínas generalmente se anota como una cadena de letras, enumerando los aminoácidos que comienzan en el extremo amino-terminal hasta el extremo carboxilo-terminal. Se puede utilizar un código de tres letras o de una sola letra para representar los 20 aminoácidos naturales, así como mezclas o aminoácidos ambiguos (similar a la notación de ácido nucleico).
Los péptidos pueden secuenciarse directamente o deducirse de secuencias de ADN. Ahora existen grandes bases de datos de secuencias que recopilan secuencias de proteínas conocidas.
| Amino Acid | 3-Letter | 1-Carta |
|---|---|---|
| Alanine | Ala | A |
| Arginine | Arg | R |
| Asparagine | Asn | N |
| Ácido aspartico | Asp | D |
| Cysteine | Cys | C |
| Ácido glutamico | Glu | E |
| Glutamina | Gln | Q |
| Glycine | Gly | G |
| Histidina | Su | H |
| Isoleucine | Ile | I |
| Leucine | Leu | L |
| Lysine | Lys | K |
| Metionina | Met | M |
| Fenilalanina | Phe | F |
| Proline | Pro | P |
| Serine | Ser | S |
| Threonine | # | T |
| Tryptophan | Trp | W |
| Tyrosine | Tyr | Y |
| Valine | Val | V |
| Signatura | Descripción | Residuos representados |
|---|---|---|
| X | Cualquier aminoácido, o desconocido | Todos |
| B | Aspartate or Asparagine | D, N |
| Z | Glutamate o Glutamine | E, Q |
| J | Leucine o Isoleucine | I, L |
| CCPR | Hidrofobia | V, I, L, F, W, M |
| Ω | Aromatic | F, W, Y, H |
| Ψ | Aliphatic | V, I, L, M |
| π | Pequeña | P, G, A, S |
| Especificaciones | Hidrofílico | S, T, H, N, Q, E, D, K, R, Y |
| + | Carga positiva | K, R, H |
| - | Carga negativa | D, E |
Modificación
En general, los polipéptidos son polímeros no ramificados, por lo que su estructura primaria a menudo se puede especificar por la secuencia de aminoácidos a lo largo de su columna vertebral. Sin embargo, las proteínas pueden entrecruzarse, más comúnmente por enlaces disulfuro, y la estructura primaria también requiere especificar los átomos de entrecruzamiento, por ejemplo, especificar las cisteínas involucradas en los enlaces disulfuro de la proteína. Otros enlaces cruzados incluyen desmosina.
Isomerización
Los centros quirales de una cadena de polipéptidos pueden ser objeto de racemización. Aunque no cambia la secuencia, afecta las propiedades químicas de la secuencia. En particular, L-aminoácidos normalmente encontrados en proteínas pueden isomerizar espontáneamente en el Cα α {displaystyle mathrm {}}} átomo para formar D-aminoácidos, que no pueden ser blanqueados por la mayoría de las proteas. Además, proline puede formar trans-isómeros estables en el enlace péptidos.
Modificación postraduccional
Finalmente, la proteína puede sufrir una variedad de modificaciones postraduccionales, que se resumen brevemente aquí.
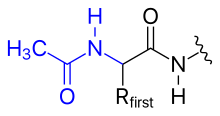
El grupo amino N-terminal de un polipéptido se puede modificar covalentemente, por ejemplo,
- acetilación − − C()=O)− − CH3{displaystyle mathrm {-C(=O)-CH_{3}}
- La carga positiva en el grupo de aminos N-terminal puede eliminarse cambiandolo a un grupo acetil (bloqueo N-terminal).
- formylation − − C()=O)H{displaystyle mathrm {-C(=O)H}
- La metionina N-terminal generalmente se encuentra después de la traducción tiene un N-terminus bloqueado con un grupo de formalo. Este grupo de formal (y a veces el residuo de metionina en sí mismo, si sigue Gly o Ser) es eliminado por la enzima deformilasa.
- piroglutamate
- Un glutamina N-terminal puede atacarse a sí mismo, formando un grupo cíclico piroglutamato.
- myristoylation − − C()=O)− − ()CH2)12− − CH3{displaystyle mathrm {-C(=O)-left(CH_{2}right)_{12}-CH_{3}}
- Similar a la acetilación. En lugar de un simple grupo metil, el grupo miristoil tiene una cola de 14 carbonos hidrofóbicos, lo que lo hace ideal para anclar proteínas a membranas celulares.
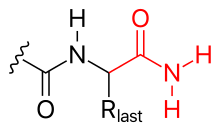
El grupo carboxilato C-terminal de un polipéptido también se puede modificar, por ejemplo,
- amination (véase el gráfico)
- El C-terminus también puede ser bloqueado (por eso, neutralizando su carga negativa) por amination.
- glycosyl phosphatidylinositol (GPI) adjunto
- Glycosyl phosphatidylinositol (GPI) es un grupo de prótesis fosfolípido grande e hidrofóbico que ancla proteínas a membranas celulares. Se adjunta al polipéptido C-terminus a través de un enlace de amida que luego se conecta a la etanolamina, de ahí a azúcares sundrios y finalmente a la fosfatidylinositol lípido.
Finalmente, las cadenas laterales peptídicas también se pueden modificar covalentemente, por ejemplo,
- fosforilación
- Aparte del escote, la fosforilación es quizás la modificación química más importante de las proteínas. Un grupo de fosfato se puede adjuntar al grupo hidroxilo sidechain de residuos de serina, tronina y tirosina, agregando una carga negativa en ese sitio y produciendo un aminoácido antinatural. Tales reacciones son catalizadas por kinases y la reacción inversa es catalizada por fosfatasas. Las tirosinas fosforiladas se utilizan a menudo como "manos" por las cuales las proteínas pueden unirse entre sí, mientras que la fosforilación de Ser/Thr a menudo induce cambios conformacionales, presumiblemente debido a la carga negativa introducida. Los efectos de la fosforilación Ser/Thr a veces se pueden simular mutando el residuo Ser/Thr a glutamato.
- glicosylation
- Un nombre completo para un conjunto de modificaciones químicas muy comunes y muy heterogéneas. Moieties de azúcar se pueden adjuntar a los grupos hidroxilos de la cadena lateral de Ser/Thr o a los grupos de amide de la cadena lateral de Asn. Tales apegos pueden servir muchas funciones, desde la creciente solubilidad hasta el reconocimiento complejo. Toda glucosilación se puede bloquear con ciertos inhibidores, como la tunicamicina.
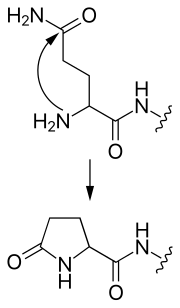
- deamida (formación succinimida)
- En esta modificación, una cadena lateral asparagina o aspartada ataca el siguiente enlace peptide, formando un intermedio succinimide simétrico. Hidrolisis de los productos intermedios ya sea aspartato o el β-aminoácido, iso(Asp). Para la asparagina, cualquiera de los productos resulta en la pérdida del grupo de amida, por lo tanto "deamidación".
- hidroxilatación
- Los residuos de prolina pueden ser hidroxilatos en cualquiera de dos átomos, como puede lisina (en un átomo). La hidroxiprolina es un componente crítico del colágeno, que se vuelve inestable sobre su pérdida. La reacción de hidroxilación es catalizada por una enzima que requiere ácido ascórbico (vitamina C), deficiencias en las que se derivan muchas enfermedades de tejido conectivo como el escorvo.
- metilación
- Varios residuos de proteína pueden ser metilados, sobre todo los grupos positivos de lisina y arginina. Los residuos de arginina interactúan con la columna vertebral del fosfato ácido nucleico y comúnmente forman enlaces de hidrógeno con los residuos de base, particularmente guanina, en complejos de proteínas-DNA. Los residuos de lisina se pueden metilar de forma doble e incluso triplicada. La metilación sí. no alterar la carga positiva en la cadena lateral, sin embargo.
- acetilación
- La acetilación de los grupos de amino lisina es químicamente análoga a la acetilación de la N-terminus. Funcionalmente, sin embargo, la acetilación de residuos de lisina se utiliza para regular la unión de proteínas a ácidos nucleicos. La cancelación de la carga positiva en la lisina debilita la atracción electrostática para los ácidos nucleicos (negativamente cargados).
- sulfación
- Las tirosinas pueden ser sulfizadas en sus O.. {displaystyle mathrm} átomo. Algo inusual, esta modificación ocurre en el aparato Golgi, no en el reticulum endoplasmático. Similar a las tirosinas fosforiladas, las tirosinas sulfadas se utilizan para el reconocimiento específico, por ejemplo, en los receptores de quimioquinas en la superficie celular. Como con la fosforilación, la sulfación añade una carga negativa a un sitio previamente neutral.
- prenylation y palmitoilación − − C()=O)− − ()CH2)14− − CH3{displaystyle mathrm {-C(=O)-left(CH_{2}right)_{14}-CH_{3}}
- El isopreno hidrofóbico (por ejemplo, grupos de farnesilo, geranilo y geranilgeranilo) y grupos de palmitoilo se pueden añadir a los grupos Sγ γ {displaystyle mathrm {cH00}} átomo de residuos de cisteína para anclar proteínas a membranas celulares. A diferencia de los anclajes GPI y myritoyl, estos grupos no se añaden necesariamente en el termini.
- carboxylation
- Una modificación relativamente rara que añade un grupo extra de carboxilatos (y, por lo tanto, una doble carga negativa) a una cadena lateral de glutamato, produciendo un residuo de Gla. Esto se utiliza para fortalecer la unión a iones metálicos " duros" como el calcio.
- ADP-ribosylation
- El gran grupo ADP-ribosyl se puede transferir a varios tipos de cadenas laterales dentro de proteínas, con efectos heterogéneos. Esta modificación es un objetivo para las poderosas toxinas de bacterias dispares, por ejemplo, Vibrio cholerae, Corynebacterium diphtheriae y Bordetella pertussis.
- ubiquitación y SUMOYA
- Varias proteínas de longitud completa, plegadas se pueden adjuntar en su C-termini a los grupos de amonio de cadena lateral de lisas de otras proteínas. La Ubiquitina es la más común de estos, y generalmente indica que la proteína que contiene ubiquitina debe ser degradada.
La mayoría de las modificaciones polipeptídicas enumeradas anteriormente se producen posteriormente a la traducción, es decir, después de que la proteína se ha sintetizado en el ribosoma, normalmente en el retículo endoplásmico, un orgánulo subcelular de la célula eucariota.
Los químicos han aplicado muchas otras reacciones químicas (por ejemplo, la cianilación) a las proteínas, aunque no se encuentran en los sistemas biológicos.
Escisión y ligadura
Además de las enumeradas anteriormente, la modificación más importante de la estructura primaria es la escisión de péptidos (por hidrólisis química o por proteasas). Las proteínas a menudo se sintetizan en forma de precursor inactivo; típicamente, un segmento N-terminal o C-terminal bloquea el sitio activo de la proteína, inhibiendo su función. La proteína se activa al escindir el péptido inhibidor.
Algunas proteínas incluso tienen el poder de escindirse. Por lo general, el grupo hidroxilo de una serina (rara vez, treonina) o el grupo tiol de un residuo de cisteína atacarán el carbono carbonilo del enlace peptídico anterior, formando un intermedio con enlace tetraédrico [clasificado como hidroxioxazolidina (Ser/Thr) o hidroxitiazolidina (Cys) intermedio]. Este intermedio tiende a volver a la forma de amida, expulsando al grupo atacante, ya que la forma de amida suele verse favorecida por la energía libre (presumiblemente debido a la fuerte estabilización por resonancia del grupo peptídico). Sin embargo, las interacciones moleculares adicionales pueden hacer que la forma de amida sea menos estable; en su lugar, se expulsa el grupo amino, lo que da como resultado un enlace éster (Ser/Thr) o tioéster (Cys) en lugar del enlace peptídico. Esta reacción química se llama cambio de N-O acilo.
El enlace éster/tioéster se puede resolver de varias formas:
- La hidrólisis simple dividirá la cadena de polipéptidos, donde el grupo amino desplazado se convierte en el nuevo N-terminus. Esto se ve en la maduración de glicosylasparaginasa.
- Una reacción β-elimination también divide la cadena, pero resulta en un grupo pyruvoyl en el nuevo N-terminus. Este grupo pyruvoyl puede ser utilizado como covalentemente unido cofactor catalítico en algunas enzimas, especialmente decarboxylasas como S-adenosylmethionine decarboxylase (SAMDC) que explotan el poder electron-retirador del grupo piruvoyl.
- Transesterificación intramolecular, resultando en ramificados polipéptidos. En las inteínas, el nuevo vínculo ester se rompe por un ataque intramolecular por el pronto-a-be C-terminal asparagine.
- La transesterificación intermolecular puede transferir un segmento entero de un polipéptido a otro, como se ve en el autoprocesamiento de la proteína Hedgehog.
Compresión de secuencia
La compresión de secuencias de aminoácidos es una tarea comparativamente desafiante. Los compresores de secuencias de aminoácidos especializados existentes son bajos en comparación con los compresores de secuencias de ADN, principalmente debido a las características de los datos. Por ejemplo, modelar inversiones es más difícil debido a la pérdida de información inversa (desde los aminoácidos hasta la secuencia de ADN). El compresor de datos sin pérdida actual que proporciona mayor compresión es AC2. AC2 combina varios modelos de contexto utilizando redes neuronales y codifica los datos mediante codificación aritmética.
Historia
La propuesta de que las proteínas eran cadenas lineales de α-aminoácidos fue realizada casi simultáneamente por dos científicos en la misma conferencia en 1902, la 74.ª reunión de la Sociedad Alemana de Científicos y Médicos, celebrada en Karlsbad. Franz Hofmeister hizo la propuesta por la mañana, basándose en sus observaciones de la reacción de biuret en las proteínas. Hofmeister fue seguido unas horas más tarde por Emil Fischer, quien había acumulado una gran cantidad de detalles químicos que respaldaban el modelo de enlace peptídico. Para completar, la propuesta de que las proteínas contenían enlaces amida fue realizada ya en 1882 por el químico francés E. Grimaux.
A pesar de estos datos y la evidencia posterior de que las proteínas digeridas proteolíticamente produjeron solo oligopéptidos, la idea de que las proteínas eran polímeros de aminoácidos lineales y no ramificados no se aceptó de inmediato. Algunos científicos muy respetados, como William Astbury, dudaron de que los enlaces covalentes fueran lo suficientemente fuertes para mantener unidas moléculas tan largas; temían que las agitaciones térmicas hicieran pedazos moléculas tan largas. Hermann Staudinger enfrentó prejuicios similares en la década de 1920 cuando argumentó que el caucho estaba compuesto de macromoléculas.
Así, surgieron varias hipótesis alternativas. El hipotesis de proteína coloides afirmó que las proteínas eran conjuntos coloides de moléculas más pequeñas. Esta hipótesis fue refutada en la década de 1920 por mediciones de ultracentrifugación de Theodor Svedberg que mostraban que las proteínas tenían un peso molecular bien definido y reproducible y por mediciones electroforeticas de Arne Tiselius que indicaban que las proteínas eran moléculas individuales. Una segunda hipótesis, la Hipótesis ciclol avanzado por Dorothy Wrinch, propuso que el polipéptido lineal se sometiera a un reordenamiento del ciclol químico C=O + HN → → {displaystyle rightarrow } C(OH)-N que cruzaba sus grupos de anida de columna vertebral, formando un bidimensional tela. Otras estructuras primarias de proteínas fueron propuestas por varios investigadores, como las modelo diketopiperazine of Emil Abderhalden and the modelo pyrrol/piperidine de Troensegaard en 1942. Aunque nunca se dio mucha credibilidad, estos modelos alternativos fueron finalmente refutados cuando Frederick Sanger secuencia con éxito la insulina y por la determinación cristalográfica de la mioglobina y la hemoglobina de Max Perutz y John Kendrew.
Estructura primaria en otras moléculas
Se puede decir que cualquier heteropolímero de cadena lineal tiene una "estructura primaria" por analogía con el uso del término para proteínas, pero este uso es raro en comparación con el uso extremadamente común en referencia a las proteínas. En el ARN, que también tiene una estructura secundaria extensa, la cadena lineal de bases generalmente se conoce simplemente como la 'secuencia'. como lo es en el ADN (que suele formar una doble hélice lineal con poca estructura secundaria). También se puede considerar que otros polímeros biológicos, como los polisacáridos, tienen una estructura primaria, aunque el uso no es estándar.
Relación con la estructura secundaria y terciaria
La estructura primaria de un polímero biológico determina en gran medida la forma tridimensional (estructura terciaria). La secuencia de proteínas se puede utilizar para predecir características locales, como segmentos de estructura secundaria o regiones transmembrana. Sin embargo, la complejidad del plegamiento de proteínas actualmente prohíbe predecir la estructura terciaria de una proteína a partir de su secuencia únicamente. Conocer la estructura de una secuencia homóloga similar (por ejemplo, un miembro de la misma familia de proteínas) permite una predicción muy precisa de la estructura terciaria mediante modelos de homología. Si se dispone de la secuencia completa de la proteína, es posible estimar sus propiedades biofísicas generales, como su punto isoeléctrico.
Las familias de secuencias a menudo se determinan mediante el agrupamiento de secuencias, y los proyectos de genómica estructural tienen como objetivo producir un conjunto de estructuras representativas para cubrir el espacio de secuencias de posibles secuencias no redundantes.
Notas y referencias
- ^ a b SANGER F (1952). "El arreglo de aminoácidos en proteínas". En M.L. Anson; Kenneth Bailey; John T. Edsall (eds.). Avances en Química Proteína. Vol. 7. pp. 1–67. doi:10.1016/S0065-3233(08)60017-0. ISBN 9780120342075. PMID 14933251.
- ^ Aasland, Rein; Abrams, Charles; Ampe, Christophe; Ball, Linda J.; Bedford, Mark T.; Cesareni, Gianni; Gimona, Mario; Hurley, James H.; Jarchau, Thomas (2002-02-20). "Normalización de la nomenclatura para motivos de péptidos como ligandos de dominios de proteína modulares". Cartas FEBS. 513 (1): 141–144. doi:10.1016/S0014-5793(01)03295-1. ISSN 1873-3468.
- ^ Aasland R, Abrams C, Ampe C, Ball LJ, Bedford MT, Cesareni G, Gimona M, Hurley JH, Jarchau T, Lehto VP, Lemmon MA, Linding R, Mayer BJ, Nagai M, Sudol M, Walter U, Winder SJ (1968-07-01). "Notación de una sola carta para las secuencias de ácido aminoácidos*". European Journal of Biochemistry. 5 (2): 151–153. doi:10.1111/j.1432-1033.1968.tb00350.x. ISSN 1432-1033. PMID 11911894.
- ^ a b Hausman, Robert E.; Cooper, Geoffrey M. (2004). La célula: un enfoque molecular. Washington, D.C.: ASM Press. p. 51. ISBN 978-0-87893-214-6.
- ^ Silva M, Pratas D, Pinho AJ (abril 2021). "AC2: Una herramienta de compresión de proteína eficiente utilizando redes neuronales artificiales y modelos de cache-Hash". Entropy. 23 (5): 530. Bibcode:2021Intrp..23..530S. doi:10.3390/e23050530. PMC8146440. PMID 33925812.
{{cite journal}}: CS1 maint: múltiples nombres: lista de autores (link) - ^ a b Fruton JS (mayo de 1979). "Teorías de la estructura proteica". Ann. N. Y. Acad. Sci. 325 (1): xiv, 1–18. Bibcode:1979NYASA.325....1F. doi:10.1111/j.1749-6632.1979.tb14125.x. PMID 378063. S2CID 39125170.
Contenido relacionado
Pavo
Conservante
Lamiáceas