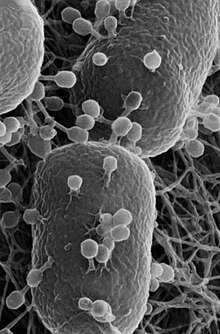
Escherichia coli
Escherichia coli (), también conocida como E. coli (), es una bacteria coliforme gramnegativa, anaeróbica facultativa, con forma de bastón, del género Escherichia que se encuentra comúnmente en el intestino inferior de sangre caliente organismos La mayoría E. coli son inofensivas, pero algunos serotipos (EPEC, ETEC, etc.) pueden causar una intoxicación alimentaria grave en sus huéspedes y, en ocasiones, son responsables de incidentes de contaminación alimentaria que provocan la retirada de productos. La mayoría de las cepas no causan enfermedades en humanos y son parte de la microbiota normal del intestino; tales cepas son inofensivas o incluso beneficiosas para los humanos (aunque estas cepas tienden a ser menos estudiadas que las patógenas). Por ejemplo, algunas cepas de E. coli benefician a sus huéspedes al producir vitamina K2 o al prevenir la colonización del intestino por bacterias patógenas. Estas relaciones mutuamente beneficiosas entre E. coli y los humanos son un tipo de relación biológica mutualista, donde tanto los humanos como la E. coli se benefician mutuamente. E. coli se expulsa al medio ambiente dentro de la materia fecal. La bacteria crece masivamente en materia fecal fresca en condiciones aeróbicas durante tres días, pero luego su número disminuye lentamente.
E. coli y otros anaerobios facultativos constituyen alrededor del 0,1 % de la microbiota intestinal, y la transmisión fecal-oral es la ruta principal a través de la cual las cepas patógenas de la bacteria causan enfermedades. Las células pueden sobrevivir fuera del cuerpo durante un tiempo limitado, lo que las convierte en organismos indicadores potenciales para analizar muestras ambientales en busca de contaminación fecal. Sin embargo, un creciente cuerpo de investigación ha examinado la persistente E. coli que puede sobrevivir durante muchos días y crecer fuera de un huésped.
La bacteria puede crecer y cultivarse de manera fácil y económica en un entorno de laboratorio, y se ha investigado intensamente durante más de 60 años. E. coli es un quimioheterótrofo cuyo medio químicamente definido debe incluir una fuente de carbono y energía. E. coli es el organismo modelo procariótico más ampliamente estudiado y una especie importante en los campos de la biotecnología y la microbiología, donde ha servido como organismo huésped para la mayoría de los trabajos con ADN recombinante. En condiciones favorables, tarda tan solo 20 minutos en reproducirse.
Biología y bioquímica
Tipo y morfología
E. coli es una bacteria coliforme Gram-negativa, anaerobia facultativa, no esporulante. Las células suelen tener forma de bastón y miden aproximadamente 2,0 μm de largo y 0,25–1,0 μm de diámetro, con un volumen celular de 0,6–0,7 μm3. Los antibióticos pueden tratar eficazmente la E. coli infecciones fuera del tracto digestivo y la mayoría de las infecciones intestinales, pero no se usan para tratar infecciones intestinales por una cepa de estas bacterias. Los flagelos que permiten nadar a las bacterias tienen una disposición peritricosa. También se adhiere y se borra de las microvellosidades de los intestinos a través de una molécula de adhesión conocida como intimina.
Metabolismo
E. coli puede vivir en una amplia variedad de sustratos y utiliza fermentación ácida mixta en condiciones anaeróbicas, produciendo lactato, succinato, etanol, acetato y dióxido de carbono. Dado que muchas vías en la fermentación ácida mixta producen hidrógeno gaseoso, estas vías requieren que los niveles de hidrógeno sean bajos, como es el caso cuando E. coli convive con organismos que consumen hidrógeno, como los metanógenos o las bacterias reductoras de sulfato.
Además, E. coli'el metabolismo se puede reconfigurar para usar únicamente CO2 como fuente de carbono para la producción de biomasa. En otras palabras, el metabolismo de este heterótrofo obligado puede alterarse para mostrar capacidades autótrofas mediante la expresión heteróloga de genes de fijación de carbono, así como formiato deshidrogenasa y la realización de experimentos de evolución de laboratorio. Esto se puede hacer usando formiato para reducir los transportadores de electrones y suministrar el ATP requerido en las vías anabólicas dentro de estos autótrofos sintéticos.
E. coli tiene tres vías glucolíticas nativas: EMPP, EDP y OPPP. El EMPP emplea diez pasos enzimáticos para producir dos piruvatos, dos ATP y dos NADH por molécula de glucosa, mientras que OPPP sirve como ruta de oxidación para la síntesis de NADPH. Aunque la EDP es la más favorable termodinámicamente de las tres vías, E. coli no utiliza el EDP para el metabolismo de la glucosa, confiando principalmente en el EMPP y el OPPP. El EDP permanece principalmente inactivo excepto durante el crecimiento con gluconato.
Represión catabólica
Cuando crecen en presencia de una mezcla de azúcares, las bacterias suelen consumir los azúcares de forma secuencial a través de un proceso conocido como represión catabólica. Al reprimir la expresión de los genes involucrados en el metabolismo de los azúcares menos preferidos, las células generalmente consumirán primero el azúcar que produce la tasa de crecimiento más alta, seguido por el azúcar que produce la siguiente tasa de crecimiento más alta, y así sucesivamente. Al hacerlo, las células se aseguran de que sus recursos metabólicos limitados se utilicen para maximizar la tasa de crecimiento. El ejemplo bien utilizado de esto con E. coli implica el crecimiento de la bacteria en glucosa y lactosa, donde E. coli consumirá glucosa antes que lactosa. La represión catabólica también se ha observado en E. coli en presencia de otros azúcares distintos de la glucosa, como arabinosa y xilosa, sorbitol, ramnosa y ribosa. En E. coli, la represión de catabolitos de glucosa está regulada por el sistema de fosfotransferasa, una cascada de fosforilación de múltiples proteínas que acopla la captación de glucosa y el metabolismo.
Crecimiento cultural
Crecimiento óptimo de E. coli ocurre a 37 °C (99 °F), pero algunas cepas de laboratorio pueden multiplicarse a temperaturas de hasta 49 °C (120 °F). E. coli crece en una variedad de medios de laboratorio definidos, como caldo de lisogenia o cualquier medio que contenga glucosa, fosfato monobásico de amonio, cloruro de sodio, sulfato de magnesio, fosfato dibásico de potasio y agua. El crecimiento puede ser impulsado por la respiración aeróbica o anaeróbica, utilizando una gran variedad de pares redox, incluida la oxidación de ácido pirúvico, ácido fórmico, hidrógeno y aminoácidos, y la reducción de sustratos como oxígeno, nitrato, fumarato, dimetilsulfóxido, y N-óxido de trimetilamina. E. coli está clasificado como anaerobio facultativo. Utiliza oxígeno cuando está presente y disponible. Sin embargo, puede seguir creciendo en ausencia de oxígeno mediante fermentación o respiración anaeróbica. El tipo de respiración es administrado en parte por el sistema de arco. La capacidad de seguir creciendo en ausencia de oxígeno es una ventaja para las bacterias porque su supervivencia aumenta en ambientes donde predomina el agua.
Ciclo celular
El ciclo celular bacteriano se divide en tres etapas. El período B ocurre entre la finalización de la división celular y el comienzo de la replicación del ADN. El período C abarca el tiempo que lleva replicar el ADN cromosómico. El período D se refiere a la etapa entre la conclusión de la replicación del ADN y el final de la división celular. La tasa de duplicación de E. coli es mayor cuando hay más nutrientes disponibles. Sin embargo, la duración de los períodos C y D no cambia, incluso cuando el tiempo de duplicación se vuelve menor que la suma de los períodos C y D. En las tasas de crecimiento más rápidas, la replicación comienza antes de que se complete la ronda de replicación anterior, lo que da como resultado múltiples horquillas de replicación a lo largo del ADN y ciclos celulares superpuestos.
El número de bifurcaciones de replicación en E. coli generalmente sigue a 2n (n = 1, 2 o 3). Esto solo sucede si la replicación se inicia simultáneamente desde todos los orígenes de las replicaciones y se denomina replicación síncrona. Sin embargo, no todas las células de un cultivo se replican sincrónicamente. En este caso, las células no tienen múltiplos de dos horquillas de replicación. Entonces se hace referencia a que el inicio de la replicación es asíncrono. Sin embargo, la asincronía puede ser causada por mutaciones, por ejemplo, en DnaA o en la proteína DiaA asociada al iniciador de DnaA.
Adaptación genética
E. coli y las bacterias relacionadas poseen la capacidad de transferir ADN a través de la conjugación o transducción bacteriana, lo que permite que el material genético se propague horizontalmente a través de una población existente. El proceso de transducción, que utiliza el virus bacteriano llamado bacteriófago, es donde la propagación del gen que codifica la toxina Shiga de la bacteria Shigella a E. coli ayudó a producir E. coli O157:H7, la cepa productora de toxina Shiga de E. coli.
Diversidad
E. coli abarca una enorme población de bacterias que exhiben un alto grado de diversidad tanto genética como fenotípica. La secuenciación del genoma de muchos aislamientos de E. coli y bacterias relacionadas muestra que sería deseable una reclasificación taxonómica. Sin embargo, esto no se ha hecho, en gran parte debido a su importancia médica, y E. coli sigue siendo una de las especies bacterianas más diversas: solo el 20% de los genes en una típica E. coli el genoma es compartido entre todas las cepas.
De hecho, desde el punto de vista más constructivo, los miembros del género Shigella (S. dysenteriae, S. flexneri, S. boydii y S. sonnei) deben clasificarse como E. coli cepas, un fenómeno denominado taxones disfrazados. De manera similar, otras cepas de E. coli (por ejemplo, la cepa K-12 comúnmente utilizada en el trabajo de ADN recombinante) son lo suficientemente diferentes como para merecer una reclasificación.
Una cepa es un subgrupo dentro de la especie que tiene características únicas que la distinguen de otras cepas. Estas diferencias a menudo son detectables solo a nivel molecular; sin embargo, pueden provocar cambios en la fisiología o el ciclo de vida de la bacteria. Por ejemplo, una cepa puede ganar capacidad patógena, la capacidad de utilizar una fuente de carbono única, la capacidad de asumir un nicho ecológico particular o la capacidad de resistir a los agentes antimicrobianos. Diferentes cepas de E. coli a menudo son específicos del huésped, lo que permite determinar la fuente de contaminación fecal en muestras ambientales. Por ejemplo, saber qué E. coli están presentes en una muestra de agua permite a los investigadores hacer suposiciones sobre si la contaminación se originó en un ser humano, otro mamífero o un ave.
Serotipos
Un sistema de subdivisión común de E. coli, pero no se basa en la relación evolutiva, es por serotipo, que se basa en los principales antígenos de superficie (antígeno O: parte de la capa de lipopolisacárido; H: flagelina; antígeno K: cápsula), p. O157:H7). Sin embargo, es común citar solo el serogrupo, es decir, el antígeno O. En la actualidad, se conocen alrededor de 190 serogrupos. La cepa de laboratorio común tiene una mutación que impide la formación de un antígeno O y, por lo tanto, no es tipificable.
Plasticidad y evolución del genoma
Como todas las formas de vida, nuevas cepas de E. coli evolucionan a través de los procesos biológicos naturales de mutación, duplicación de genes y transferencia horizontal de genes; en particular, el 18 % del genoma de la cepa de laboratorio MG1655 se adquirió horizontalmente desde la divergencia de Salmonella. E. coli K-12 y E. coli Las cepas B son las variedades más utilizadas con fines de laboratorio. Algunas cepas desarrollan rasgos que pueden ser dañinos para un animal huésped. Estas cepas virulentas generalmente causan un ataque de diarrea que a menudo es autolimitante en adultos sanos pero que con frecuencia es letal para los niños en el mundo en desarrollo. Las cepas más virulentas, como la O157:H7, causan enfermedades graves o la muerte en los ancianos, los muy jóvenes o los inmunocomprometidos.
Los géneros Escherichia y Salmonella divergieron hace unos 102 millones de años (intervalo de credibilidad: 57–176 mya), un evento que no está relacionado con el anterior (ver Synapsid) divergencia de sus anfitriones: el primero se encuentra en mamíferos y el segundo en aves y reptiles. A esto le siguió una división de un ancestro de Escherichia en cinco especies (E. albertii, E. coli, E. fergusonii, E. hermannii y E. vulneris). El último E. coli ancestro se dividió hace entre 20 y 30 millones de años.
Los experimentos de evolución a largo plazo con E. coli, iniciados por Richard Lenski en 1988, han permitido la observación directa de la evolución del genoma durante más de 65.000 generaciones en el laboratorio. Por ejemplo, E. coli normalmente no tienen la capacidad de crecer aeróbicamente con citrato como fuente de carbono, que se utiliza como criterio de diagnóstico para diferenciar E. coli de otras bacterias estrechamente relacionadas, como Salmonella. En este experimento, una población de E. coli desarrolló inesperadamente la capacidad de metabolizar aeróbicamente el citrato, un cambio evolutivo importante con algunas características de la especiación microbiana.
En el mundo microbiano se puede establecer una relación de depredación similar a la observada en el mundo animal. Considerado, se ha visto que E. coli es presa de múltiples depredadores generalistas, como Myxococcus xanthus. En esta relación depredador-presa se observa una evolución paralela de ambas especies a través de modificaciones genómicas y fenotípicas, en el caso de E. coli las modificaciones se modifican en dos aspectos implicados en su virulencia como son la producción de mucoide (producción excesiva de alginato de ácido exoplásmico) y la supresión del gen OmpT, produciendo en futuras generaciones una mejor adaptación de una de las especies que es contrarrestado por la evolución del otro, siguiendo un modelo coevolutivo demostrado por la hipótesis de la Reina Roja.
Cepa neotípica
E. coli es la especie tipo del género (Escherichia) y a su vez Escherichia es el género tipo de la familia Enterobacteriaceae, de donde no procede el apellido el género Enterobacter + "i" (sic.) + "aceae", pero de "enterobacterium" + "aceas" (Enterobacterium no es un género, sino un nombre trivial alternativo a bacteria entérica).
Se cree que la cepa original descrita por Escherich se ha perdido, por lo que se eligió una nueva cepa tipo (neotipo) como representante: la cepa neotipo es U5/41T, también conocida bajo el depósito nombra DSM 30083, ATCC 11775 y NCTC 9001, que es patógena para los pollos y tiene un serotipo O1:K1:H7. Sin embargo, en la mayoría de los estudios, se usaron O157:H7, K-12 MG1655 o K-12 W3110 como E representativo. coli. El genoma de la cepa tipo se ha secuenciado recientemente.
Filogenia de las cepas de E. coli
Se han aislado y caracterizado numerosas cepas pertenecientes a esta especie. Además del serotipo (vide supra), se pueden clasificar según su filogenia, es decir, la historia evolutiva inferida, como se muestra a continuación, donde la especie se divide en seis grupos. Particularmente, el uso de secuencias del genoma completo produce filogenias altamente respaldadas. Con base en tales datos, cinco subespecies de E. coli fueron distinguidos.
El vínculo entre la distancia filogenética ("relación") y la patología es pequeño, por ejemplo las cepas del serotipo O157:H7, que forman un clado ("un grupo exclusivo& #34;)—grupo E a continuación—son todas las cepas enterohemorrágicas (EHEC), pero no todas las cepas de EHEC están estrechamente relacionadas. De hecho, cuatro especies diferentes de Shigella anidan entre E. coli (vide supra), mientras que E. albertii y E. fergusonii están fuera de este grupo. De hecho, todas las especies de Shigella se ubicaron dentro de una sola subespecie de E. coli en un estudio filogenómico que incluyó la cepa tipo, por lo que es difícil una reclasificación acorde. Todas las cepas de investigación comúnmente utilizadas de E. coli pertenecen al grupo A y se derivan principalmente de la cepa K-12 de Clifton (λ+ F+; O16) y en menor grado de la cepa Bacillus coli de d'Herelle's (cepa B) (O7).
Salmonella enterica | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Genómica
La primera secuencia completa de ADN de una E. coli (derivado de la cepa de laboratorio K-12 MG1655) se publicó en 1997. Es una molécula de ADN circular de 4,6 millones de pares de bases de longitud, que contiene 4288 genes codificadores de proteínas anotados (organizados en 2584 operones), siete ARN ribosomal (ARNr) operones y 86 genes de ARN de transferencia (ARNt). A pesar de haber sido objeto de intensos análisis genéticos durante unos 40 años, muchos de estos genes eran desconocidos anteriormente. Se encontró que la densidad de codificación era muy alta, con una distancia media entre genes de solo 118 pares de bases. Se observó que el genoma contenía un número significativo de elementos genéticos transponibles, elementos repetidos, profagos crípticos y restos de bacteriófagos.
Se conocen más de trescientas secuencias genómicas completas de las especies Escherichia y Shigella. La secuencia del genoma de la cepa tipo de E. coli se agregó a esta colección antes de 2014. La comparación de estas secuencias muestra una cantidad notable de diversidad; solo alrededor del 20% de cada genoma representa secuencias presentes en cada uno de los aislados, mientras que alrededor del 80% de cada genoma puede variar entre aislados. Cada genoma individual contiene entre 4000 y 5500 genes, pero el número total de genes diferentes entre todos los E. coli cepas (el pangenoma) supera las 16.000. Se ha interpretado que esta gran variedad de genes componentes significa que dos tercios de las E. coli el pangenoma se originó en otras especies y llegó a través del proceso de transferencia horizontal de genes.
Nomenclatura de genes
Genes en E. coli generalmente se nombran de acuerdo con la nomenclatura uniforme propuesta por Demerec et al. Los nombres de genes son acrónimos de 3 letras que se derivan de su función (cuando se conoce) o fenotipo mutante y están en cursiva. Cuando varios genes tienen el mismo acrónimo, los diferentes genes se designan con una letra mayúscula después del acrónimo y también en cursiva. Por ejemplo, recA lleva el nombre de su papel en la recombinación homóloga más la letra A. Los genes funcionalmente relacionados se llaman recB, recC, recD etc. Las proteínas se nombran con siglas en mayúsculas, p. RecA, RecB, etc. Cuando el genoma de E. coli cepa K-12 substr. Se secuenció MG1655, todos los genes codificadores de proteínas conocidos o previstos se numeraron (más o menos) en su orden en el genoma y se abreviaron con números b, como b2819 (= recD). El "b" los nombres se crearon en honor a Fred Blattner, quien dirigió el esfuerzo de secuenciación del genoma. Se introdujo otro sistema de numeración con la secuencia de otro E. coli subcepa K-12, W3110, que se secuenció en Japón y, por lo tanto, utiliza números que empiezan por JW... (Japanese W3110), p. JW2787 (= recD). Por lo tanto, recD = b2819 = JW2787. Tenga en cuenta, sin embargo, que la mayoría de las bases de datos tienen su propio sistema de numeración, p. la base de datos EcoGene utiliza EG10826 para recD. Finalmente, los números de ECK se usan específicamente para alelos en la cepa MG1655 de E. coli K-12. Las listas completas de genes y sus sinónimos se pueden obtener de bases de datos como EcoGene o Uniprot.
Proteómica
Proteoma
La secuencia del genoma de E. coli predice 4288 genes que codifican proteínas, de los cuales el 38 por ciento inicialmente no tenía una función atribuida. La comparación con otros cinco microbios secuenciados revela familias de genes ubicuos y de distribución estrecha; muchas familias de genes similares dentro de E. coli también son evidentes. La familia más grande de proteínas parálogas contiene 80 transportadores ABC. El genoma en su conjunto está sorprendentemente organizado con respecto a la dirección local de replicación; guaninas, oligonucleótidos posiblemente relacionados con la replicación y la recombinación, y la mayoría de los genes están tan orientados. El genoma también contiene elementos de secuencia de inserción (IS), restos de fagos y muchos otros parches de composición inusual que indican la plasticidad del genoma a través de la transferencia horizontal.
Varios estudios han investigado experimentalmente el proteoma de E. coli. Para 2006, 1627 (38%) de las proteínas predichas (marcos abiertos de lectura, ORF) se habían identificado experimentalmente. Mateus et al. 2020 detectó 2586 proteínas con al menos 2 péptidos (60% de todas las proteínas).
Modificaciones postraduccionales (PTM)
Aunque muchas menos proteínas bacterianas parecen tener modificaciones postraduccionales (PTM) en comparación con las proteínas eucariotas, un número considerable de proteínas se modifican en E. coli. Por ejemplo, Potel et al. (2018) encontraron 227 fosfoproteínas de las cuales 173 estaban fosforiladas en histidina. Curiosamente, la mayoría de los aminoácidos fosforilados fueron serina (1220 sitios) con solo 246 sitios en histidina y 501 treoninas fosforiladas y 162 tirosinas.
Interactoma
El interactoma de E. coli ha sido estudiada por purificación por afinidad y espectrometría de masas (AP/MS) y analizando las interacciones binarias entre sus proteínas.
Complejos proteicos. Un estudio de 2006 purificó 4339 proteínas de cultivos de la cepa K-12 y encontró socios que interactuaban para 2667 proteínas, muchas de las cuales tenían funciones desconocidas en ese momento. Un estudio de 2009 encontró 5993 interacciones entre proteínas de la misma E. coli, aunque estos datos mostraron poca superposición con los de la publicación de 2006.
Interacciones binarias. Rajagopala et al. (2014) han llevado a cabo cribados sistemáticos de dos híbridos de levadura con la mayoría de E. coli proteínas, y encontró un total de 2.234 interacciones proteína-proteína. Este estudio también integró interacciones genéticas y estructuras de proteínas y mapeó 458 interacciones dentro de 227 complejos de proteínas.
Microbiota normal
E. coli pertenece a un grupo de bacterias conocidas informalmente como coliformes que se encuentran en el tracto gastrointestinal de los animales de sangre caliente. E. coli normalmente coloniza el tracto gastrointestinal de un bebé dentro de las 40 horas posteriores al nacimiento, llegando con alimentos o agua o de las personas que manipulan al niño. En el intestino, E. coli se adhiere a la mucosidad del intestino grueso. Es el principal anaerobio facultativo del tracto gastrointestinal humano. (Los anaerobios facultativos son organismos que pueden crecer en presencia o ausencia de oxígeno). Mientras estas bacterias no adquieran elementos genéticos que codifiquen factores de virulencia, seguirán siendo comensales benignos.
Uso terapéutico
Debido al bajo costo y la velocidad con la que se puede cultivar y modificar en entornos de laboratorio, E. coli es una plataforma de expresión popular para la producción de proteínas recombinantes utilizadas en la terapéutica. Una ventaja de usar E. coli sobre otra plataforma de expresión es que E. coli naturalmente no exporta muchas proteínas al periplasma, lo que facilita la recuperación de una proteína de interés sin contaminación cruzada. El E. coli Las cepas K-12 y sus derivados (DH1, DH5α, MG1655, RV308 y W3110) son las cepas más utilizadas por la industria biotecnológica. E no patógena. coli cepa Nissle 1917 (EcN), (Mutaflor) y E. coli O83:K24:H31 (Colinfant)) se utilizan como agentes probióticos en medicina, principalmente para el tratamiento de diversas enfermedades gastrointestinales, incluida la enfermedad inflamatoria intestinal. Se cree que la cepa EcN podría impedir el crecimiento de patógenos oportunistas, incluidos Salmonella y otros enteropatógenos coliformes, a través de la producción de proteínas de microcina y la producción de sideróforos.
Papel en la enfermedad
La mayoría E. coli no causan enfermedades, viven naturalmente en el intestino, pero las cepas virulentas pueden causar gastroenteritis, infecciones del tracto urinario, meningitis neonatal, colitis hemorrágica y enfermedad de Crohn. Los signos y síntomas comunes incluyen calambres abdominales intensos, diarrea, colitis hemorrágica, vómitos y, a veces, fiebre. En casos más raros, las cepas virulentas también son responsables de la necrosis intestinal (muerte del tejido) y la perforación sin progresar al síndrome urémico hemolítico, peritonitis, mastitis, sepsis y neumonía por gramnegativos. Los niños muy pequeños son más susceptibles a desarrollar enfermedades graves, como el síndrome urémico hemolítico; sin embargo, las personas sanas de todas las edades corren el riesgo de sufrir las graves consecuencias que pueden surgir como resultado de la infección por E. coli.
Algunas cepas de E. coli, por ejemplo O157:H7, puede producir la toxina Shiga (clasificada como agente de bioterrorismo). La toxina Shiga provoca respuestas inflamatorias en las células diana del intestino, dejando lesiones que provocan la diarrea sanguinolenta que es un síntoma de una infección por E. coli productora de toxina Shiga (STEC). Esta toxina además provoca la destrucción prematura de los glóbulos rojos, que luego obstruyen el sistema de filtración del cuerpo, los riñones, en algunos casos raros (generalmente en niños y ancianos) causando el síndrome urémico hemolítico (SUH), que puede provocar insuficiencia renal e incluso la muerte. Los signos del síndrome urémico hemolítico incluyen disminución de la frecuencia de micción, letargo y palidez de las mejillas y dentro de los párpados inferiores. En el 25% de los pacientes con HUS, ocurren complicaciones del sistema nervioso, lo que a su vez provoca accidentes cerebrovasculares. Además, esta tensión provoca la acumulación de líquido (ya que los riñones no funcionan), lo que provoca edema alrededor de los pulmones, las piernas y los brazos. Este aumento en la acumulación de líquido, especialmente alrededor de los pulmones, impide el funcionamiento del corazón, lo que provoca un aumento de la presión arterial.
La E. coli uropatógena (UPEC) es una de las principales causas de infecciones del tracto urinario. Es parte de la microbiota normal del intestino y se puede introducir de muchas maneras. En particular para las mujeres, la dirección de limpieza después de la defecación (limpiarse de atrás hacia adelante) puede conducir a la contaminación fecal de los orificios urogenitales. El coito anal también puede introducir esta bacteria en la uretra masculina, y al cambiar de coito anal a vaginal, el hombre también puede introducir UPEC en el sistema urogenital femenino.
E. coli enterotoxigénica (ETEC) es la causa más común de diarrea del viajero, con hasta 840 millones de casos en todo el mundo en países en desarrollo cada año. La bacteria, que generalmente se transmite a través de alimentos o agua potable contaminados, se adhiere al revestimiento intestinal, donde secreta cualquiera de los dos tipos de enterotoxinas, lo que provoca diarrea acuosa. La tasa y la gravedad de las infecciones son más altas entre los niños menores de cinco años, e incluyen hasta 380 000 muertes al año.
En mayo de 2011, un E. coli, O104:H4, fue objeto de un brote bacteriano que comenzó en Alemania. Ciertas cepas de E. coli son una de las principales causas de enfermedades transmitidas por los alimentos. El brote comenzó cuando varias personas en Alemania se infectaron con la bacteria E. coli enterohemorrágica (EHEC), lo que provocó el síndrome urémico hemolítico (SUH), una emergencia médica que requiere tratamiento urgente. El brote no solo afectó a Alemania, sino también a otros 15 países, incluidas regiones de América del Norte. El 30 de junio de 2011, el Bundesinstitut für Risikobewertung (BfR) alemán (Instituto Federal para la Evaluación de Riesgos, un instituto federal dentro del Ministerio Federal de Alimentación, Agricultura y Protección del Consumidor de Alemania) anunció que las semillas de fenogreco de Egipto probablemente fueron la causa del brote de ECEH.
Algunos estudios han demostrado una ausencia de E. coli en la flora intestinal de sujetos con el trastorno metabólico Fenilcetonuria. Se plantea la hipótesis de que la ausencia de estas bacterias normales afecta la producción de las vitaminas clave B2 (riboflavina) y K2 (menaquinona), vitaminas que están implicadas en muchos procesos fisiológicos. funciones en los seres humanos, como el metabolismo celular y óseo, y por lo tanto contribuye al trastorno.
E. resistente a carbapenémicos . coli (E. coli) productoras de carbapenemasas que son resistentes a la clase de antibióticos carbapenem, considerados los fármacos de último recurso para este tipo de infecciones. Son resistentes porque producen una enzima llamada carbapenemasa que desactiva la molécula del fármaco.
Período de incubación
El tiempo entre la ingestión de la bacteria STEC y la sensación de malestar se denomina "período de incubación". El período de incubación suele ser de 3 a 4 días después de la exposición, pero puede ser tan corto como 1 día o tan largo como 10 días. Los síntomas a menudo comienzan lentamente con dolor abdominal leve o diarrea sin sangre que empeora durante varios días. El SHU, si se presenta, se desarrolla en promedio 7 días después de los primeros síntomas, cuando la diarrea va mejorando.
Diagnóstico
El diagnóstico de la diarrea infecciosa y la identificación de la resistencia a los antimicrobianos se realiza mediante un cultivo de heces con pruebas posteriores de sensibilidad a los antibióticos. Se requiere un mínimo de 2 días y un máximo de varias semanas para cultivar patógenos gastrointestinales. Las tasas de sensibilidad (verdadero positivo) y especificidad (verdadero negativo) para el cultivo de heces varían según el patógeno, aunque una serie de patógenos humanos no se pueden cultivar. Para las muestras con cultivo positivo, la prueba de resistencia a los antimicrobianos tarda de 12 a 24 horas adicionales en realizarse.
Las pruebas diagnósticas moleculares actuales en el punto de atención pueden identificar E. coli y la resistencia a los antimicrobianos en las cepas identificadas mucho más rápido que las pruebas de cultivo y sensibilidad. Las plataformas basadas en micromatrices pueden identificar cepas patógenas específicas de E. coli y E. coli-gens AMR específicos en dos horas o menos con alta sensibilidad y especificidad, pero el tamaño del panel de prueba (es decir, patógenos totales y genes de resistencia a los antimicrobianos) es limitado. Actualmente se están desarrollando nuevas plataformas de diagnóstico de enfermedades infecciosas basadas en la metagenómica para superar las diversas limitaciones del cultivo y todas las tecnologías de diagnóstico molecular actualmente disponibles.
Tratamiento
El pilar del tratamiento es la evaluación de la deshidratación y la reposición de líquidos y electrolitos. Se ha demostrado que la administración de antibióticos acorta el curso de la enfermedad y la duración de la excreción de E. enterotoxigénica. coli (ETEC) en adultos en áreas endémicas y en la diarrea del viajero, aunque la tasa de resistencia a los antibióticos de uso común está aumentando y generalmente no se recomiendan. El antibiótico utilizado depende de los patrones de susceptibilidad en la región geográfica particular. Actualmente, los antibióticos de elección son las fluoroquinolonas o la azitromicina, con un rol emergente para la rifaximina. La rifaximina oral, un derivado semisintético de la rifamicina, es un antibacteriano eficaz y bien tolerado para el tratamiento de adultos con diarrea del viajero no invasiva. La rifaximina fue significativamente más efectiva que el placebo y no menos efectiva que la ciprofloxacina para reducir la duración de la diarrea. Si bien la rifaximina es efectiva en pacientes con E. coli, con predominio de la diarrea del viajero, parece ineficaz en pacientes infectados con enteropatógenos inflamatorios o invasivos.
Prevención
ETEC es el tipo de E. coli en el que se centran la mayoría de los esfuerzos de desarrollo de vacunas. Los anticuerpos contra la LT y las principales CF de ETEC brindan protección contra las CF homólogas que expresan ETEC y producen LT. Se han desarrollado vacunas orales inactivadas que consisten en antígeno de la toxina y células completas, es decir, la subunidad B del cólera recombinante autorizada (rCTB)-WC vacuna contra el cólera Dukoral. Actualmente no hay vacunas autorizadas para ETEC, aunque varias se encuentran en diversas etapas de desarrollo. En diferentes ensayos, la vacuna contra el cólera rCTB-WC proporcionó una protección alta (85-100%) a corto plazo. Una vacuna candidata oral de ETEC que consta de rCTB y E inactivada con formalina. coli que expresan las principales FC se ha demostrado en ensayos clínicos que es segura, inmunogénica y eficaz contra la diarrea grave en viajeros estadounidenses, pero no contra la diarrea por ETEC en niños pequeños en Egipto. Una vacuna ETEC modificada que consta de E. coli que sobreexpresan los principales CF y un toxoide híbrido más parecido a LT llamado LCTBA, se están sometiendo a pruebas clínicas.
Otros métodos de prevención probados para E. coli incluyen lavarse las manos y mejorar el saneamiento y el agua potable, ya que la transmisión se produce a través de la contaminación fecal de los alimentos y el agua. Además, cocinar bien la carne y evitar el consumo de bebidas crudas sin pasteurizar, como jugos y leche, son otros métodos comprobados para prevenir la E. coli. Por último, se debe evitar la contaminación cruzada de utensilios y espacios de trabajo al preparar los alimentos.
Organismo modelo en la investigación de ciencias de la vida

Esta pintura muestra una sección transversal a través de una Escherichia coli celular. La característica pared celular de dos membranas de bacterias gramnegativas se muestra en verde, con muchas cadenas de lipopolysaccharide que se extienden desde la superficie y una red de hilos peptidoglycan cruzados entre las membranas. El genoma de la célula forma un "nucleoide" delimitado, mostrado aquí en amarillo, e interactúa con muchas proteínas de unión de ADN, mostradas en bronceado y naranja. Grandes moléculas solubles, como ribosomas (colorados en púrpura rojiza), ocupan principalmente el espacio alrededor del nucleóide.
Debido a su larga historia de cultivo de laboratorio y facilidad de manipulación, E. coli juega un papel importante en la ingeniería biológica moderna y la microbiología industrial. El trabajo de Stanley Norman Cohen y Herbert Boyer en E. coli, utilizando plásmidos y enzimas de restricción para crear ADN recombinante, se convirtió en la base de la biotecnología.
E. coli es un huésped muy versátil para la producción de proteínas heterólogas, y se han desarrollado varios sistemas de expresión de proteínas que permiten la producción de proteínas recombinantes en E. coli. Los investigadores pueden introducir genes en los microbios utilizando plásmidos que permiten un alto nivel de expresión de proteína, y dicha proteína puede producirse en masa en procesos de fermentación industrial. Una de las primeras aplicaciones útiles de la tecnología del ADN recombinante fue la manipulación de E. coli para producir insulina humana.
Muchas proteínas que antes se consideraban difíciles o imposibles de expresar en E. coli en forma plegada se han expresado con éxito en E. coli. Por ejemplo, las proteínas con múltiples enlaces disulfuro se pueden producir en el espacio periplásmico o en el citoplasma de mutantes que se oxidan lo suficiente como para permitir la formación de enlaces disulfuro, mientras que las proteínas que requieren modificación postraduccional, como la glicosilación para la estabilidad o la función, se han expresado utilizando el sistema de glicosilación ligado a N de Campylobacter jejuni diseñado en E. coli.
Modificado E. coli se han utilizado en el desarrollo de vacunas, biorremediación, producción de biocombustibles, iluminación y producción de enzimas inmovilizadas.
La cepa K-12 es una forma mutante de E. coli que sobreexpresa la enzima fosfatasa alcalina (ALP). La mutación surge debido a un defecto en el gen que codifica constantemente la enzima. Se dice que un gen que produce un producto sin ninguna inhibición tiene actividad constitutiva. Esta forma mutante particular se usa para aislar y purificar la enzima antes mencionada.
La cepa OP50 de Escherichia coli se utiliza para el mantenimiento de cultivos de Caenorhabditis elegans.
La cepa JM109 es una forma mutante de E. coli que es recA y endA deficiente. La cepa se puede utilizar para la detección azul/blanca cuando las células portan el episoma del factor de fertilidad. La falta de recA disminuye la posibilidad de una restricción no deseada del ADN de interés y la falta de endA inhibe la descomposición del ADN plasmídico. Por lo tanto, JM109 es útil para sistemas de clonación y expresión.
Organismo modelo
E. coli se utiliza con frecuencia como organismo modelo en estudios de microbiología. Las cepas cultivadas (por ejemplo, E. coli K12) están bien adaptadas al entorno de laboratorio y, a diferencia de las cepas de tipo salvaje, han perdido su capacidad de prosperar en el intestino. Muchas cepas de laboratorio pierden su capacidad para formar biopelículas. Estas características protegen a las cepas de tipo salvaje de los anticuerpos y otros ataques químicos, pero requieren un gran gasto de energía y recursos materiales. E. coli se utiliza a menudo como un microorganismo representativo en la investigación de nuevos métodos de esterilización y tratamiento del agua, incluida la fotocatálisis. Mediante métodos estándar de recuento en placa, luego de diluciones secuenciales y crecimiento en placas de gel de agar, se puede evaluar la concentración de organismos viables o CFU (Unidades Formadoras de Colonias) en un volumen conocido de agua tratada, lo que permite la evaluación comparativa del rendimiento de los materiales.
En 1946, Joshua Lederberg y Edward Tatum describieron por primera vez el fenómeno conocido como conjugación bacteriana usando E. coli como bacteria modelo, y sigue siendo el modelo principal para estudiar la conjugación. E. coli fue una parte integral de los primeros experimentos para comprender la genética de los fagos, y los primeros investigadores, como Seymour Benzer, utilizaron E. coli y el fago T4 para comprender la topografía de la estructura génica. Antes de la investigación de Benzer, no se sabía si el gen tenía una estructura lineal o si tenía un patrón de ramificación.
E. coli fue uno de los primeros organismos en tener su genoma secuenciado; el genoma completo de E. coli K12 fue publicado por Science en 1997.
De 2002 a 2010, un equipo de la Academia Húngara de Ciencias creó una cepa de Escherichia coli llamada MDS42, que ahora vende Scarab Genomics de Madison, WI, con el nombre de "Limpiar Genoma E. coli", donde se eliminó el 15% del genoma de la cepa parental (E. coli K-12 MG1655) para ayudar en la eficiencia de la biología molecular, eliminando elementos IS, pseudogenes y fagos, lo que da como resultado un mejor mantenimiento de genes tóxicos codificados por plásmidos, que a menudo son inactivados por transposones. La bioquímica y la maquinaria de replicación no se alteraron.
Al evaluar la posible combinación de nanotecnologías con la ecología del paisaje, se pueden generar paisajes de hábitats complejos con detalles a nanoescala. En tales ecosistemas sintéticos, los experimentos evolutivos con E. coli se han realizado para estudiar la biofísica espacial de la adaptación en un chip de biogeografía insular.
En otros estudios, la E. coli se ha utilizado como un microorganismo modelo para comprender los efectos de la microgravedad simulada (en la Tierra) sobre el mismo.
Usos en computación biológica
Desde 1961, los científicos propusieron la idea de circuitos genéticos utilizados para tareas computacionales. La colaboración entre biólogos e informáticos ha permitido diseñar puertas lógicas digitales sobre el metabolismo de E. coli. Como el operón Lac es un proceso de dos etapas, la regulación genética en las bacterias se utiliza para realizar funciones informáticas. El proceso se controla en la etapa de transcripción del ADN en ARN mensajero.
Se están realizando estudios para intentar programar E. coli para resolver problemas matemáticos complicados, como el problema del camino hamiltoniano.
Una computadora para controlar la producción de proteínas de E. coli dentro de las células de levadura. También se ha desarrollado un método para utilizar bacterias para que se comporten como una pantalla LCD.
En julio de 2017, experimentos separados con E. coli publicado en Nature mostró el potencial del uso de células vivas para tareas informáticas y almacenamiento de información. Un equipo formado con colaboradores del Instituto de Biodiseño de la Universidad Estatal de Arizona y el Instituto Wyss de Ingeniería de Inspiración Biológica de Harvard desarrolló una computadora biológica dentro de E. coli que respondió a una docena de entradas. El equipo llamó a la computadora 'ribocomputadora', ya que estaba compuesta de ácido ribonucleico. Mientras tanto, los investigadores de Harvard probaron que es posible almacenar información en bacterias después de archivar con éxito imágenes y películas en el ADN de E vivos. coli células. En 2021, un equipo dirigido por el biofísico Sangram Bagh realizó un estudio con E. coli para resolver problemas de laberinto de 2 × 2 para probar el principio de computación distribuida entre células.
Historia
En 1885, el pediatra germano-austríaco Theodor Escherich descubrió este organismo en las heces de individuos sanos. La llamó Bacterium coli commune porque se encuentra en el colon. Las primeras clasificaciones de procariotas los colocaron en un puñado de géneros en función de su forma y motilidad (en ese momento estaba en vigor la clasificación de Ernst Haeckel de bacterias en el reino Monera).
Bacterium coli era la especie tipo del género ahora inválido Bacterium cuando se reveló que la especie tipo anterior ("Bacterium triloculare") Estaba faltando. Después de una revisión de Bacterium, Migula la reclasificó como Bacillus coli en 1895 y luego la reclasificó en el género recién creado Escherichia, llamado así por su descubridor original, por Aldo Castellani y Albert John Chalmers.
En 1996, el peor brote mundial hasta la fecha de E. coli ocurrió una intoxicación alimentaria en Wishaw, Escocia, que mató a 21 personas. Este número de muertes se superó en 2011, cuando el brote de E. coli O104:H4 en Alemania de 2011, relacionado con los brotes orgánicos de fenogreco, mató a 53 personas.
Usos
E. coli tiene varios usos prácticos además de su uso como vector para experimentos y procesos genéticos. Por ejemplo, E. coli se puede utilizar para generar propano sintético y hormona de crecimiento humana recombinante.
Bases de datos y enlaces externos
Contenido relacionado
EO Wilson
Trastorno bipolar I
Cuerpo