Empalmadosoma
Un spliceosoma es un gran complejo de ribonucleoproteína (RNP) que se encuentra principalmente en el núcleo de las células eucariotas. El spliceosoma se ensambla a partir de pequeños ARN nucleares (snRNA) y numerosas proteínas. Las moléculas pequeñas de ARN nuclear (snRNA) se unen a proteínas específicas para formar un pequeño complejo de ribonucleoproteína nuclear (snRNP, pronunciado "snurps"), que a su vez se combina con otros snRNP para formar un gran complejo de ribonucleoproteína llamado spliceosoma. El spliceosoma elimina los intrones de un pre-ARNm transcrito, un tipo de transcripción primaria. Este proceso se conoce generalmente como empalme. Una analogía es un editor de cine, que recorta selectivamente material irrelevante o incorrecto (equivalente a los intrones) de la película inicial y envía la versión limpia al director para el corte final.
Sin embargo, a veces el ARN dentro del intrón actúa como una ribozima, empalmándose a sí mismo sin el uso de un empalmesoma o enzimas proteicas.
Historia
En 1977, el trabajo de los laboratorios Sharp y Roberts reveló que los genes de los organismos superiores están "divididos" o presente en varios segmentos distintos a lo largo de la molécula de ADN. Las regiones codificantes del gen están separadas por ADN no codificante que no está involucrado en la expresión de proteínas. La estructura del gen dividido se encontró cuando los ARNm adenovirales se hibridaron con fragmentos de escisión de endonucleasa de ADN viral monocatenario. Se observó que los mRNA de los híbridos mRNA-DNA contenían 5' y 3' colas de regiones no enlazadas por hidrógeno. Cuando se usaron fragmentos más grandes de ADN viral, se observaron estructuras bifurcadas de ADN en bucle cuando se hibridaron con los ARNm virales. Se descubrió que las regiones en bucle, los intrones, se eliminan de los ARNm precursores en un proceso que Sharp denominó "empalme". Posteriormente se descubrió que la estructura del gen dividido era común a la mayoría de los genes eucarióticos. Phillip Sharp y Richard J. Roberts recibieron el Premio Nobel de Fisiología o Medicina de 1993 por el descubrimiento de los intrones y el proceso de empalme.
Composición
Cada spliceosoma se compone de cinco pequeños ARN nucleares (ARNsn) y una variedad de factores proteicos asociados. Cuando estos pequeños ARN se combinan con los factores proteicos, forman complejos ARN-proteína llamados snRNP (small nnuclear ribo nucleoproteins, pronunciado "snurps"). Los snRNA que componen el principal spliceosoma se denominan U1, U2, U4, U5 y U6, llamados así porque son ricos en uridina y participan en varias interacciones ARN-ARN y ARN-proteína.
El ensamblaje del empalmosoma se produce en cada pre-ARNm (también conocido como ARN nuclear heterogéneo, ARN-hn) en cada unión exón:intrón. Los intrones de pre-ARNm contienen elementos de secuencia específicos que se reconocen y utilizan durante el ensamblaje del spliceosoma. Estos incluyen el 5' sitio de empalme final, la secuencia del punto de ramificación, el tracto de polipirimidina y el 3' sitio de empalme final. El spliceosoma cataliza la eliminación de intrones y la ligadura de los exones flanqueantes.
Los intrones suelen tener una secuencia de nucleótidos GU en el extremo 5' sitio de empalme final y un AG en el 3' sitio de empalme final. El 3' el sitio de empalme se puede definir aún más por una longitud variable de polipirimidinas, llamado tracto de polipirimidina (PPT), que cumple la función dual de reclutar factores para el 3' sitio de empalme y posiblemente factores de reclutamiento para la secuencia del punto de ramificación (BPS). El BPS contiene la adenosina conservada necesaria para el primer paso del empalme.
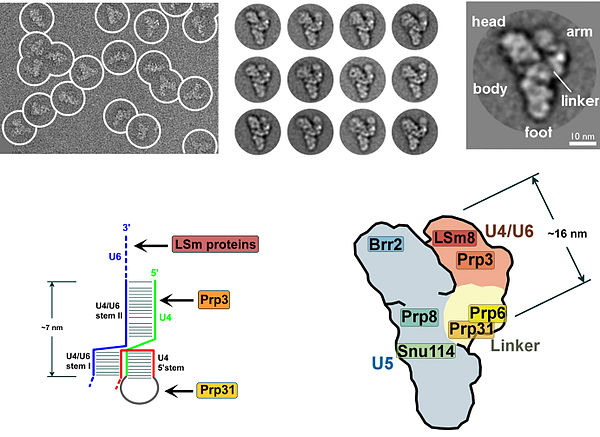
Muchas proteínas exhiben un motivo de unión al zinc, lo que subraya la importancia del zinc en el mecanismo de corte y empalme. La primera reconstrucción de resolución molecular del complejo de ribonucleoproteína nuclear pequeña triple U4/U6.U5 (tri-snRNP) se informó en 2016.
Cryo-EM ha sido aplicado ampliamente por Shi et al. para dilucidar la estructura casi atómica / atómica del spliceosoma tanto en levaduras como en humanos. El marco molecular del spliceosoma a una resolución casi atómica demuestra que el componente Spp42 de U5 snRNP forma un andamio central y ancla el centro catalítico en la levadura. La estructura atómica del spliceosoma humano ilustra el paso II del componente Slu7 que adopta una estructura extendida, preparada para la selección del sitio de empalme 3'. Los cinco metales (asignados como Mg2+) en el complejo de levadura se conservan en el complejo humano.
Empalme alternativo
El empalme alternativo (la recombinación de diferentes exones) es una fuente importante de diversidad genética en eucariotas. Las variantes de empalme se han utilizado para dar cuenta del número relativamente pequeño de genes que codifican proteínas en el genoma humano, actualmente estimado en alrededor de 20.000. Se ha especulado que un gen particular de Drosophila, Dscam, se empalma alternativamente en 38 000 ARNm diferentes, asumiendo que todos sus exones pueden empalmarse independientemente unos de otros.
Montaje
El modelo para la formación del sitio activo del spliceosoma implica un ensamblaje ordenado y gradual de partículas discretas de snRNP en el sustrato pre-ARNm. El primer reconocimiento de pre-ARNm implica la unión de U1 snRNP al 5' sitio de empalme final del pre-ARNm y otros factores asociados no snRNP para formar el complejo de compromiso, o complejo temprano (E) en mamíferos. El complejo de compromiso es un complejo independiente de ATP que compromete el pre-ARNm en la ruta de empalme. U2 snRNP se recluta en la región de la rama a través de interacciones con el componente complejo E U2AF (factor auxiliar U2 snRNP) y posiblemente U1 snRNP. En una reacción dependiente de ATP, U2 snRNP se asocia estrechamente con la secuencia del punto de ramificación (BPS) para formar el complejo A. Un dúplex formado entre U2 snRNP y la región de ramificación pre-mRNA sobresale de la rama de adenosina y la especifica como el nucleófilo para la primera transesterificación.
La presencia de un residuo de pseudouridina en U2 snRNA, casi opuesto al sitio de ramificación, da como resultado una conformación alterada del dúplex de ARN-ARN tras la unión de U2 snRNP. Específicamente, la estructura alterada del dúplex inducido por la pseudouridina coloca al 2' OH de la adenosina abultada en una posición favorable para el primer paso de empalme. El tri-snRNP U4/U5/U6 (ver Figura 1) se recluta para el ensamblaje del spliceosoma para formar el complejo B, y luego de varios reordenamientos, el complejo C se activa para la catálisis. No está claro cómo se recluta el tri-snRNP en el complejo A, pero este proceso puede estar mediado por interacciones proteína-proteína y/o interacciones de apareamiento de bases entre U2 snRNA y U6 snRNA.
El snRNP U5 interactúa con secuencias en el 5' y 3' los sitios de empalme a través del bucle invariable de U5 snRNA y los componentes de la proteína U5 interactúan con el 3' región del sitio de empalme.
Tras el reclutamiento de tri-snRNP, varios reordenamientos de ARN-ARN preceden al primer paso catalítico y se producen más reordenamientos en el spliceosoma catalíticamente activo. Varias de las interacciones ARN-ARN son mutuamente excluyentes; sin embargo, no se sabe qué desencadena estas interacciones, ni el orden de estos reordenamientos. El primer reordenamiento es probablemente el desplazamiento de U1 snRNP del 5' sitio de empalme y formación de una interacción U6 snRNA. Se sabe que U1 snRNP solo se asocia débilmente con empalmesomas completamente formados, y U1 snRNP inhibe la formación de un U6-5' interacción del sitio de empalme en un modelo de oligonucleótido de sustrato que contiene un 5' corto; exón y 5' sitio de empalme. La unión de U2 snRNP a la secuencia de punto de ramificación (BPS) es un ejemplo de una interacción ARN-ARN que desplaza una interacción proteína-ARN. Tras el reclutamiento de U2 snRNP, la proteína de unión a rama SF1 en el complejo de compromiso se desplaza ya que el sitio de unión de U2 snRNA y SF1 son eventos mutuamente excluyentes.
Dentro del snRNA U2, existen otros reordenamientos mutuamente excluyentes que ocurren entre conformaciones en competencia. Por ejemplo, en la forma activa, se favorece el bucle de tallo IIa; en la forma inactiva predomina una interacción mutuamente excluyente entre el bucle y una secuencia aguas abajo. No está claro cómo se desplaza U4 de U6 snRNA, aunque el ARN se ha implicado en el ensamblaje del empalmesoma y puede funcionar para desenrollar U4/U6 y promover la formación de una interacción U2/U6 snRNA. Las interacciones de los bucles de tallo I y II de U4/U6 se disocian y la región liberada del bucle de tallo II de U6 se pliega sobre sí misma para formar un bucle de tallo intramolecular y ya no se requiere U4 en el ensamblaje posterior del spliceosoma. La región del bucle de tallo I liberada de los pares de bases U6 con el snRNA de U2 formando la hélice I de U2/U6. Sin embargo, la estructura de la hélice I es mutuamente excluyente con el 3' la mitad de un interior de 5' región de bucle de tallo de U2 snRNA.
Spliceosoma menor
Algunos eucariotas tienen un segundo spliceosoma, el llamado spliceosoma menor. Un grupo de snRNA menos abundantes, U11, U12, U4atac y U6atac, junto con U5, son subunidades del spliceosoma menor que empalma una clase rara de intrones pre-mRNA, denominados tipo U12. El spliceosoma menor se encuentra en el núcleo como su contraparte principal, aunque hay excepciones en algunas células especializadas, incluidas las plaquetas anucleadas y el dendroplasma (citoplasma dendrítico) de las células neuronales.
