Electroforesis en gel de ácidos nucleicos
La electroforesis de ácidos nucleicos es una técnica analítica utilizada para separar fragmentos de ADN o ARN por tamaño y reactividad. Las moléculas de ácido nucleico que se van a analizar se colocan sobre un medio viscoso, el gel, donde un campo eléctrico induce a los ácidos nucleicos (que están cargados negativamente debido a su cadena principal de azúcar-fosfato) a migrar hacia el ánodo (que está cargado positivamente porque esta es una celda electrolítica en lugar de galvánica). La separación de estos fragmentos se logra aprovechando las movilidades con las que las moléculas de diferentes tamaños pueden atravesar el gel. Las moléculas más largas migran más lentamente porque experimentan más resistencia dentro del gel. Debido a que el tamaño de la molécula afecta su movilidad, los fragmentos más pequeños terminan más cerca del ánodo que los más largos en un período determinado. Después de algún tiempo, se elimina el voltaje y se analiza el gradiente de fragmentación. Para separaciones más grandes entre fragmentos de tamaño similar, se puede aumentar el voltaje o el tiempo de funcionamiento. Las corridas extendidas a través de un gel de bajo voltaje producen la resolución más precisa. Sin embargo, el voltaje no es el único factor para determinar la electroforesis de los ácidos nucleicos.
El ácido nucleico a separar se puede preparar de varias formas antes de la separación por electroforesis. En el caso de moléculas de ADN grandes, el ADN se corta con frecuencia en fragmentos más pequeños usando una endonucleasa de restricción de ADN (o enzima de restricción). En otros casos, como las muestras amplificadas por PCR, las enzimas presentes en la muestra que podrían afectar la separación de las moléculas se eliminan por diversos medios antes del análisis. Una vez que el ácido nucleico se prepara correctamente, las muestras de la solución de ácido nucleico se colocan en los pocillos del gel y se aplica un voltaje a través del gel durante un período de tiempo específico.
Los fragmentos de ADN de diferentes longitudes se visualizan utilizando un tinte fluorescente específico para el ADN, como el bromuro de etidio. El gel muestra bandas correspondientes a diferentes poblaciones de moléculas de ácido nucleico con diferente peso molecular. El tamaño del fragmento generalmente se informa en "nucleótidos", "pares de bases" o "kb" (para miles de pares de bases) dependiendo de si se ha separado el ácido nucleico monocatenario o bicatenario. La determinación del tamaño de los fragmentos se realiza típicamente por comparación con marcadores de ADN disponibles en el mercado que contienen fragmentos de ADN lineales de longitud conocida.
Los tipos de gel más utilizados para la electroforesis de ácidos nucleicos son la agarosa (para moléculas de ADN relativamente largas) y la poliacrilamida (para alta resolución de moléculas de ADN cortas, por ejemplo, en la secuenciación de ADN). Los geles se han corrido convencionalmente en una "placa" formato como el que se muestra en la figura, pero la electroforesis capilar se ha vuelto importante para aplicaciones como la secuenciación de ADN de alto rendimiento. Las técnicas de electroforesis utilizadas en la evaluación del daño del ADN incluyen la electroforesis en gel alcalino y la electroforesis en gel de campo pulsado.
Para segmentos de ADN cortos, como ADN de doble cadena de 20 a 60 pb, ejecutarlos en gel de poliacrilamida (PAGE) brindará una mejor resolución (condición nativa). De manera similar, el ARN y el ADN monocatenario se pueden ejecutar y visualizar mediante geles PAGE que contienen agentes desnaturalizantes como la urea. Los geles PAGE se utilizan ampliamente en técnicas como la huella de ADN, EMSA y otras técnicas de interacción ADN-proteína.
La medición y el análisis se realizan principalmente con un software de análisis de gel especializado. Los resultados de la electroforesis capilar generalmente se muestran en una vista de seguimiento llamada electroferograma.
Factores que afectan la migración de ácidos nucleicos
Varios factores pueden afectar la migración de los ácidos nucleicos: la dimensión de los poros del gel, el voltaje utilizado, la fuerza iónica del tampón y la concentración del colorante intercalado, como el bromuro de etidio, si se utiliza durante la electroforesis.
Tamaño del ADN
El gel tamiza el ADN según el tamaño de la molécula de ADN, por lo que las moléculas más pequeñas viajan más rápido. El ADN de doble cadena se mueve a una velocidad que es aproximadamente inversamente proporcional al logaritmo del número de pares de bases. Sin embargo, esta relación se rompe con fragmentos de ADN muy grandes y no es posible separarlos mediante electroforesis en gel de agarosa estándar. El límite de resolución depende de la composición del gel y la intensidad del campo. y la movilidad del ADN circular más grande puede verse más afectada que la del ADN lineal por el tamaño de los poros del gel. La separación de fragmentos de ADN muy grandes requiere electroforesis en gel de campo pulsado (PFGE). En la electroforesis en gel de inversión de campo (FIGE, un tipo de PFGE), es posible tener "inversión de banda" - donde las moléculas grandes pueden moverse más rápido que las moléculas pequeñas.
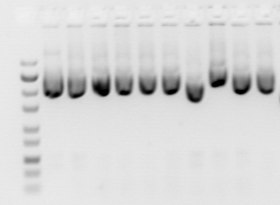
Conformación del ADN
La conformación de la molécula de ADN puede afectar significativamente el movimiento del ADN, por ejemplo, el ADN superenrollado generalmente se mueve más rápido que el ADN relajado porque está muy enrollado y, por lo tanto, es más compacto. En una preparación de ADN de plásmido normal, pueden estar presentes múltiples formas de ADN, y el gel de la electroforesis de los plásmidos normalmente mostraría una banda principal que sería la forma superenrollada negativamente, mientras que otras formas de ADN podrían aparecer como bandas menores más débiles. Estas bandas menores pueden ser ADN mellado (forma circular abierta) y la forma circular cerrada y relajada que normalmente corre más lentamente que el ADN superenrollado, y la forma monocatenaria (que a veces puede aparecer dependiendo de los métodos de preparación) puede moverse por delante del ADN superenrollado.. Sin embargo, la velocidad a la que se mueven las diversas formas puede cambiar usando diferentes condiciones de electroforesis, por ejemplo, el ADN lineal puede funcionar más rápido o más lento que el ADN superenrollado según las condiciones, y la movilidad del ADN circular más grande puede verse más afectada que la del ADN lineal por el poro. tamaño del gel. A menos que se utilicen marcadores de ADN superenrollados, el tamaño de un plásmido similar al ADN circular puede medirse con mayor precisión después de haber sido linealizado mediante digestión de restricción.
El daño al ADN debido al aumento de la reticulación también reducirá la migración electroforética del ADN de forma dependiente de la dosis.
Concentración de bromuro de etidio
El ADN circular se ve más afectado por la concentración de bromuro de etidio que el ADN lineal si el bromuro de etidio está presente en el gel durante la electroforesis. Todos los círculos de ADN naturales están enrollados por debajo, pero el bromuro de etidio, que se intercala en el ADN circular, puede cambiar la carga, la longitud y la superhelicidad de la molécula de ADN, por lo que su presencia durante la electroforesis puede afectar su movimiento en el gel. El aumento de bromuro de etidio intercalado en el ADN puede cambiarlo de una molécula superenrollada negativamente a una forma completamente relajada, y luego a una superhélice enrollada positivamente en la intercalación máxima. La electroforesis en gel de agarosa se puede utilizar para resolver el ADN circular con una topología de superenrollamiento diferente.
Concentración de gel
La concentración del gel determina el tamaño de poro del gel que afecta la migración del ADN. La resolución del ADN cambia con el porcentaje de concentración del gel. El aumento de la concentración de agarosa de un gel reduce la velocidad de migración y mejora la separación de las moléculas de ADN más pequeñas, mientras que la reducción de la concentración del gel permite separar las moléculas de ADN grandes. Para una electroforesis en gel de agarosa estándar, una concentración de gel al 0,7 % brinda una buena separación o resolución de fragmentos grandes de ADN de 5 a 10 kb, mientras que una concentración de gel al 2 % brinda una buena resolución para fragmentos pequeños de 0,2 a 1 kb. Se puede usar una concentración de gel de hasta el 3 % para separar fragmentos muy pequeños, pero un gel de poliacrilamida vertical sería más apropiado para resolver fragmentos pequeños. Sin embargo, las altas concentraciones de gel requieren tiempos de ejecución más prolongados (a veces días) y los geles de alto porcentaje a menudo son quebradizos y es posible que no fragüen uniformemente. Los geles de agarosa de alto porcentaje deben procesarse con PFGE o FIGE. Los geles de bajo porcentaje (0,1-0,2 %) son frágiles y pueden romperse. Los geles al 1% son comunes para muchas aplicaciones.
Campo aplicado
A voltajes bajos, la tasa de migración del ADN es proporcional al voltaje aplicado, es decir, cuanto mayor sea el voltaje, más rápido se mueve el ADN. Sin embargo, al aumentar la fuerza del campo eléctrico, la movilidad de los fragmentos de ADN de alto peso molecular aumenta diferencialmente y el rango efectivo de separación disminuye y, por lo tanto, la resolución es menor a alto voltaje. Para una resolución óptima de ADN de más de 2 kb de tamaño en electroforesis en gel estándar, se recomienda de 5 a 8 V/cm. El voltaje también está limitado por el hecho de que calienta el gel y puede hacer que el gel se derrita si se ejecuta un gel a alto voltaje durante un período prolongado, particularmente para gel de agarosa de bajo punto de fusión.
Sin embargo, la movilidad del ADN puede cambiar en un campo inestable. En un campo que se invierte periódicamente, la movilidad del ADN de un tamaño particular puede disminuir significativamente en una frecuencia de ciclo particular. Este fenómeno puede dar lugar a una inversión de bandas en la que los fragmentos de ADN más grandes se mueven más rápido que los más pequeños en PFGE.
Mecanismo de migración y separación
La carga negativa de su columna vertebral de fosfato mueve el ADN hacia el ánodo cargado positivamente durante la electroforesis. Sin embargo, la migración de moléculas de ADN en solución, en ausencia de matriz de gel, es independiente del peso molecular durante la electroforesis, es decir, no hay separación por tamaño sin matriz de gel. La interacción hidrodinámica entre diferentes partes del ADN se interrumpe mediante la transmisión de contraiones que se mueven en la dirección opuesta, por lo que no existe ningún mecanismo para generar una dependencia de la velocidad con la longitud en una escala mayor que la longitud de detección de aproximadamente 10 nm. Esto lo hace diferente de otros procesos como la sedimentación o la difusión donde la interacción hidrodinámica de largo alcance es importante.
Por lo tanto, la matriz de gel es responsable de la separación del ADN por tamaño durante la electroforesis, sin embargo, el mecanismo preciso responsable de la separación no está del todo claro. Existe una serie de modelos para el mecanismo de separación de biomoléculas en matriz de gel, uno ampliamente aceptado es el modelo de Ogston que trata la matriz de polímero como un tamiz que consta de una red distribuida aleatoriamente de poros interconectados. Una proteína globular o un ADN en espiral aleatorio se mueve a través de los poros conectados lo suficientemente grandes como para acomodar su paso, y es más probable que el movimiento de moléculas más grandes se vea obstaculizado y ralentizado por colisiones con la matriz de gel, y por lo tanto las moléculas de diferentes tamaños pueden separarse en este proceso de tamizado.
Sin embargo, el modelo de Ogston se descompone para moléculas grandes en las que los poros son significativamente más pequeños que el tamaño de la molécula. Para moléculas de ADN de tamaño superior a 1 kb, se usa más comúnmente un modelo de reptación (o sus variantes). Este modelo asume que el ADN puede arrastrarse en forma de "serpiente" forma (de ahí "reptación") a través de los poros como una molécula alargada. A mayor intensidad de campo eléctrico, esto se convirtió en un modelo de repetición sesgado, en el que el extremo delantero de la molécula se sesga fuertemente en la dirección de avance, y este borde delantero tira del resto de la molécula. En el modo completamente sesgado, la movilidad alcanzó un punto de saturación y el ADN más allá de cierto tamaño no se puede separar. Sin embargo, en la práctica no se observa una alineación paralela perfecta de la cadena con el campo, ya que eso significaría la misma movilidad para moléculas largas y cortas. El refinamiento adicional del modelo de reptación sesgada tiene en cuenta las fluctuaciones internas de la cadena.
El modelo de reptación sesgada también se ha utilizado para explicar la movilidad del ADN en PFGE. La orientación del ADN se construye progresivamente por repetición después del inicio de un campo, y el tiempo en que alcanza la velocidad de estado estable depende del tamaño de la molécula. Cuando se cambia el campo, las moléculas más grandes tardan más en reorientarse, por lo que es posible discriminar entre las cadenas largas que no pueden alcanzar su velocidad constante de las cortas que viajan la mayor parte del tiempo a velocidad constante. Sin embargo, también existen otros modelos.
La microscopía de fluorescencia en tiempo real de las moléculas teñidas mostró una dinámica más sutil durante la electroforesis, con el ADN mostrando una elasticidad considerable a medida que se estiraba alternativamente en la dirección del campo aplicado y luego se contraía en una bola o se enganchaba en forma de U. cuando queda atrapado en las fibras de polímero. Esta observación puede denominarse la "oruga" modelo. Otro modelo propone que el ADN se enreda con la matriz polimérica, y cuanto más grande es la molécula, más probable es que se enrede y se impida su movimiento.
Visualización
El tinte más común que se usa para hacer visibles las bandas de ADN o ARN para la electroforesis en gel de agarosa es el bromuro de etidio, generalmente abreviado como EtBr. Fluoresce bajo la luz ultravioleta cuando se intercala en el surco principal del ADN (o ARN). Al pasar el ADN a través de un gel tratado con EtBr y visualizarlo con luz ultravioleta, cualquier banda que contenga más de ~20 ng de ADN se vuelve claramente visible. EtBr es un mutágeno conocido y existen alternativas más seguras, como GelRed, producido por Biotium, que se une al surco menor.
SYBR Green I es otra tinción de dsDNA, producida por Invitrogen. Es más caro, pero 25 veces más sensible y posiblemente más seguro que el EtBr, aunque no hay datos que aborden su mutagenicidad o toxicidad en humanos.
SYBR Safe es una variante de SYBR Green que ha demostrado tener niveles lo suficientemente bajos de mutagenicidad y toxicidad para ser considerado un desecho no peligroso según las regulaciones federales de EE. UU. Tiene niveles de sensibilidad similares a EtBr, pero, como SYBR Green, es significativamente más caro. Sin embargo, en países donde la eliminación segura de desechos peligrosos es obligatoria, los costos de eliminación de EtBr pueden superar fácilmente la diferencia de precio inicial.
Dado que el ADN teñido con EtBr no es visible a la luz natural, los científicos mezclan el ADN con tampones de carga cargados negativamente antes de agregar la mezcla al gel. Los tampones de carga son útiles porque son visibles con luz natural (a diferencia de la luz ultravioleta para el ADN teñido con EtBr) y sedimentan conjuntamente con el ADN (lo que significa que se mueven a la misma velocidad que el ADN de cierta longitud). El cianol de xileno y el azul de bromofenol son tintes comunes que se encuentran en los tampones de carga; corren aproximadamente a la misma velocidad que los fragmentos de ADN que tienen una longitud de 5000 pb y 300 pb respectivamente, pero la posición precisa varía con el porcentaje del gel. Otros marcadores de progreso que se usan con menos frecuencia son Cresol Red y Orange G, que funcionan a aproximadamente 125 pb y 50 pb, respectivamente.
La visualización también se puede lograr mediante la transferencia de ADN después de SDS-PAGE a una membrana de nitrocelulosa seguida de exposición a una sonda de hibridación. Este proceso se denomina transferencia de Southern.
Para los tintes fluorescentes, después de la electroforesis, el gel se ilumina con una lámpara ultravioleta (generalmente colocándolo en una caja de luz, mientras se usa equipo de protección para limitar la exposición a la radiación ultravioleta). El aparato iluminador en su mayoría también contiene un aparato de formación de imágenes que toma una imagen del gel, después de la iluminación con radiación UV. El bromuro de etidio emite una fluorescencia de color naranja rojizo en presencia de ADN, ya que se ha intercalado con el ADN. La banda de ADN también se puede cortar del gel y luego se puede disolver para recuperar el ADN purificado. A continuación, el gel se puede fotografiar normalmente con una cámara digital o polaroid. Aunque el ácido nucleico teñido emite una fluorescencia de color naranja rojizo, las imágenes suelen mostrarse en blanco y negro (véanse las figuras). El daño UV a la muestra de ADN puede reducir la eficiencia de la manipulación posterior de la muestra, como la ligadura y la clonación. Las radiaciones UV de longitud de onda más corta (302 o 312 nm) causan un mayor daño, por ejemplo, la exposición durante tan solo 45 segundos puede reducir significativamente la eficiencia de transformación. Por lo tanto, si el ADN se va a utilizar para procedimientos posteriores, se debe limitar la exposición a radiaciones UV de longitud de onda más corta; en su lugar, se debe utilizar radiación UV de longitud de onda más alta (365 nm) que causa menos daño. Sin embargo, las radiaciones de mayor longitud de onda producen una fluorescencia más débil, por lo tanto, si es necesario capturar la imagen del gel, se puede utilizar una luz ultravioleta de menor longitud de onda durante un tiempo breve. La adición de citidina o guanosina al tampón de electroforesis a una concentración de 1 mM puede proteger el ADN del daño. Como alternativa, se puede utilizar una fuente de excitación de luz azul con una mancha azul excitable como SYBR Green o GelGreen.
La investigación sobre electroforesis en gel suele aprovechar las herramientas de análisis de imágenes basadas en software, como ImageJ.
| 1 | 2 | 3 |
|---|---|---|
Contenido relacionado
Cola (desambiguación)
Laboratorio de tierra
Columna vertebral





