DMSO reductasa
La DMSO reductasa es una enzima que contiene molibdeno y que cataliza la reducción del dimetilsulfóxido (DMSO) a sulfuro de dimetilo (DMS). Esta enzima actúa como reductasa terminal en condiciones anaeróbicas en algunas bacterias, siendo el DMSO el aceptor terminal de electrones. Durante el transcurso de la reacción, el átomo de oxígeno del DMSO se transfiere al molibdeno y luego se reduce a agua.
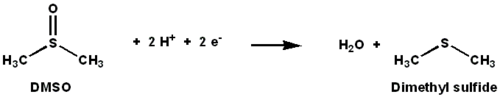
La DMSO reductasa (DMSOR) y otros miembros de la familia de la DMSO reductasa son exclusivos de las bacterias y las arqueas. Las enzimas de esta familia participan en la fosforilación oxidativa anaeróbica y la respiración litotrófica basada en donantes inorgánicos. Estas enzimas han sido diseñadas para degradar oxoaniones. La DMSOR cataliza la transferencia de dos electrones y un átomo de oxígeno en la reacción: el sitio activo de la DMSOR contiene molibdeno, que es poco común en biología.
Estructura terciaria y sitio activo
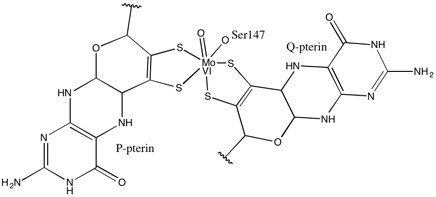
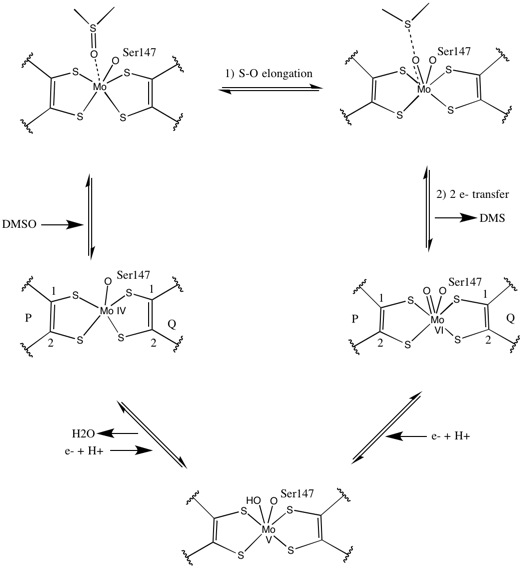
Al igual que otros miembros de la familia de la DMSO reductasa, la estructura terciaria de la DMSOR está compuesta por dominios I-IV que rodean a Mo, con el dominio IV interactuando fuertemente con los cofactores Mo de piranopterinditioleno (P- y Q-pterina) del sitio activo. Los miembros de la familia de la DMSO reductasa difieren en términos de sus sitios activos. En el caso de la DMSOR, el centro Mo se encuentra en dos ditiolenos proporcionados por dos cofactores de piranopterina. Estos cofactores orgánicos, llamados molibdopterinas, están unidos a GMP para crear una forma de dinucleótido. Un quinto ligando adicional similar a una tapa es la cadena lateral O del residuo de serina-147, lo que clasifica aún más a la enzima como DMSO reductasa de tipo III. En los tipos I y II, la serina se reemplaza por residuos de cisteína y aspartato, respectivamente. Dependiendo del estado redox del Mo, que fluctúa entre IV, V o VI a medida que avanza la reacción, el núcleo de Mo del sitio activo también puede estar ligado a un átomo de oxígeno de un grupo aqua-, hidroxi- u oxo-, respectivamente. Los estudios han demostrado que la identidad particular del aminoácido utilizado para coordinar el núcleo de Mo influye en gran medida en el potencial de punto medio redox del Mo y el estado de protonación de la ligadura del grupo oxígeno, que son determinantes clave en el mecanismo de catálisis de la enzima.
Mecanismo
Los estudios isotópicos iniciales del DMSO18 establecieron un mecanismo de doble oxotransferasa para la DMSOR de R. sphaeroides. En este mecanismo, el O18 marcado se transfiere del sustrato a Mo, que luego transfiere el O18 al 1,3,5-triaza-7-fosfaadamantano (PTA) para producir PTAO18. En un mecanismo análogo, el DMSO transfiere O a Mo y el centro Mo(VI)O resultante se reduce, produciendo agua.
Los estudios sobre complejos sintéticos de bis-ditioleno de Mo sugieren que se produce transferencia de oxígeno y transferencia de electrones. Mediante el uso de XAS y DFT de borde K de S, estos estudios de modelos apuntan a una escisión S-O concertada y transferencia de electrones. Las tasas son proporcionales a la disminución de la fuerza del enlace X-O del sustrato y al aumento de la afinidad protónica del sustrato.
La cristalografía de rayos X estableció que la estructura terciaria general de la enzima permanece constante a lo largo de la progresión de la reacción. Sin embargo, varios experimentos diferentes realizados en DMSOR de R. sphaeroides informaron diferentes resultados para la actividad de coordinación de los cuatro ligandos de ditioleno potenciales. Mientras que una investigación de cristalografía de rayos X concluyó una coordinación equidistante de los cuatro ligandos Mo-S en la forma oxidada, que está respaldada por numerosos estudios de espectroscopia de absorción de rayos X (XAS), un estudio diferente caracterizó distancias asimétricas de Mo-S. Ambos estudios, así como los estudios de resonancia paramagnética electrónica (EPR), han predicho que el sitio activo de Mo es altamente flexible en términos de posición y grado de coordinación de ligandos potenciales.
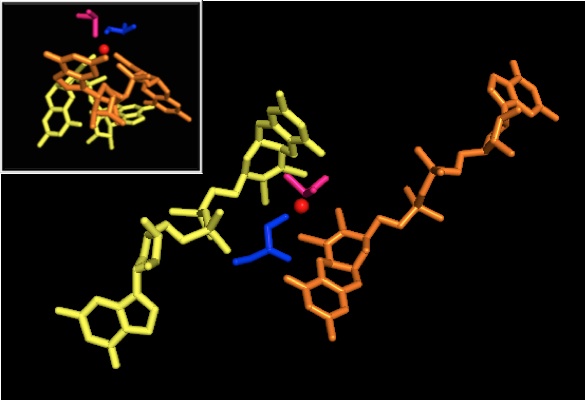
Los datos que sugerían dos cofactores de piranopterina significativamente asimétricos se utilizaron para proponer un mecanismo de reacción. En la forma Mo VI completamente oxidada del sitio activo, el grupo oxo y los ligandos de serina estaban coordinados a distancias de 1,7 A del centro de Mo. S1 y S2 de la pterina P y S1 de la pterina Q estaban ubicados a 2,4 A de distancia de Mo, y S2 de la pterina Q estaba ubicado a 3,1 A de distancia. Esta asimetría de la pterina puede ser el resultado del efecto trans del grupo oxo que debilita el enlace S2-Mo, que está ubicado directamente opuesto al grupo oxo.
En cambio, la estructura de la forma Mo IV totalmente reducida del sitio activo mostró que la P-pterina S1 y S2 y la Q-pterina S1 mantenían una coordinación completa, sin embargo, la S2 de la Q-pterina se alejó del centro metálico, lo que indica una coordinación reducida. Este cambio en la longitud del enlace ligando-Mo es consistente con el mecanismo propuesto de transferencia directa de oxígeno desde el sustrato DMSO al Mo. Una coordinación más débil del ditioleno en la forma enzimática reducida podría facilitar la unión directa del S=O. En la reducción de Mo y la protonación del grupo oxo, se propone que una fuente de electrones del citocromo podría unirse a una depresión por encima del sitio activo y reducir directamente el centro de Mo, o alternativamente, este citocromo podría unirse a un bucle polipeptídico bien solvatado en proximidad a la Q-pterina, y la Q-pterina podría mediar esta transferencia de electrones.
Ubicación y regulación celular
En R. sphaeroides, DMSOR es una proteína soluble en agua de una sola subunidad que no requiere cofactores adicionales más allá de la pterina. En E. coli, DMSOR está incrustado dentro de la membrana y tiene tres subunidades únicas, una de las cuales incluye el cofactor característico pterina, otra que contiene cuatro grupos 4Fe:4S y una subunidad transmembrana final que se une y oxida el menaquinol. La transferencia de un e- desde el menaquinol a los grupos 4Fe:4S y finalmente al sitio activo de pterina-Mo genera un gradiente de protones utilizado para la generación de ATP.
El DMSOR se regula predominantemente a nivel transcripcional. Está codificado por el gen dor y se expresa cuando se activa mediante una cascada de señales, que está bajo la regulación de las proteínas DorS, DorR y DorC. Un estudio de fusiones de lacZ (genes reporteros) con los promotores dorS, dorR y dorC correspondientes concluyó que la expresión de DorR y DorC aumentó en entornos con oxígeno reducido, pero la expresión de DorS no se vio afectada por la concentración de oxígeno. La expresión de DorC también aumentó con concentraciones crecientes de DMSO.
Impacto ambiental
El DMS, un producto de DMSOR, es un componente del ciclo del azufre. El DMS se oxida a metanosulfonatos, que forman núcleos de condensación de nubes sobre océanos abiertos, donde no existe la fuente alternativa de nucleación, el polvo. La formación de nubes es un componente clave para aumentar el albedo de la Tierra y regular la temperatura atmosférica, por lo que esta enzima y la reacción que cataliza podrían resultar útiles en la frontera del control climático.
Referencias
- ^ Kappler U, Schäfer H (2014). "Capítulo 11. Transformaciones de Dimethylsulfide". En Kroneck PM, Torres ME (eds.). La Biogeoquímica Metal-Driven de Complejos Gaseosos en el Medio Ambiente. Hiones de metal en Ciencias de la Vida. Vol. 14. Springer. pp. 279–313. doi:10.1007/978-94-017-9269-1_11. ISBN 978-94-017-9268-4. PMID 25416398.
- ^ a b c d McEwan AG, Kappler U (2004). "La familia de la reductasa DMSO de las enzimas del molibdeno microbiano" (PDF). Australian Biochemist. 35 (3): 17-20. Archivado desde el original (PDF) el 2014-03-07. Retrieved 2014-02-27.
- ^ a b c d e McEwan AG, Ridge JP, McDevitt CA, Hugenholtz P (2002). "La Familia de Reductasa DMSO de Enzimas de Molibdeno Microbial; Propiedades Moleculares y Papel en la Reducción Disimilar de Elementos Tóxicos". Geomicrobiology Journal. 19 (1): 3–21. Bibcode:2002GmbJ...19....3M. doi:10.1080/014904502317246138. S2CID 85091949.
- ^ PDB: 1DMS; Schneider F, Löwe J, Huber R, Schindelin H, Kisker C, Knäblein J (octubre de 1996). "Crystal structure of dimethyl sulfoxide reductase from Rhodobacter capsulatus at 1.88 A resolution". Journal of Molecular Biology. 263 (1): 53–69. doi:10.1006/jmbi.1996.0555. PMID 8890912.
- ^ PDB: 4DMR; McAlpine AS, McEwan AG, Bailey S (enero de 1998). "La estructura de cristal de alta resolución de la reductasa DMSO en complejo con DMSO". Journal of Molecular Biology. 275 (4): 613–23. doi:10.1006/jmbi.1997.1513. PMID 9466935.
- ^ Schultz BE, Hille R, Holm RH (1995), "Transferencia de oxígeno directo en el mecanismo de acción de Rhodobacter sphaeroides dimethyl sulfoxide reductase", Journal of the American Chemical Society, 117 (2): 827–828, doi:10.1021/ja00107a031, ISSN 0002-7863
- ^ a b c d e Kisker C, Schindelin H, Rees DC (1997). "Enzimas que contienen molibdeno-cofactor: estructura y mecanismo" (PDF). Examen anual de la bioquímica. 66: 233–67. doi:10.1146/annurev.biochem.66.1.233. PMID 9242907.
- ^ Tenderholt AL, Wang JJ, Szilagyi RK, Holm RH, Hodgson KO, Hedman B, Solomon EI (junio de 2010). "Sulfur K-edge Extroscopia de absorción de rayos X y cálculos funcionales de densidad en Mo(IV) y Mo(VI)=O bis-dithiolenes: información sobre el mecanismo de transferencia de oxo en DMSO reductasa y análogos funcionales relacionados". Journal of the American Chemical Society. 132 (24): 8359–71. doi:10.1021/ja910369c. PMC 2907113. PMID 20499905.
- ^ McAlpine AS, McEwan AG, Shaw AL, Bailey S (1997). "Molybdenum active centre of DMSO reductase from Rhodobacter capsulatus: Crystal structure of the oxidised enzima at 1.82- Una resolución y la enzima ditionita reducida a 2.8-A resolución". Journal of Biological Inorganic Chemistry. 2 (6): 690–701. doi:10.1007/s007750050185. S2CID 23027986.
- ^ Gunsalus RP (noviembre de 1992). "Control del flujo de electrones en Escherichia coli: transcripción coordinada de genes de vías respiratorias". Journal of Bacteriology. 174 (22): 7069-74. doi:10.1128/jb.174.22.7069-7074.1992. PMC 207394. PMID 1331024.
- ^ Sarkar B (21 de marzo de 2002). Heavy Metals In The Environment. CRC Press. p. 456. ISBN 978-0-8247-4475-5.