Dedo de cinc
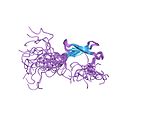
Un dedo de zinc es un pequeño motivo estructural de proteína que se caracteriza por la coordinación de uno o más iones de zinc (Zn2+) para estabilizar el pliegue. Se acuñó originalmente para describir la apariencia similar a un dedo de una estructura hipotética del factor de transcripción IIIA de la rana con garras africana (Xenopus laevis). Sin embargo, se ha encontrado que abarca una amplia variedad de estructuras proteicas diferentes en células eucariotas. Originalmente se demostró que Xenopus laevis TFIIIA contiene zinc y requiere el metal para funcionar en 1983, el primer requerimiento de zinc informado para una proteína reguladora de genes seguido poco después por el factor Krüppel en Drosophila. A menudo aparece como un dominio de unión a metales en proteínas multidominio.
Las proteínas que contienen dedos de zinc (proteínas con dedos de zinc) se clasifican en varias familias estructurales diferentes. A diferencia de muchas otras estructuras supersecundarias claramente definidas, como las llaves griegas o las horquillas β, existen varios tipos de dedos de zinc, cada uno con una arquitectura tridimensional única. Una clase particular de proteína con dedos de zinc está determinada por esta estructura tridimensional, pero también se puede reconocer en función de la estructura primaria de la proteína o la identidad de los ligandos que coordinan el ion de zinc. Sin embargo, a pesar de la gran variedad de estas proteínas, la gran mayoría suele funcionar como módulos de interacción que se unen a ADN, ARN, proteínas u otras moléculas pequeñas y útiles, y las variaciones en la estructura sirven principalmente para alterar la especificidad de unión de una proteína en particular..
Desde su descubrimiento original y la elucidación de su estructura, estos módulos de interacción han demostrado ser ubicuos en el mundo biológico y pueden encontrarse en el 3 % de los genes del genoma humano. Además, los dedos de zinc se han vuelto extremadamente útiles en varias capacidades terapéuticas y de investigación. La ingeniería de dedos de zinc para que tengan afinidad por una secuencia específica es un área de investigación activa, y las nucleasas de dedos de zinc y los factores de transcripción de dedos de zinc son dos de las aplicaciones más importantes que se han realizado hasta la fecha.
Historia
Los dedos de zinc se identificaron por primera vez en un estudio de transcripción en la rana con garras africana, Xenopus laevis en el laboratorio de Aaron Klug. Un estudio de la transcripción de una secuencia particular de ARN reveló que la fuerza de unión de un factor de transcripción pequeño (factor de transcripción IIIA; TFIIIA) se debía a la presencia de estructuras similares a dedos que coordinan zinc. La secuenciación de aminoácidos de TFIIIA reveló nueve secuencias en tándem de 30 aminoácidos, incluidos dos pares invariantes de residuos de cisteína e histidina. La estructura fina de absorción de rayos X extendida confirmó la identidad de los ligandos de zinc: dos cisteínas y dos histidinas. Se pensaba que el bucle de unión al ADN formado por la coordinación de estos ligandos por el zinc se parecía a los dedos, de ahí el nombre. Esto fue seguido poco después por el descubrimiento del factor Krüppel en Drosophila por el equipo de Schuh en 1986. Trabajos más recientes en la caracterización de proteínas en varios organismos han revelado la importancia de los iones de zinc en la estabilización de polipéptidos.
Las estructuras cristalinas de los complejos de dedos de zinc-ADN resueltos en 1991 y 1993 revelaron el patrón canónico de interacciones de los dedos de zinc con el ADN. Se encuentra que la unión del dedo de zinc es distinta de muchas otras proteínas de unión al ADN que se unen al ADN a través de la doble simetría de la doble hélice, en cambio, los dedos de zinc se unen linealmente en tándem para unir secuencias de ácido nucleico de longitud variable. Los dedos de zinc a menudo se unen a una secuencia de ADN conocida como caja GC. La naturaleza modular del motivo de dedos de zinc permite unir una gran cantidad de combinaciones de secuencias de ADN y ARN con un alto grado de afinidad y especificidad y, por lo tanto, es ideal para la ingeniería de proteínas que pueden dirigirse y unirse a secuencias de ADN específicas. En 1994, se demostró que una proteína de tres dedos construida artificialmente puede bloquear la expresión de un oncogén en una línea celular de ratón. Desde entonces, se han construido dedos de zinc fusionados con varios otros dominios efectores, algunos con importancia terapéutica.
Dominio
Los dominios de dedos de zinc (Znf) son motivos proteicos relativamente pequeños que contienen múltiples protuberancias similares a dedos que hacen contactos en tándem con su molécula objetivo. Algunos de estos dominios se unen al zinc, pero muchos no lo hacen, sino que se unen a otros metales como el hierro oa ningún metal. Por ejemplo, algunos miembros de la familia forman puentes de sal para estabilizar los pliegues en forma de dedos. Se identificaron por primera vez como un motivo de unión al ADN en el factor de transcripción TFIIIA de Xenopus laevis (rana con garras africana), sin embargo, ahora se reconoce que se unen a sustratos de ADN, ARN, proteínas y/o lípidos. Sus propiedades de unión dependen de la secuencia de aminoácidos de los dominios de los dedos y del conector entre los dedos, así como de las estructuras de orden superior y del número de dedos. Los dominios Znf a menudo se encuentran en grupos, donde los dedos pueden tener diferentes especificidades de unión. Los motivos Znf ocurren en varias superfamilias de proteínas no relacionadas, que varían tanto en secuencia como en estructura. Muestran una versatilidad considerable en los modos de unión, incluso entre miembros de la misma clase (p. ej., algunos se unen a ADN, otros a proteínas), lo que sugiere que los motivos Znf son andamios estables que han desarrollado funciones especializadas. Por ejemplo, las proteínas que contienen Znf funcionan en la transcripción de genes, la traducción, el tráfico de ARNm, la organización del citoesqueleto, el desarrollo epitelial, la adhesión celular, el plegamiento de proteínas, la remodelación de la cromatina y la detección de zinc, por nombrar solo algunos. Los motivos de unión a zinc son estructuras estables y rara vez experimentan cambios conformacionales al unirse a su objetivo.
Clases
Inicialmente, el término dedo de zinc se utilizó únicamente para describir el motivo de unión al ADN que se encuentra en Xenopus laevis; sin embargo, ahora se usa para referirse a cualquier cantidad de estructuras relacionadas por su coordinación de un ion de zinc. En general, los dedos de zinc coordinan los iones de zinc con una combinación de residuos de cisteína e histidina. Originalmente, el número y orden de estos residuos se utilizó para clasificar diferentes tipos de dedos de zinc (por ejemplo, Cys2His2, Cys4, y Cys6). Más recientemente, se ha utilizado un método más sistemático para clasificar las proteínas con dedos de zinc. Este método clasifica las proteínas con dedos de zinc en "grupos de pliegues" basado en la forma general del esqueleto de la proteína en el dominio plegado. Los "grupos de pliegues" más comunes de dedos de zinc son el Cys2His2-like (el "dedo de zinc clásico"), la clave de sol y la cinta de zinc.
La siguiente tabla muestra las diferentes estructuras y sus características clave:
Cys2His2
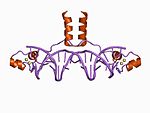
El grupo de pliegues similar a Cys2His2 (C2H2) es, con mucho, la clase de dedos de zinc mejor caracterizada y es común en los factores de transcripción de mamíferos. Dichos dominios adoptan un pliegue ββα simple y tienen el motivo de secuencia de aminoácidos:
- X2-Cys-X2,4-Cys-X12- Hola-X3,4,5- Hola.
Esta clase de dedos de zinc puede tener una variedad de funciones, como unirse al ARN y mediar en las interacciones proteína-proteína, pero es más conocido por su papel en las proteínas de unión al ADN de secuencia específica, como Zif268 (Egr1). En tales proteínas, los dominios de dedos de zinc individuales típicamente ocurren como repeticiones en tándem con dos, tres o más dedos que comprenden el dominio de unión al ADN de la proteína. Estas matrices en tándem pueden unirse en el surco principal del ADN y, por lo general, están espaciadas a intervalos de 3 pb. La hélice α de cada dominio (a menudo denominada "hélice de reconocimiento") puede establecer contactos específicos de secuencia con las bases de ADN; los residuos de una sola hélice de reconocimiento pueden ponerse en contacto con cuatro o más bases para producir un patrón superpuesto de contactos con dedos de zinc adyacentes.
Nudillo de mordaza
Este grupo de pliegues está definido por dos hebras β cortas conectadas por un giro (nudillo de zinc) seguido de una hélice o bucle corto y se parece al clásico Cys2His2 motivo con gran parte de la hélice y horquilla β truncada.
La proteína de la nucleocápside retroviral (NC) del VIH y otros retrovirus relacionados son ejemplos de proteínas que poseen estos motivos. El dedo de zinc del nudillo de la mordaza en la proteína NC del VIH es el objetivo de una clase de medicamentos conocidos como inhibidores del dedo de zinc.
Clave de sol
El motivo de la clave de sol consta de una horquilla β en el extremo N y una hélice α en el extremo C, cada una de las cuales aporta dos ligandos para la unión del zinc, aunque un bucle y una segunda horquilla β de longitud variable y la conformación puede estar presente entre la horquilla β N-terminal y la hélice α C-terminal. Estos dedos están presentes en un grupo diverso de proteínas que frecuentemente no comparten secuencia o similitud funcional entre sí. Las proteínas mejor caracterizadas que contienen dedos de zinc de clave de sol son los receptores de hormonas nucleares.
Cinta de zinc
El pliegue de cinta de zinc se caracteriza por dos horquillas beta que forman dos subsitios de unión de zinc estructuralmente similares.
Zn2/Cys6
Los miembros canónicos de esta clase contienen un grupo de zinc binuclear en el que dos iones de zinc están unidos por seis residuos de cisteína. Estos dedos de zinc se pueden encontrar en varios factores de transcripción, incluida la proteína de levadura Gal4.
Varios
La proteína antiviral con dedos de zinc (ZAP) se une al sitio CpG. Se utiliza en mamíferos para la defensa antiviral.
Aplicaciones
Se pueden usar varias técnicas de ingeniería de proteínas para alterar la especificidad de unión al ADN de los dedos de zinc y se pueden usar repeticiones en tándem de dichos dedos de zinc modificados para apuntar a las secuencias de ADN genómico deseadas. La fusión de un segundo dominio de proteína, como un activador o represor transcripcional, a una matriz de dedos de zinc diseñados que se unen cerca del promotor de un gen dado puede usarse para alterar la transcripción de ese gen. Las fusiones entre matrices de dedos de zinc diseñadas y dominios de proteínas que escinden o modifican el ADN también se pueden usar para dirigir esas actividades a los loci genómicos deseados. Las aplicaciones más comunes para las matrices de dedos de zinc diseñadas incluyen factores de transcripción de dedos de zinc y nucleasas de dedos de zinc, pero también se han descrito otras aplicaciones. Las matrices típicas de dedos de zinc diseñadas tienen entre 3 y 6 motivos de dedos de zinc individuales y se unen a sitios objetivo que van desde 9 pares de bases hasta 18 pares de bases de longitud. Las matrices con motivos de 6 dedos de zinc son particularmente atractivas porque se unen a un sitio objetivo que es lo suficientemente largo como para tener una buena posibilidad de ser único en un genoma de mamífero.
Nucleasas con dedos de zinc
Las matrices de dedos de zinc diseñadas a menudo se fusionan con un dominio de división de ADN (por lo general, el dominio de división de FokI) para generar nucleasas de dedos de zinc. Tales fusiones de dedo de zinc-FokI se han convertido en reactivos útiles para manipular genomas de muchos organismos superiores, incluidos Drosophila melanogaster, Caenorhabditis elegans, tabaco, maíz, pez cebra, varios tipos de células de mamíferos, y ratas Dirigir una ruptura de doble cadena a un locus genómico deseado puede usarse para introducir mutaciones de cambio de marco en la secuencia de codificación de un gen debido a la naturaleza propensa a errores de la vía de reparación del ADN no homólogo. Si una "secuencia donante" de ADN homóloga también se utiliza, entonces el locus genómico se puede convertir en una secuencia definida a través de la ruta de reparación dirigida por homología. Un ensayo clínico en curso está evaluando las nucleasas con dedos de zinc que interrumpen el gen CCR5 en las células T humanas CD4+ como un posible tratamiento para el VIH/SIDA.
Métodos de ingeniería de conjuntos de dedos de zinc
La mayoría de las matrices de dedos de zinc diseñadas se basan en el dominio de dedos de zinc del factor de transcripción murino Zif268, aunque algunos grupos han utilizado matrices de dedos de zinc basadas en el factor de transcripción humano SP1. Zif268 tiene tres motivos de dedos de zinc individuales que se unen colectivamente a una secuencia de 9 pb con alta afinidad. La estructura de esta proteína unida al ADN se resolvió en 1991 y estimuló una gran cantidad de investigación sobre matrices de dedos de zinc diseñadas. En 1994 y 1995, varios grupos utilizaron la presentación de fagos para alterar la especificidad de un solo dedo de zinc de Zif268. Hay dos métodos principales que se utilizan actualmente para generar matrices de dedos de zinc diseñadas, ensamblaje modular y un sistema de selección bacteriana, y existe cierto debate sobre qué método es el más adecuado para la mayoría de las aplicaciones.
El método más directo para generar nuevas matrices de dedos de zinc es combinar "módulos" de dedos de zinc más pequeños; de especificidad conocida. La estructura de la proteína con dedos de zinc Zif268 unida al ADN descrita por Pavletich y Pabo en su publicación de 1991 ha sido clave para gran parte de este trabajo y describe el concepto de obtener dedos para cada uno de los 64 posibles tripletes de pares de bases y luego mezclarlos y combinarlos. dedos para diseñar proteínas con cualquier especificidad de secuencia deseada. El proceso de ensamblaje modular más común implica la combinación de dedos de zinc separados, cada uno de los cuales puede reconocer una secuencia de ADN de 3 pares de bases para generar matrices de 3, 4, 5 o 6 dedos que reconocen sitios objetivo que van desde 9 pares de bases hasta 18 pares de bases en longitud. Otro método utiliza módulos de 2 dedos para generar matrices de dedos de zinc con hasta seis dedos de zinc individuales. El Laboratorio Barbas del Instituto de Investigación Scripps utilizó la visualización de fagos para desarrollar y caracterizar dominios de dedos de zinc que reconocen la mayoría de las secuencias de tripletes de ADN, mientras que otro grupo aisló y caracterizó dedos individuales del genoma humano. Un inconveniente potencial con el ensamblaje modular en general es que las especificidades de los dedos de zinc individuales pueden superponerse y pueden depender del contexto de los dedos de zinc y el ADN circundantes. Un estudio reciente demostró que una alta proporción de matrices de dedos de zinc de 3 dedos generadas por ensamblaje modular no logran unirse a su objetivo deseado con suficiente afinidad en un ensayo bacteriano de dos híbridos y no funcionan como nucleasas de dedos de zinc, pero la tasa de éxito fue algo mayor cuando se orientaron sitios del tipo GNNGNNGNN.
Un estudio posterior utilizó ensamblaje modular para generar nucleasas con dedos de zinc con matrices de 3 y 4 dedos y observó una tasa de éxito mucho mayor con matrices de 4 dedos. También se ha informado de una variante de ensamblaje modular que tiene en cuenta el contexto de los dedos vecinos y este método tiende a producir proteínas con un rendimiento mejorado en relación con el ensamblaje modular estándar.
Se han utilizado numerosos métodos de selección para generar matrices de dedos de zinc capaces de identificar las secuencias deseadas. Los esfuerzos de selección iniciales utilizaron la visualización de fagos para seleccionar proteínas que se unían a un objetivo de ADN determinado de un gran grupo de matrices de dedos de zinc parcialmente aleatorias. Esta técnica es difícil de usar en más de un solo dedo de zinc a la vez, por lo que se desarrolló un proceso de varios pasos que generó una matriz de 3 dedos completamente optimizada agregando y optimizando un solo dedo de zinc a la vez. Esfuerzos más recientes han utilizado sistemas de un híbrido de levadura, sistemas de un híbrido y dos híbridos bacterianos, y células de mamífero. Un método nuevo y prometedor para seleccionar matrices novedosas de dedos de zinc de 3 dedos utiliza un sistema bacteriano de dos híbridos y se ha denominado 'ABIERTO'. por sus creadores. Este sistema combina grupos preseleccionados de dedos de zinc individuales que se seleccionaron para unirse a un triplete determinado y luego utiliza una segunda ronda de selección para obtener matrices de 3 dedos capaces de unirse a una secuencia deseada de 9 pb. Este sistema fue desarrollado por el Consorcio de dedos de zinc como una alternativa a las fuentes comerciales de arreglos de dedos de zinc diseñados. Es algo difícil comparar directamente las propiedades de unión de las proteínas generadas con este método con las proteínas generadas por ensamblaje modular, ya que nunca se informaron los perfiles de especificidad de las proteínas generadas por el método OPEN.
Ejemplos
Esta entrada representa el dominio de dedo de zinc de tipo CysCysHisCys (C2HC) que se encuentra en eucariotas. Las proteínas que contienen estos dominios incluyen:
- MYST familia histone acetyltransferases
- Factor de transcripción de Myelin Myt1
- Suppressor of tumourigenicity protein 18 (ST18)
Contenido relacionado
Abedul
Redox
Concha