Crossover (algoritmo genético)
En algoritmos genéticos y computación evolutiva, el cruzamiento, también llamado recombinación, es un operador genético que se utiliza para combinar la información genética de dos padres para generar una nueva descendencia. Es una forma de generar estocásticamente nuevas soluciones a partir de una población existente y es análoga al cruce que ocurre durante la reproducción sexual en biología. También se pueden generar soluciones clonando una solución existente, lo que es análogo a la reproducción asexual. Las soluciones recién generadas pueden sufrir mutaciones antes de agregarse a la población.
Diferentes algoritmos en computación evolutiva pueden utilizar diferentes estructuras de datos para almacenar información genética, y cada representación genética puede recombinarse con diferentes operadores cruzados. Las estructuras de datos típicas que se pueden recombinar mediante cruce son matrices de bits, vectores de números reales o árboles.
La lista de operadores que se presenta a continuación no está completa y sirve principalmente como una ilustración ejemplar de este tipo de operador genético diádico. Se pueden encontrar más operadores y más detalles en la literatura.
Cruce para matrices binarias
Los algoritmos genéticos tradicionales almacenan información genética en un cromosoma representado por una matriz de bits. Los métodos de cruce para matrices de bits son populares y constituyen un ejemplo ilustrativo de recombinación genética.
Cruce de un punto
Un punto sobre la conveniencia de ambos padres. Los cromosomas se seleccionan al azar y se designan como un "punto de cruce". Los bits a la derecha de ese punto se intercambian entre los dos cromosomas padres. Esto da como resultado dos descendientes, cada uno con cierta información genética de ambos padres.
Cruce de dos puntos y puntos k
En el cruce de dos puntos, se seleccionan aleatoriamente dos puntos de cruce de los cromosomas originales. Los bits entre los dos puntos se intercambian entre los organismos padres.
El cruce de dos puntos equivale a realizar dos cruces de un solo punto con diferentes puntos de cruce. Esta estrategia se puede generalizar a un cruce de k puntos para cualquier entero positivo k, eligiendo k puntos de cruce.
Cruce uniforme
En el cruce uniforme, normalmente, cada bit se elige de cualquiera de los padres con la misma probabilidad. A veces se utilizan otras proporciones de mezcla, lo que da como resultado que la descendencia herede más información genética de un padre que del otro. En un cruce uniforme, no dividimos el cromosoma en segmentos, sino que tratamos cada gen por separado. En esto, básicamente lanzamos una moneda por cada cromosoma para decidir si se incluirá o no en la descendencia.
Cruce para genomas enteros o de valor real
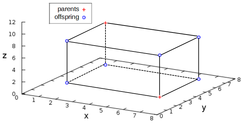
Para los operadores de crossover presentados arriba y para la mayoría de los operadores de crossover para cadenas de bits, sostiene que también se pueden aplicar en consecuencia a genomas enteros o de valor real cuyos genes cada uno consiste en un número entero o valor real. En lugar de bits individuales, números enteros o reales son entonces simplemente copiados en el genoma infantil. La descendencia se encuentra en las esquinas restantes del hipercuerpo azotado por los dos padres y , como se ejemplifica en la imagen adjunta para el caso tridimensional.
Recombinación discreta
Si las reglas del cruce uniforme para cadenas de bits se aplican durante la generación de la descendencia, esto también se denomina recombinación discreta.
Recombinación intermedia

En este operador de recombinación, los valores de alelo del genoma infantil se generan mezclando los alelos de los dos genomas padres y :
- aleatoriamente distribuido por gen
La elección del intervalo causa que además del interior del hipercuerpo abarcado por los valores de alelos de los genes padres, además de cierto entorno para la gama de valores de la descendencia está en cuestión. Un valor se recomienda para contrarrestar la tendencia a reducir los valores de alelo que de otro modo existe .
La figura adyacente muestra para el caso bidimensional la gama de posibles alelos nuevos de los dos padres ejemplares y en recombinación intermedia. La descendencia de la recombinación discreta y también están conspirados. La recombinación intermedia satisface el cálculo aritmético de los valores de alelo del genoma infantil requerido por la teoría del alfabeto virtual. La recombinación discreta e intermedia se utiliza como un estándar en la estrategia de evolución.
Cruce para permutaciones
Para tareas combinatorias, las permutaciones se utilizan generalmente que están específicamente diseñados para los genomas que son ellos mismos permutaciones de un conjunto. El conjunto subyacente es generalmente un subconjunto de o . Si el cruce de 1 punto o n o uniforme para genomas enteros se utiliza para tales genomas, un genoma infantil puede contener algunos valores dos veces y otros pueden estar desaparecidos. Esto puede ser remediado reparación genética, por ejemplo, reemplazando los genes redundantes en fidelidad posicional para los desaparecidos del otro genoma infantil.
Para evitar la generación de descendencia inválida, se han desarrollado operadores cruzados especiales para permutaciones que cumplen los requisitos básicos de dichos operadores para permutaciones, es decir, que todos los elementos de la permutación inicial también estén presentes en la nueva y solo los Se cambia el orden. Se puede distinguir entre tareas combinatorias, donde todas las secuencias son admisibles, y aquellas donde existen restricciones en forma de secuencias parciales inadmisibles. Un representante bien conocido del primer tipo de tarea es el problema del viajante de comercio (TSP), donde el objetivo es visitar un conjunto de ciudades exactamente una vez en el recorrido más corto. Un ejemplo del tipo de tarea restringida es la programación de múltiples flujos de trabajo. Los flujos de trabajo implican restricciones de secuencia en algunos de los pasos de trabajo individuales. Por ejemplo, no se puede cortar una rosca hasta que se haya perforado el orificio correspondiente en una pieza de trabajo. Estos problemas también se denominan permutaciones basadas en orden.
A continuación, se presentan dos operadores de cruce como ejemplos: el cruce parcialmente mapeado (PMX) motivado por el TSP y el cruce de orden (OX1) diseñado para permutaciones basadas en orden. En cada caso se puede generar una segunda descendencia intercambiando los cromosomas originales.
Cruce parcialmente mapeado (PMX)
El operador PMX fue diseñado como un operador de recombinación para problemas similares a TSP. La explicación del procedimiento se ilustra con un ejemplo:
| Procedimiento | Ejemplo | Ejemplo cromosoma | ||
| Se les dará dos permutaciones del mismo conjunto. | y | |||
| Aleatoriamente seleccione dos puntos de cruce que forman un segmento de genes en . | Aquí desde la posición del gen 4 a 6. | |||
| La sección seleccionada se copia al cromosoma infantil en la misma posición. | Las posiciones abiertas se indican por marcas de preguntas. | |||
| Busque genes que no hayan sido copiados en el segmento correspondiente comenzando en el primer punto de cruce. Para cada gen encontrado (llamado ), mirar hacia arriba en la descendencia que elemento (llamado ) fue copiado en su lugar de . Copiado a la posición de dentro si no está ocupado. De lo contrario, continúe con el siguiente paso. | Gene es el primer gen no copiado en el segmento correspondiente : . Gene fue copiado de en su lugar en . | |||
| La posición de dentro es la posición más derecha y será colocado allí dentro . | ||||
| Si el lugar es ocupado dentro ya está ocupado por un elemento en el descendiente, es puesto en el lugar tomado por dentro . | El siguiente gen en es y esto ya ha sido copiado en el cromosoma infantil. Así, el siguiente gen a ser manejado es . Su posición en la descendencia sería la posición de dentro . Sin embargo, este lugar ya está ocupado por gen . Así que... es copiado a la posición de dentro . | |||
| Después de procesar los genes del segmento seleccionado en , las posiciones restantes en la descendencia se llenan con los genes de que aún no han sido copiados, en el orden de su apariencia. Esto resulta en el genoma infantil terminado. | Los genes copiados de son y . | |||
Cruce de órdenes (OX1)
El cruce de orden se remonta a Davis en su forma original y se presenta aquí en una versión ligeramente generalizada con más de dos puntos de cruce. Transfiere información sobre el orden relativo del segundo padre a la descendencia. En primer lugar, el número y la posición de los puntos de cruce se determinan aleatoriamente. Luego, las secuencias genéticas resultantes se procesan como se describe a continuación:
| Procedimiento | Ejemplo | Ejemplo cromosoma | ||
| Se les dará dos permutaciones del mismo conjunto. | y | |||
| Selección aleatoria de segmentos de genes en . | Aquí dos segmentos de la posición de genes 1 a 2 y de 6 a 8. | |||
| Como permutación infantil, se genera una permutación que contiene los segmentos de genes seleccionados de en la misma posición. | Las posiciones abiertas se indican por marcas de preguntas. | |||
| Los genes restantes desaparecidos son transferidos ahora, pero en el orden en que aparecen en . | Los genes desaparecidos en el ejemplo son: | |||
| Esto resulta en el genoma infantil completado. | Los genes transferidos se subrayan: | |||
Entre otras cosas, el cruce de pedidos es muy adecuado para programar múltiples flujos de trabajo, cuando se utiliza junto con el cruce de 1 y n puntos.
Otros operadores cruzados para permutaciones
Con el tiempo, se ha propuesto una gran cantidad de operadores de cruce para permutaciones, por lo que la siguiente lista es solo una pequeña selección. Para obtener más información, se remite al lector a la literatura.
- ciclo crossover (CX)
- cruce de órdenes (OX2)
- crossover basado en la posición (POS)
- recombinación de bordes
- (VR)
- alternancia-posiciones crossover (AP)
- maximal preservativo crossover (MPX)
- fusión de crossover (MX)
- operador de crossover constructivo secuencial (SCX)
El enfoque habitual para resolver problemas similares a los TSP mediante algoritmos genéticos o, más generalmente, evolutivos, presentado anteriormente, es reparar los descendientes ilegales o ajustar los operadores apropiadamente para que, en primer lugar, no surja descendencia ilegal. Alternativamente, Riazi sugiere el uso de una representación cromosómica doble, lo que evita la descendencia ilegal.







![{displaystyle alpha _{i}=alpha _{i,P_{1}}cdot beta _{i}+alpha _{i,P_{2}}cdot left(1-beta _{i}right)quad {mathsf {with}}quad beta _{i}in left[-d,1+dright]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/fc170b904e37aa9566766d4923975ca1bb1a0176)

![{displaystyle [-d,1+d]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0aed59f903817803e978e0eeb9b7c516db5f43b5)





































