Cromosoma 5
El Cromosoma 5 es uno de los 23 pares de cromosomas del ser humano. Las personas normalmente tienen dos copias de este cromosoma. El cromosoma 5 abarca alrededor de 182 millones de pares de bases (los componentes básicos del ADN) y representa casi el 6% del ADN total de las células. El cromosoma 5 es el quinto cromosoma humano más grande, pero tiene una de las densidades genéticas más bajas. Esto se explica en parte por numerosas regiones pobres en genes que muestran un grado notable de conservación sinténica y no codificante con vertebrados no mamíferos, lo que sugiere que están funcionalmente limitadas.
Debido a que el cromosoma 5 es responsable de muchas formas de crecimiento y desarrollo (divisiones celulares), los cambios pueden causar cáncer. Un ejemplo sería la leucemia mieloide aguda (AML).
Genes
Número de genes
Las siguientes son algunas de las estimaciones del recuento de genes del cromosoma 5 humano. Debido a que los investigadores utilizan diferentes enfoques para la anotación del genoma, sus predicciones sobre la cantidad de genes en cada cromosoma varían (para obtener detalles técnicos, consulte predicción de genes). Entre varios proyectos, el proyecto colaborativo de secuencia de codificación por consenso (CCDS) adopta una estrategia extremadamente conservadora. Por lo tanto, la predicción del número de genes del CCDS representa un límite inferior en el número total de genes que codifican proteínas humanas.
| Estimación | genes de codificación de proteínas | genes de ARN no codificación | Pseudogenes | Fuente | Fecha de lanzamiento |
|---|---|---|---|---|---|
| CCDS | 839 | — | — | 2016-09-08 | |
| HGNC | 790 | 355 | 574 | 2017-05-12 | |
| Ensembl | 882 | 1.207 | 707 | 2017-03-29 | |
| UniProt | 875 | — | — | 2018-02-28 | |
| NCBI | 886 | 981 | 785 | 2017-05-19 |
Lista de genes
La siguiente es una lista parcial de genes en el cromosoma 5 humano. Para obtener una lista completa, consulte el enlace en el cuadro de información a la derecha.
- ABLIM3: proteína de codificación Proteína de LIM de unión de Actina 3
- ADAMTS2: ADAM metallopeptidase con trombospondin tipo 1 motivo, 2
- AGXT2: Alanine-glyoxylate aminotransferase 2
- ANKRD31: proteína de codificación Ankyrin repetir dominio 31
- APBB3: proteína de codificación Amyloid beta A4 precursor familia de unión de proteínas B miembro 3
- APC: adenomatosis poliposis coli
- ARL15: proteína de codificación ADP-ribosylation factor-like 15
- BRIX1: Proteína de biogénesis ríboa BRX1 homolog (también BXDC2)
- C1QTNF3: proteína relacionada con el factor de necrosis tumoral C1q 3
- C5orf45: cromosoma 5 marco de lectura abierta 45
- C5orf47: proteína de codificación cromosoma 5 marco de lectura abierta 47
- C5orf49: proteína de codificación cromosoma 5 marco de lectura abierta 49
- CAST: Calpastatin
- CDO1: proteína de codificación Cisteine dioxygenasa tipo 1
- CPLANE1: Ciliogénesis y Efector de Polaridad Planar 1
- CPLX2: Complexin-2
- CREBRF: proteína de codificación CREB3 factor regulador
- CXXC5: proteína de los dedos de zing tipo CXXC 5
- DPYSL3: Proteína similar a la dihidropirimidinasa 3
- EGR1: proteína de respuesta al crecimiento temprano 1
- ERAP1: endoplasmática reticulum aminopeptidase 1 (previously called ARTS-1)
- ERAP2: endoplasmática reticulum aminopeptidase 2
- ESM1: molécula endotelial 1
- DTDST: transportador de displasia diastrófica
- EIF4E1B: proteína de codificación Eukaryotic factor de iniciación de la traducción 4E miembro de la familia 1B
- ERCC8: reparación de escisión deficiencia de reparación de roedores, grupo de complementación 8
- FAF2: proteína de codificación Fas asociado factor familiar 2
- FAM172A: proteína de codificación UPF0528 proteína FAM172A
- FAM105B: proteína de codificación Familia con semejanza de secuencia 105, miembro B
- FAM114A2: proteína de codificación FAM114A2
- FAM71B: proteína de codificación Familia con semejanza de secuencia 71 miembro B
- FASTKD3: proteína de dominio de la cinosa FAST 3
- FBXL7: F-box/LRR-repeat protein 7
- FCHSD1: FCH y doble proteína de dominio SH3 1
- FGF1: factor de crecimiento fibroblasto 1 (factor de crecimiento fibroblasto adictivo)
- FGFR4: receptor del factor de crecimiento fibroblasto 4
- GM2A: GM2 ganglioside activador
- GNPDA1: Glucosamina-6-phosphate isomerase 1
- GPBP1: Vasculin
- HEXB: hexosaminidase B (beta polipeptide)
- HMGXB3: proteína de codificación HMG-box que contiene 3
- IK: Protein Red
- IRX1: Iroquois-class homeodomain protein (human)
- LARP1: Proteína relacionada con la La 1
- LMAN2: Manguera de Lectin vinculante 2
- Proteína de codificación LNCR3 Susceptibilidad al cáncer de pulmón 3
- LPCAT1: Lysophosphatidylcholine acyltransferase 1
- LYRM7: proteína de codificación LYR motivo que contiene 7
- LYSMD3: LysM y peptidoglycan putative proteína que contiene dominio 3
- MAN2A1: Alpha-mannosidase 2
- MASS1: susceptibilidad a las incautaciones monogénicas 1 homolog (mouse)
- MCC: proteína de cáncer mutante colorrectal
- MCCC2: metilcrotonoyl-Coenzyme Un carboxilase 2 (beta)
- MCEF: factor de transcripción Familia AF4/FMR2, miembro 4
- MEF2C: Factor de potenciador específico de miocito 2C
- MEF2C-AS1: proteína de codificación ARN antisentidos MEF2C 1
- MGAT1: Mannosil (alpha-1,3-)-glycoproteína beta-1,2-N-acetylglucosaminyltransferase
- MINAR2: proteína de codificación Kiaa1024 como
- MIR1271: microRNA 1271
- MIR146A: microRNA 146a
- MTRR: 5-metiltetrahidrofolate-homocysteine Reductas de metiltransferasa
- MZB1: Zona marginal Proteína específica B y B1
- NIPBL: Nipped-B homolog (Drosophila)
- NREP: proteína relacionada con la regeneración neuronal
- Proteína de codificación NSA2 proteína nuclear inducible TGF 1
- NSD1: Proteína del Coregulator de la transcripción
- NSUN2: familia de dominio NOP2/Sun, miembro 2
- NR2F1: Receptor de hormona nuclear
- NSG2: proteína de codificación Proteína Hmp19
- NUDCD2: proteína de dominio NudC 2
- P4HA2: Prolyl 4-hidroxilase subunit alpha-2
- PCBD2: Pterin-4-alpha-carbinolamine dehydratase 2
- PELO: Pelota homolog
- PGT1B: proteína de codificación Proteína geranilgeranyltransferase tipo I subunidad beta
- PHAX: Adaptador fosforilado para la exportación de ARN
- Pikachurin: Responsable del funcionamiento de la cinta sinapsis; permite que el ojo rastree objetos móviles
- PFDN1: Subunidad de precarburos 1
- POLR3G: proteína de codificación Polimerasa (RNA) III (DNA dirigida) polipéptido G (32kD)
- PPIP5K2: Diphosphoinositol pentakisphosphate kinase 2
- PRCC1: Bobina en espiral rica en prolina 1
- PRR16: proteína de codificación Proteína rica en proteínas 16
- PURA: Proteína de unión de elementos ricos en purina A
- PWWP2A: proteína de codificación PWWP dominio que contiene 2A
- RANBP3L: proteína de codificación RAN proteína de unión 3-como
- RASGEF1C: proteína de codificación RasGEF miembro de la familia de dominio 1C
- RMND5B: Necesario para la división nuclear meiotica 5 homolog B
- SFXN1: Sideroflexin- 1
- SKIV2L2: Ski2 como ARN helicase 2
- SLC22A5: familia portadora de soluto 22 (transportador de cación orgánica), miembro 5
- SLC26A2: familia portadora de soluto 26 (transportador sulfate), miembro 2
- SH3TC2: dominio y tetratricopeptide repite 2
- SLCO4C1: transportador de anión orgánico de alta velocidad familiar 4c1
- SLU7: factor de multiplicación previa al ARN
- SMN1: neurona motor de supervivencia 1, telomeric
- SMN2: neurona motor de supervivencia 2, centromeric
- SNCAIP: sínucleina, proteína de interacción alfa (sinfilina)
- SPEF2: proteína de la bandera de esperma 2
- SPINK5: inhibidor de la serina proteasa Kazal-type 5 (LEKTI)
- SPINK6: inhibidor de la proteasa serina
- SPINK9: inhibidor de la serina proteasa Kazal-type 9 (LEKTI-2)
- SPZ1: Proteína de cremallera de leucina espermatógena 1
- STC2: Stanniocalcin- 2
- TBCA: Tubulin-specific chaperone A
- TCOF1: síndrome de Treacher Collins-Franceschetti 1
- TGFBI: keratoepithelin
- THG1L: Probable tRNA(Sus) guanylyltransferase
- TICAM2: molécula de adaptador de dominio TIR 2
- TMEM171: proteína de codificación Proteína transmembrana 171
- TNFAIP8: Factor de necrosis tumoral, proteína alfa 8
- TSSK1B: proteína de codificación Testis específico de la serina kinase 1b
- TTC37: dominio de repetición de tetratricopeptide 37
- UPF0488: codifica la vía de señalización de proteínas del receptor refrigerada por proteínas G
- YIPF5: Yip1 miembro de la familia de dominio 5
- YTHDC2: proteína de codificación YTH dominio que contiene 2
- ZBED3: Dedo de zinc proteína de dominio BED 3
- ZNF608: proteína de codificación Proteína de dedo zinc 608
Enfermedades y trastornos
Las siguientes son algunas de las enfermedades relacionadas con genes ubicados en el cromosoma 5:
- Trastorno de déficit de atención/hiperactividad
- Achondrogenesis tipo 1B
- Atelosteogenesis, tipo II
- Trastorno bipolar
- Síndrome de atrofia óptica Bosch-Boonstra-Schaaf
- Enfermedad de Charcot-Marie-Tooth, tipo 4
- Síndrome de cockayne
- Síndrome de Cornelia de Lange
- Distrofia corneal de capa Bowman
- Cri du chat
- Displasia diastrófica
- Síndrome de Ehlers-Danlos
- Poliposis adenomatoso familiar
- Ditrofia granular tipo I
- Distrofia granular de maíz II
- GM2-gangliosidosis, variante AB
- Homocystinuria
- 3-Methylcrotonyl-CoA deficiencia de carboxylase
- Síndrome mielodisplásico
- Síndrome de Netherton
- Dependencia de Nicotina
- Enfermedad de Parkinson
- Deficiencia de carnitina primaria
- Displasia epifiseal múltiple recesiva
- Enfermedad de Sandhoff
- Atrofia muscular espinal
- Síndrome de sotos
- Atrofia muscular espinal del motor de supervivencia
- Síndrome de Treacher Collins
- Síndrome Tricho-hepato-enteric
- Síndrome de usher
Condiciones cromosómicas
Las siguientes afecciones son causadas por cambios en la estructura o el número de copias del cromosoma 5:
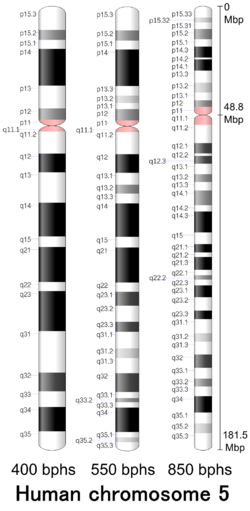
- El síndrome de Cri-du-chat es causado por una eliminación del extremo del brazo corto (p) del cromosoma 5. Este cambio cromosómico está escrito como 5p-. Los signos y síntomas del síndrome de cri-du-chat probablemente están relacionados con la pérdida de múltiples genes en esta región. Los investigadores no han identificado todos estos genes o determinado cómo su pérdida conduce a las características del trastorno. Ellos han descubierto, sin embargo, que una eliminación mayor tiende a dar lugar a retrasos mentales más graves y retrasos en el desarrollo en personas con síndrome de cri-du-chat.
- Los investigadores han definido regiones estrechas del brazo corto del cromosoma 5 que están asociadas con características particulares del síndrome de cri-du-chat. Una región específica designada 5p15.3 está asociada con un llanto tipo gato, y una región cercana llamada 5p15.2 está asociada con retraso mental, cabeza pequeña (microcefalia), y características faciales distintivas.
- Adenomatous familiar La poliposis es causada por la eliminación del gen supresor tumoral APC en el brazo largo (q) del cromosoma 5. Este cambio cromosómico resulta en miles de pólipos colónicos que le da al paciente un riesgo del 100% de cáncer de colon si no se realiza la colectomía total.
- El síndrome de eliminación cromosoma 5q es causado por la eliminación del brazo q (arriba larga) del cromosoma 5. Esta eliminación se ha relacionado con varios trastornos relacionados con la sangre, como el síndrome mielodisplásico y la eritroblastopenia. Esta es una condición diferente a Cri-du-chat que se mencionó anteriormente.
- Otros cambios en el número o estructura del cromosoma 5 pueden tener una variedad de efectos, incluyendo el crecimiento retardado y el desarrollo, características faciales distintivas, defectos de nacimiento y otros problemas médicos. Los cambios al cromosoma 5 incluyen un segmento extra del brazo corto (p) o largo (q) del cromosoma en cada célula (tristomía parcial 5p o 5q), un segmento perdido del brazo largo del cromosoma en cada célula (monotomía parcial 5q), y una estructura circular llamada cromosoma del anillo 5. Un cromosoma de anillo ocurre cuando ambos extremos de un cromosoma roto se reúnen.
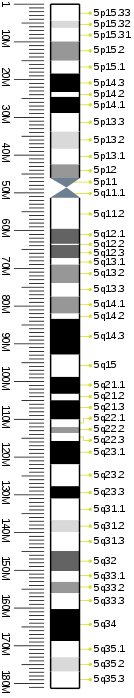
Banda citogenética
| Chr. | Arma | Banda | ISCN Empieza | ISCN Para | Basepair Empieza | Basepair Para | Stain | Densidad |
|---|---|---|---|---|---|---|---|---|
| 5 | p | 15.33 | 0 | 278 | 1 | 4.400.000 | gneg | |
| 5 | p | 15.32 | 278 | 401 | 4.400.001 | 6.300.000 | gpos | 25 |
| 5 | p | 15.31 | 401 | 555 | 6.300.001 | 9.900.000 | gneg | |
| 5 | p | 15.2 | 555 | 802 | 9.900.001 | 15,000,000 | gpos | 50 |
| 5 | p | 15.1 | 802 | 972 | 15.000.001 | 18.400.000 | gneg | |
| 5 | p | 14.3 | 972 | 1234 | 18.400.001 | 23.300,000 | gpos | 100 |
| 5 | p | 14.2 | 1234 | 1281 | 23.300.001 | 24.600,000 | gneg | |
| 5 | p | 14.1 | 1281 | 1543 | 24.600.001 | 28.900,000 | gpos | 100 |
| 5 | p | 13.3 | 1543 | 1836 | 28.900.001 | 33,800,000 | gneg | |
| 5 | p | 13.2 | 1836 | 2068 | 33.800.001 | 38.400,000 | gpos | 25 |
| 5 | p | 13.1 | 2068 | 2253 | 38.400.001 | 42,500,000 | gneg | |
| 5 | p | 12 | 2253 | 2407 | 42.500.001 | 46,100.000 | gpos | 50 |
| 5 | p | 11 | 2407 | 2592 | 46,100,001 | 48,800,000 | acen | |
| 5 | q | 11.1 | 2592 | 2839 | 48.800.001 | 51,400,000 | acen | |
| 5 | q | 11.2 | 2839 | 3271 | 51,400,001 | 59.600.000 | gneg | |
| 5 | q | 12.1 | 3271 | 3518 | 59.600.001 | 63.600.000 | gpos | 75 |
| 5 | q | 12.2 | 3518 | 3580 | 63.600.001 | 63,900,000 | gneg | |
| 5 | q | 12.3 | 3580 | 3765 | 63.900.001 | 67.400.000 | gpos | 75 |
| 5 | q | 13.1 | 3765 | 4012 | 67.400.001 | 69.100.000 | gneg | |
| 5 | q | 13.2 | 4012 | 4197 | 69.100.001 | 74,000,000 | gpos | 50 |
| 5 | q | 13.3 | 4197 | 4397 | 74.000.001 | 77.600,000 | gneg | |
| 5 | q | 14.1 | 4397 | 4752 | 77.600.001 | 82.100.000 | gpos | 50 |
| 5 | q | 14.2 | 4752 | 4907 | 82.100.001 | 83.500,000 | gneg | |
| 5 | q | 14.3 | 4907 | 5400 | 83.500.001 | 93,000,000 | gpos | 100 |
| 5 | q | 15 | 5400 | 5678 | 93.000.001 | 98.900,000 | gneg | |
| 5 | q | 21.1 | 5678 | 5879 | 98.900.001 | 103.400.000 | gpos | 100 |
| 5 | q | 21.2 | 5879 | 5987 | 103.400.001 | 105.100.000 | gneg | |
| 5 | q | 21.3 | 5987 | 6295 | 105.100.001 | 110,200,000 | gpos | 100 |
| 5 | q | 22.1 | 6295 | 6419 | 110.200.001 | 112.200.000 | gneg | |
| 5 | q | 22.2 | 6419 | 6527 | 112.200.001 | 113.800.000 | gpos | 50 |
| 5 | q | 22.3 | 6527 | 6666 | 113.800.001 | 115,900,000 | gneg | |
| 5 | q | 23.1 | 6666 | 6943 | 115.900.001 | 122.100.000 | gpos | 100 |
| 5 | q | 23.2 | 6943 | 7267 | 122.100.001 | 127.900,000 | gneg | |
| 5 | q | 23.3 | 7267 | 7468 | 127.900.001 | 131.200.000 | gpos | 100 |
| 5 | q | 31.1 | 7468 | 7807 | 131.200.001 | 136,900,000 | gneg | |
| 5 | q | 31.2 | 7807 | 8008 | 136.900.001 | 140.100.000 | gpos | 25 |
| 5 | q | 31.3 | 8008 | 8316 | 140.100.001 | 145.100.000 | gneg | |
| 5 | q | 32 | 8316 | 8625 | 145.100.001 | 150.400.000 | gpos | 75 |
| 5 | q | 33.1 | 8625 | 8887 | 150.400.001 | 153.300.000 | gneg | |
| 5 | q | 33.2 | 8887 | 9072 | 153.300.001 | 156.300,000 | gpos | 50 |
| 5 | q | 33.3 | 9072 | 9304 | 156.300.001 | 160.500,000 | gneg | |
| 5 | q | 34 | 9304 | 9690 | 160.500.001 | 169,000,000 | gpos | 100 |
| 5 | q | 35.1 | 9690 | 9952 | 169.000.001 | 173,300,000 | gneg | |
| 5 | q | 35.2 | 9952 | 10183 | 173,300,001 | 177.100.000 | gpos | 25 |
| 5 | q | 35.3 | 10183 | 10600 | 177.100.001 | 181,538,259 | gneg |