Cre recombinasa
Cre recombinasa es una enzima tirosina recombinasa derivada del bacteriófago P1. La enzima utiliza un mecanismo similar a la topoisomerasa I para llevar a cabo eventos de recombinación específicos de un sitio. La enzima (38 kDa) es miembro de la familia integrasa de recombinasas específicas de sitio y se sabe que cataliza el evento de recombinación específica de sitio entre dos sitios de reconocimiento de ADN (sitios LoxP). Este sitio de reconocimiento loxP de 34 pares de bases (pb) consta de dos secuencias palindrómicas de 13 pb que flanquean una región espaciadora de 8 pb. Los productos de la recombinación mediada por Cre en los sitios loxP dependen de la ubicación y orientación relativa de los sitios loxP. Dos especies de ADN separadas que contienen sitios loxP pueden sufrir fusión como resultado de la recombinación mediada por Cre. Se dice que las secuencias de ADN que se encuentran entre dos sitios loxP están "floxadas". En este caso, los productos de la recombinación mediada por Cre dependen de la orientación de los sitios loxP. El ADN que se encuentre entre dos sitios loxP orientados en la misma dirección se cortará como un bucle circular de ADN, mientras que el ADN intermedio entre dos sitios loxP que están orientados de manera opuesta se invertirá. La enzima no requiere cofactores adicionales (como ATP) ni proteínas accesorias para su función.
La enzima desempeña funciones importantes en el ciclo de vida del bacteriófago P1, como la ciclación del genoma lineal y la resolución de los cromosomas diméricos que se forman después de la replicación del ADN.
La Cre recombinasa es una herramienta ampliamente utilizada en el campo de la biología molecular. El sistema de recombinación único y específico de la enzima se aprovecha para manipular genes y cromosomas en una amplia gama de investigaciones, como estudios de eliminación o activación de genes. La capacidad de la enzima para operar eficientemente en una amplia gama de entornos celulares (incluidos mamíferos, plantas, bacterias y levaduras) permite que el sistema de recombinación Cre-Lox se utilice en una gran cantidad de organismos, lo que lo convierte en una herramienta particularmente útil. herramienta en la investigación científica.
Discovery
Los estudios realizados en 1981 por Sternberg y Hamilton demostraron que el bacteriófago 'P1' tenía un sistema de recombinación específico de sitio único. Se generaron fragmentos EcoRI del genoma del bacteriófago P1 y se clonaron en vectores lambda. Se encontró que un fragmento EcoRI de 6,5 kb (Fragmento 7) permitía eventos de recombinación eficientes. Se sabía que el mecanismo de estos eventos de recombinación era único, ya que ocurrían en ausencia de las proteínas bacterianas RecA y RecBCD. Los componentes de este sistema de recombinación se dilucidaron mediante estudios de mutagénesis por deleción. Estos estudios demostraron que para que se produjeran eventos de recombinación eficientes se necesitaba un producto del gen P1 y un sitio de recombinación. El producto del gen P1 se denominó Cre (cprovoca la combinación re) y el El sitio de recombinación se denominó loxP (locus de cruce (x) over, P1). La proteína Cre se purificó en 1983 y se descubrió que era una proteína de 35.000 Da. No se requieren cofactores de alta energía como ATP o proteínas accesorias para la actividad recombinasa de la proteína purificada. Los primeros estudios también demostraron que Cre se une a secuencias de ADN no específicas mientras que tiene una afinidad 20 veces mayor por las secuencias loxP y los resultados de los primeros estudios de huella de ADN también sugirieron que las moléculas de Cre se unen a sitios loxP como dímeros.

| Miembros de la familia Tyrosine recombinase |
|---|
| S.cerevisiae Flp recombinase |
| Bacterial XerC recombinase |
| Bacterial XerD recombinase |
| Proteína de integración |
| Proteína de integración HP1 |
Estructura
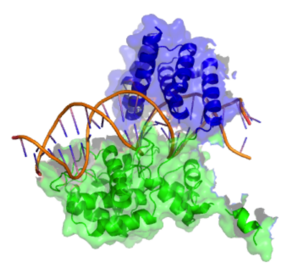
Cre recombinasa consta de 343 aminoácidos que forman dos dominios distintos. El dominio amino terminal abarca los residuos 20 a 129 y este dominio contiene 5 segmentos alfa helicoidales unidos por una serie de bucles cortos. Hélices A y E están involucrados en la formación del tetrámero de recombinasa y se sabe que la región del extremo C de la hélice E forma contactos con el dominio C terminal de las subunidades adyacentes. Hélices B & D forman contactos directos con el surco principal del ADN loxP. Se cree que estas dos hélices establecen tres contactos directos con las bases del ADN en el sitio loxP. El dominio carboxi terminal de la enzima consta de los aminoácidos 132 a 341 y alberga el sitio activo de la enzima. La estructura general de este dominio comparte un gran parecido estructural con el dominio catalítico de otras enzimas de la misma familia, como λ Integrase y HP1 Integrase. Este dominio tiene una estructura predominantemente helicoidal con 9 hélices distintas (F-N). La hélice terminal (N) sobresale del cuerpo principal del dominio carboxi y se cree que esta hélice desempeña un papel en la mediación de interacciones con otras subunidades. Las estructuras cristalinas demuestran que esta hélice N terminal entierra su superficie hidrofóbica en un bolsillo aceptor de una subunidad Cre adyacente.
El efecto de la estructura de dos dominios es formar una abrazadera en forma de C que sujeta el ADN desde lados opuestos.
Sitio activo

El sitio activo de la enzima Cre consta de los residuos de la tríada catalítica conservados Arg 173, His 289, Arg 292, así como de los residuos nucleofílicos conservados Tyr 324 y Trp 315. A diferencia de algunas enzimas recombinasas como la recombinasa Flp, Cre no Forman un sitio activo compartido entre subunidades separadas y todos los residuos que contribuyen al sitio activo se encuentran en una sola subunidad. En consecuencia, cuando dos moléculas de Cre se unen en un único sitio loxP, hay dos sitios activos presentes. La recombinación mediada por Cre requiere la formación de una sinapsis en la que dos complejos Cre-LoxP se asocian para formar lo que se conoce como tetrámero de sinapsis en el que están presentes cuatro sitios activos distintos. Tyr 324 actúa como nucleófilo para formar un enlace covalente 3'-fosfotirosina al sustrato de ADN. El fosfato escindible (fosfato dirigido al ataque nucleofílico en el sitio de escisión) está coordinado por las cadenas laterales de los 3 residuos de aminoácidos de la tríada catalítica (Arg 173, His 289 y Trp 315). El nitrógeno indol del triptófano 315 también forma un enlace de hidrógeno con este fosfato divisible. (nb A Histidina ocupa este sitio en otros miembros de la familia de la tirosina recombinasa y realiza la misma función). Esta reacción escinde el ADN y libera un grupo hidroxilo 5'. Este proceso ocurre en el sitio activo de dos de las cuatro subunidades de recombinasa presentes en el tetrámero de la sinapsis. Si los grupos 5' hidroxilo atacan el enlace 3'-fosfotirosina, un par de cadenas de ADN se intercambiarán para formar un intermediario de unión Holliday.
Aplicaciones
Papel en bacteriófago P1
La recombinasa Cre desempeña funciones importantes en el ciclo de vida del bacteriófago P1. Tras la infección de una célula, el sistema Cre-loxP se utiliza para provocar la circularización del ADN P1. Además de esto, Cre también se utiliza para resolver el ADN P1 lisogénico dimérico que se forma durante la división celular del fago.
Uso en investigación
La activación inducible de Cre se logra utilizando la variante CreER (receptor de estrógeno), que solo se activa después de la administración de tamoxifeno. Esto se hace mediante la fusión de un dominio de unión de ligando mutado del receptor de estrógeno a la recombinasa Cre, lo que hace que Cre se active específicamente por el tamoxifeno. En ausencia de tamoxifeno, CreER provocará el transporte de la recombinasa mutada al citoplasma. La proteína permanecerá en este lugar en su estado inactivado hasta que se administre tamoxifeno. Una vez que se introduce el tamoxifeno, se metaboliza en 4-hidroxitamoxifeno, que luego se une al RE y produce la translocación del CreER al núcleo, donde luego puede escindir los sitios lox. Es importante destacar que a veces los reporteros fluorescentes pueden activarse en ausencia de tamoxifeno, debido a la fuga de unas pocas moléculas de recombinasa Cre hacia el núcleo que, en combinación con reporteros muy sensibles, da como resultado un marcaje celular no deseado. CreER(T2) se desarrolló para minimizar la recombinación independiente del tamoxifeno y maximizar la sensibilidad al tamoxifeno.
Mejoras
En los últimos años, la recombinasa Cre se ha mejorado con la conversión a codones preferidos de mamíferos, la eliminación de sitios de empalme crípticos informados, un codón de parada alterado y un contenido reducido de CpG para reducir el riesgo de silenciamiento epigenético en mamíferos. También se han identificado varios mutantes con mayor precisión.
Véase también
- Cre-Lox recombinación
- FLP-FRT recombinación
- Cre/loxP-System (en alemán)
Referencias
- ^ Nagy A (Feb 2000). "Cre recombinase: el reactivo universal para la adaptación del genoma". Génesis. 26 (2): 99–109. doi:10.1002/(SICI)1526-968X(200002)26:2 obtenidos99::AID-GENE1⁄3.0.CO;2-B. PMID 10686599.
- ^ a b c d Abremski K, Hoess R (Feb 1984). "Bacteriophage P1 site-specific recombination. Purificación y propiedades de la proteína Cre recombinase". El Diario de Química Biológica. 259 (3): 1509-1514. doi:10.1016/S0021-9258(17)43437-5. PMID 6319400.
- ^ a b c d Van Duyne GD (2001). "Una visión estructural de la recombinación específica del sitio cre-loxp". Annual Review of Biophysics and Biomolecular Structure. 30: 87–104. doi:10.1146/annurev.biophys.30.1.87. PMID 11340053.
- ^ Ennifar E, Meyer JE, Buchholz F, Stewart AF, Suck D (Sep 2003). "La estructura Cristal de una sinapsis Cre recombinase-loxP de tipo salvaje revela una nueva conformación de espaciadora sugiriendo un mecanismo alternativo para la activación del escote de ADN". Nucleic Acids Research. 31 (18): 5449–5460. doi:10.1093/nar/gkg732. PMC 203317. PMID 12954782.
- ^ a b Sternberg N, Hamilton D (Aug 1981). "Bacteriophage P1 site-specific recombination. I. Recombination between loxP sites". Journal of Molecular Biology. 150 (4): 467–486. doi:10.1016/0022-2836(81)90375-2. PMID 6276557.
- ^ a b Guo F, Gopaul DN, van Duyne GD (Sep 1997). "Structure of Cre recombinase complexed with DNA in a site-specific recombination synapse". Naturaleza. 389 (6646): 40–46. Código:1997Natur.389...40G. doi:10.1038/37925. PMID 9288963. S2CID 4401434.
- ^ Shaikh AC, Sadowski PD (Feb 1997). "El Cre recombinase escucha el sitio de lox en trans". El Diario de Química Biológica. 272 (9): 5695–5702. doi:10.1074/jbc.272.9.5695. PMID 9038180.
- ^ Walrath JC, Hawes JJ, Van Dyke T, Reilly KM (2010). "Modelos de ratón diseñados genéticamente en investigación de cáncer". Avances en investigación del cáncer. 106: 113–64. doi:10.1016/S0065-230X(10)06004-5. ISBN 9780123747716. PMC 3533445. PMID 20399958.
- ^ Kristianto J, Johnson MG, Zastrow RK, Radcliff AB, Blank RD (junio de 2017). "Actividad espontánea de recombinase Cre-ERT2 in vivo". Transgenic Research. 26 (3): 411-417. doi:10.1007/s11248-017-0018-1. PMC 9474299. PMID 28409408. S2CID 4377498.
- ^ Álvarez-Aznar A, Martínez-Corral I, Daubel N, Betsholtz C, Mäkinen T, Gaengel K (febrero 2020). "T2 líneas". Transgenic Research. 29 (1): 53–68. doi:10.1007/s11248-019-00177-8. PMC 7000517. PMID 31641921.
- ^ Shimshek DR, Kim J, Hübner MR, Spergel DJ, Buchholz F, Casanova E, Stewart AF, Seeburg PH, Sprengel R (Jan 2002). "Condon-improbado Cre recombinase (iCre) expresión en el ratón". Génesis. 32 (1): 19–26. doi:10.1002/gene.10023. PMID 11835670. S2CID 46000513.
- ^ Eroshenko N, Church GM (Sep 2013). "Mutantes de Cre recombinase con mejor precisión". Nature Communications. 4: 2509. Bibcode:2013NatCo...4.2509E. doi:10.1038/ncomms3509. PMC 3972015. PMID 24056590.
Enlaces externos
- Cre recombinase en la Biblioteca Nacional de Medicina de EE.UU.