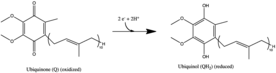Complejo respiratorio I

Complejo respiratorio I, EC 7.1.1.2 (también conocido como NADH:ubiquinona oxidorreductasa, NADH deshidrogenasa tipo I y mitocondrial complejo I) es el primer gran complejo proteico de las cadenas respiratorias de muchos organismos, desde bacterias hasta humanos. Cataliza la transferencia de electrones del NADH a la coenzima Q10 (CoQ10) y transloca protones a través de la membrana mitocondrial interna en eucariotas o la membrana plasmática de bacterias.
Esta enzima es esencial para el funcionamiento normal de las células, y las mutaciones en sus subunidades conducen a una amplia gama de trastornos neuromusculares y metabólicos hereditarios. Los defectos en esta enzima son responsables del desarrollo de varios procesos patológicos como el daño por isquemia/reperfusión (ictus e infarto cardíaco), la enfermedad de Parkinson y otros.
Función
El complejo I es la primera enzima de la cadena de transporte de electrones mitocondrial. Hay tres enzimas transductoras de energía en la cadena de transporte de electrones: NADH: ubiquinona oxidorreductasa (complejo I), coenzima Q: citocromo c reductasa (complejo III) y citocromo c oxidasa (complejo IV). El complejo I es la enzima más grande y complicada de la cadena de transporte de electrones.
La reacción catalizada por el complejo I es:
- NADH + H+ + CoQ + 4H+dentro→ NAD+ + CoQH2 + 4H+Fuera.
En este proceso, el complejo traslada cuatro protones a través de la membrana interna por molécula de NADH oxidado, lo que ayuda a generar la diferencia de potencial electroquímico que se utiliza para producir ATP. El complejo I de Escherichia coli (NADH deshidrogenasa) es capaz de translocar protones en la misma dirección al Δψ establecido, lo que demuestra que en las condiciones probadas, el ion de acoplamiento es H+. Se observó transporte de Na+ en sentido contrario, y aunque Na+ no era necesario para las actividades catalíticas o de transporte de protones, su presencia aumentaba esta última. H+ fue translocado por el complejo I de Paracoccus denitrificans, pero en este caso, el transporte de H+ no estuvo influenciado por Na+< /sup>, y no se observó transporte de Na+. Posiblemente, la E. coli complejo I tiene dos sitios de acoplamiento de energía (uno Na+ independiente y el otro Na+dependiente), como se observa para el Rhodothermus marinus complejo I, mientras que el mecanismo de acoplamiento del P. La enzima denitrificans es completamente independiente de Na+. También es posible que otro transportador catalice la captación de Na+. La transducción de energía del complejo I por bombeo de protones puede no ser exclusiva del R. marinus enzima. La actividad antiporte Na+/H+ no parece ser una propiedad general del complejo I. Sin embargo, la existencia de actividad translocadora de Na+ del complejo I todavía está en cuestión.
La reacción se puede revertir (lo que se conoce como reducción de NAD+ asistida por succinato aeróbico mediante ubiquinol) en presencia de un alto potencial de membrana, pero se desconoce el mecanismo catalítico exacto. La fuerza impulsora de esta reacción es un potencial a través de la membrana que puede mantenerse por hidrólisis de ATP o por los complejos III y IV durante la oxidación del succinato.
El complejo I puede desempeñar un papel en el desencadenamiento de la apoptosis. De hecho, se ha demostrado que existe una correlación entre las actividades mitocondriales y la muerte celular programada (PCD) durante el desarrollo del embrión somático.
El complejo I no es homólogo a la familia de la NADH deshidrogenasa (NDH) translocadora de Na+ (TC# 3.D.1), un miembro del Mrp transportador de Na+ superfamilia.
Como resultado de la oxidación de dos moléculas de NADH a NAD+, el Complejo V (ATP sintasa) puede producir tres moléculas de ATP corriente abajo en la cadena respiratoria.
Mecanismo
Mecanismo general
Todas las reacciones redox tienen lugar en el dominio hidrofílico del complejo I. El NADH inicialmente se une al complejo I y transfiere dos electrones al grupo prostético del mononucleótido de flavina (FMN) de la enzima, creando FMNH2. El aceptor de electrones, el anillo de isoaloxazina, de FMN es idéntico al de FAD. Luego, los electrones se transfieren a través del FMN a través de una serie de grupos de hierro-azufre (Fe-S) y, finalmente, a la coenzima Q10 (ubiquinona). Este flujo de electrones cambia el estado redox de la proteína, induciendo cambios conformacionales de la proteína que alteran los valores de pK de la cadena lateral ionizable y hace que se bombeen cuatro iones de hidrógeno fuera de la matriz mitocondrial. La ubiquinona (CoQ) acepta dos electrones para reducirse a ubiquinol (CoQH2).
Mecanismo de transferencia de electrones
La vía propuesta para el transporte de electrones antes de la reducción de ubiquinona es la siguiente: NADH – FMN – N3 – N1b – N4 – N5 – N6a – N6b – N2 – Q, donde Nx es una convención de etiquetado para grupos de azufre de hierro. El alto potencial de reducción del grupo N2 y la relativa proximidad de los otros grupos en la cadena permiten una transferencia de electrones eficiente a larga distancia en la proteína (con tasas de transferencia de NADH al grupo de hierro-azufre N2 de aproximadamente 100 μs).
La dinámica de equilibrio del Complejo I está impulsada principalmente por el ciclo redox de la quinona. En condiciones de alta fuerza motriz de protones (y, en consecuencia, un grupo concentrado de ubiquinol), la enzima funciona en la dirección inversa. El ubiquinol se oxida a ubiquinona y los protones liberados resultantes reducen la fuerza motriz del protón.
Mecanismo de translocación de protones
El acoplamiento de la translocación de protones y el transporte de electrones en el Complejo I se propone actualmente como indirecto (cambios conformacionales de largo alcance) en lugar de directo (intermedios redox en las bombas de hidrógeno como en los grupos hemo de los Complejos III y IV). La arquitectura de la región hidrofóbica del complejo I muestra múltiples transportadores de protones que están interconectados mecánicamente. Los tres componentes centrales que se cree que contribuyen a este evento de cambio conformacional de largo alcance son el grupo de hierro-azufre N2 acoplado al pH, la reducción de quinona y las subunidades de hélice transmembrana del brazo de la membrana. Transducción de cambios conformacionales para impulsar los transportadores transmembrana unidos por una 'varilla de conexión' durante la reducción de la ubiquinona se pueden dar cuenta de dos o tres de los cuatro protones bombeados por NADH oxidado. El protón restante debe ser bombeado por acoplamiento directo en el sitio de unión de la ubiquinona. Se propone que los mecanismos de acoplamiento directo e indirecto representen el bombeo de los cuatro protones.
La proximidad del grupo N2 a un residuo de cisteína cercano da como resultado un cambio conformacional tras la reducción de las hélices cercanas, lo que lleva a cambios pequeños pero importantes en la conformación general de la proteína. Otros estudios de resonancia paramagnética de electrones de la transferencia de electrones han demostrado que la mayor parte de la energía que se libera durante la reducción subsiguiente de CoQ se encuentra en el paso final de formación de ubiquinol a partir de semiquinona, lo que proporciona evidencia del "golpe único" Mecanismo de translocación H+ (es decir, los cuatro protones se mueven a través de la membrana al mismo tiempo). Las teorías alternativas sugieren un "mecanismo de dos tiempos" donde cada paso de reducción (semiquinona y ubiquinol) da como resultado un golpe de dos protones que ingresan al espacio intermembrana.
El ubiquinol resultante localizado en el dominio de la membrana interactúa con residuos cargados negativamente en el brazo de la membrana, estabilizando los cambios conformacionales. Se ha propuesto un mecanismo antiportador (intercambio de Na+/H+) usando evidencia de residuos de Asp conservados en el brazo de la membrana. La presencia de residuos de Lys, Glu y His permite la activación de protones (una protonación seguida de un evento de desprotonación a través de la membrana) impulsada por el pKa de los residuos.
Composición y estructura
NADH: la ubiquinona oxidorreductasa es el mayor de los complejos respiratorios. En los mamíferos, la enzima contiene 44 proteínas de membrana periférica solubles en agua separadas, que están ancladas a los constituyentes integrales de la membrana. De particular importancia funcional son el grupo prostético de flavina (FMN) y ocho grupos de hierro-azufre (FeS). De las 44 subunidades, siete están codificadas por el genoma mitocondrial.
La estructura es una "L" forma con un dominio de membrana largo (con alrededor de 60 hélices transmembrana) y un dominio hidrofílico (o periférico), que incluye todos los centros redox conocidos y el sitio de unión de NADH. Los trece de la E. coli, que comprenden la NADH deshidrogenasa I, se codifican dentro del operón nuo y son homólogas a las subunidades del complejo mitocondrial I. Cada una de las subunidades de tipo antiportador NuoL/M/N contiene 14 hélices transmembrana (TM) conservadas. Dos de ellos son discontinuos, pero la subunidad NuoL contiene una hélice α anfipática de 110 Å de largo, que abarca toda la longitud del dominio. La subunidad, NuoL, está relacionada con los antiportadores Na+/ H+ de TC# 2.A.63.1.1 (PhaA y PhaD).
Tres de las subunidades unidas a la membrana conservadas en la NADH deshidrogenasa están relacionadas entre sí y con los antiportadores de protones de sodio Mrp. El análisis estructural de dos complejos procarióticos reveló que las tres subunidades contienen cada una catorce hélices transmembrana que se superponen en alineaciones estructurales: la translocación de tres protones puede estar coordinada por una hélice lateral que los conecta.
El complejo I contiene un bolsillo de unión de ubiquinona en la interfaz de las subunidades de 49 kDa y PSST. Cerca del grupo de hierro y azufre N2, el donante de electrones inmediato propuesto para la ubiquinona, una tirosina altamente conservada constituye un elemento crítico del sitio de reducción de la quinona. Una posible ruta de intercambio de quinona conduce desde el grupo N2 a la hoja beta N-terminal de la subunidad de 49 kDa. Se han secuenciado las 45 subunidades del NDHI bovino. Cada complejo contiene FMN unido de forma no covalente, coenzima Q y varios centros de hierro y azufre. Los NDH bacterianos tienen 8-9 centros de hierro y azufre.
Un estudio reciente utilizó espectros de resonancia paramagnética de electrones (EPR) y resonancia de doble electrón-electrón (DEER) para determinar la ruta de transferencia de electrones a través de los complejos de hierro y azufre, que se encuentran en el dominio hidrofílico. Siete de estos grupos forman una cadena desde los sitios de unión de flavina a quinona; el octavo grupo está ubicado al otro lado del flavin y se desconoce su función. Los resultados de EPR y DEER sugieren un perfil de energía potencial alterno o de "montaña rusa" para la transferencia de electrones entre los sitios activos y a lo largo de los cúmulos de hierro-azufre, lo que puede optimizar la velocidad de viaje de los electrones y permitir una conversión de energía eficiente en el complejo I.
| # | Subunidad humana/bovina | Proteína humana | Descripción de proteínas (UniProt) | Familia Pfam con proteína humana | |
|---|---|---|---|---|---|
| Subunidades básicasa | |||||
| 1 | NDUFS7 / PSST / NUKM | NDUS7_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 7, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF01058 | |
| 2 | NDUFS8 / TYKY / NUIM | NDUS8_HUMAN | NADH deshidrogenasa proteína 8, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF12838 | |
| 3 | NDUFV2 / 24kD / NUHMc | NDUV2_HUMAN | NADH deshidrogenasa [ubiquinona] flavoproteína 2, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF01257 | |
| 4 | NDUFS3 / 30kD / NUGM | NDUS3_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 3, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF00329 | |
| 5 | NDUFS2 / 49kD / NUCM | NDUS2_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 2, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF00346 | |
| 6 | NDUFV1 / 51kD / NUBM | NDUV1_HUMAN | NADH deshidrogenasa [ubiquinona] flavoproteína 1, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF01512 | |
| 7 | NDUFS1 / 75kD / NUAM | NDUS1_HUMAN | NADH-ubiquinone oxidoreductase 75 kDa subunit, mitocondrial EC 1.6.5.3 EC 1.6.99.3 | Pfam PF00384 | |
| 8 | ND1 / NU1M | NU1M_HUMAN | NADH-ubiquinone oxidoreductase chain 1 EC 1.6.5.3 | Pfam PF00146 | |
| 9 | ND2 / NU2M | NU2M_HUMAN | NADH-ubiquinone oxidoreductase chain 2 EC 1.6.5.3 | Pfam PF00361, Pfam PF06444 | |
| 10 | ND3 / NU3M | NU3M_HUMAN | NADH-ubiquinone oxidoreductase chain 3 EC 1.6.5.3 | Pfam PF00507 | |
| 11 | ND4 / NU4M | NU4M_HUMAN | NADH-ubiquinone oxidoreductase chain 4 EC 1.6.5.3 | Pfam PF01059, Pfam PF00361 | |
| 12 | ND4L / NULM | NU4LM_HUMAN | NADH-ubiquinone oxidoreductase chain 4L EC 1.6.5.3 | Pfam PF00420 | |
| 13 | ND5 / NU5M | NU5M_HUMAN | NADH-ubiquinone oxidoreductase chain 5 EC 1.6.5.3 | Pfam PF00361, Pfam PF06455, Pfam PF00662 | |
| 14 | ND6 / NU6M | NU6M_HUMAN | NADH-ubiquinone oxidoreductase chain 6 EC 1.6.5.3 | Pfam PF00499 | |
| Subunidades accesorias básicasb | |||||
| 15 | NDUFS6 / 13A | NDUS6_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 6, mitocondrial | Pfam PF10276 | |
| 16 | NDUFA12 / B17.2 | NDUAC_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 12 | Pfam PF05071 | |
| 17 | NDUFS4 / AQDQ | NDUS4_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 4, mitocondrial | Pfam PF04800 | |
| 18 | NDUFA9 / 39kDa | NDUA9_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 9, mitocondrial | Pfam PF01370 | |
| 19 | NDUFAB1 / ACPM | ACPM_HUMAN | Proteína de portador de acyl, mitocondrial | Pfam PF00550 | |
| 20 | NDUFA2 / B8 | NDUA2_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 2 | Pfam PF05047 | |
| 21 | NDUFA1 / MFWE | NDUA1_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 1 | Pfam PF15879 | |
| 22 | NDUFB3 / B12 | NDUB3_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 3 | Pfam PF08122 | |
| 23 | NDUFA5 / AB13 | NDUA5_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 5 | Pfam PF04716 | |
| 24 | NDUFA6 / B14 | NDUA6_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 6 | Pfam PF05347 | |
| 25 | NDUFA11 / B14.7 | NDUAB_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 11 | Pfam PF02466 | |
| 26 | NDUFB11 / ESSS | NDUBB_HUMAN | NADH dehydrogenase [ubiquinona] 1 beta subcomplex subunit 11, mitocondrial | Pfam PF10183 | |
| 27 | NDUFS5 / PFFD | NDUS5_HUMAN | NADH deshidrogenasa [ubiquinona] proteína de hierro-sulfur 5 | Pfam PF10200 | |
| 28 | NDUFB4 / B15 | NDUB4_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 4 | Pfam PF07225 | |
| 29 | NDUFA13 /A13 | NDUAD_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 13 | Pfam PF06212 | |
| 30 | NDUFB7 / B18 | NDUB7_HUMAN | NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 7 | Pfam PF05676 | |
| 31 | NDUFA8 / PGIV | NDUA8_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 8 | Pfam PF06747 | |
| 32 | NDUFB9 / B22 | NDUB9_HUMAN | NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 9 | Pfam PF05347 | |
| 33 | NDUFB10 / PDSW | NDUBA_HUMAN | NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 10 | Pfam PF10249 | |
| 34 | NDUFB8 / ASHI | NDUB8_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 8, mitocondrial | Pfam PF05821 | |
| 35 | NDUFC2 / B14.5B | NDUC2_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad C2 | Pfam PF06374 | |
| 36 | NDUFB2 / AGGG | NDUB2_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 2, mitocondrial | Pfam PF14813 | |
| 37 | NDUFA7 / B14.5A | NDUA7_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 7 | Pfam PF07347 | |
| 38 | NDUFA3 / B9 | NDUA3_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 3 | Pfam PF14987 | |
| 39 | NDUFA4 / MLRQc,d | NDUA4_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 4 | Pfam PF06522 | |
| 40 | NDUFB5 / SGDH | NDUB5_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 5, mitocondrial | Pfam PF09781 | |
| 41 | NDUFB1 / MNLL | NDUB1_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 1 | Pfam PF08040 | |
| 42 | NDUFC1 / KFYI | NDUC1_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad C1, mitocondrial | Pfam PF15088 | |
| 43 | NDUFA10 / 42kD | NDUAA_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 10, mitocondrial | Pfam PF01712 | |
| 44 | NDUFA4L2 | NUA4L_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subunidad subcomplex alfa 4-como 2 | Pfam PF15880 | |
| 45 | NDUFV3 | NDUV3_HUMAN | NADH dehidrogenasa [ubiquinona] flavoproteína 3, 10kDa | - | |
| 46 | NDUFB6 | NDUB6_HUMAN | NADH dehidrogenasa [ubiquinona] 1 beta subcomplex subunidad 6 | Pfam PF09782 | |
| proteínas del factor de la Asamblea | |||||
| 47 | NDUFAF1c | CIA30_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subcomplex alfa, factor de montaje 1 | Pfam PF08547 | |
| 48 | NDUFAF2 | MIMIT_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subcomplex alfa, factor de montaje 2 | Pfam PF05071 | |
| 49 | NDUFAF3 | NDUF3_HUMAN | NADH dehidrogenasa [ubiquinona] 1 factor de montaje subcomplex alfa 3 | Pfam PF05071 | |
| 50 | NDUFAF4 | NDUF4_HUMAN | NADH dehidrogenasa [ubiquinona] 1 subcomplex alfa, factor de montaje 4 | Pfam PF06784 | |
Notas:
- a encontrado en todas las especies excepto hongos
- b Puede o no estar presente en ninguna especie
- c encontrado en especies fúngicas como Esquizosaccharomyces pombe
- d Investigaciones recientes han descrito NDUFA4 como una subunidad de complejo IV, y no de complejo I
Inhibidores
La bullatacina (una acetogenina que se encuentra en la fruta Asimina triloba) es el inhibidor conocido más potente de la NADH deshidrogenasa (ubiquinona) (IC50 = 1,2 nM, más fuerte que la rotenona). El inhibidor más conocido del complejo I es la rotenona (comúnmente utilizada como pesticida orgánico). La rotenona y los rotenoides son isoflavonoides que se encuentran en varios géneros de plantas tropicales como Antonia (Loganiaceae), Derris y Lonchocarpus (Faboideae, Fabaceae). Ha habido informes de los pueblos indígenas de la Guayana Francesa que usan plantas que contienen rotenona para pescar, debido a su efecto ictiotóxico, ya en el siglo XVII. La rotenona se une al sitio de unión de la ubiquinona del complejo I, así como a la piericidina A, otro potente inhibidor con un homólogo estructural cercano a la ubiquinona.
Las acetogeninas de Annonaceae son inhibidores aún más potentes del complejo I. Se entrecruzan con la subunidad ND2, lo que sugiere que ND2 es esencial para la unión de quinonas. La rolliniastatina-2, una acetogenina, es el primer inhibidor del complejo I encontrado que no comparte el mismo sitio de unión que la rotenona.
A pesar de más de 50 años de estudio del complejo I, no se han encontrado inhibidores que bloqueen el flujo de electrones dentro de la enzima. Los inhibidores hidrófobos como la rotenona o la piericidina muy probablemente interrumpen la transferencia de electrones entre el grupo terminal N2 de FeS y la ubiquinona. Se ha demostrado que la inhibición sistémica a largo plazo del complejo I por la rotenona puede inducir la degeneración selectiva de las neuronas dopaminérgicas.
El complejo I también es bloqueado por la adenosina difosfato ribosa, un inhibidor competitivo reversible de la oxidación de NADH, al unirse a la enzima en el sitio de unión del nucleótido. Tanto el NADH hidrofílico como los análogos de la ubiquinona hidrofóbica actúan al principio y al final de la ruta interna de transporte de electrones, respectivamente.
Se ha demostrado que el fármaco antidiabético metformina induce una inhibición leve y transitoria del complejo I de la cadena respiratoria mitocondrial, y esta inhibición parece desempeñar un papel clave en su mecanismo de acción.
La inhibición del complejo I se ha implicado en la hepatotoxicidad asociada con una variedad de fármacos, por ejemplo, flutamida y nefazodona.
Transición activa/inactiva
Las propiedades catalíticas del complejo eucariótico I no son simples. Existen dos formas catalítica y estructuralmente distintas en cualquier preparación dada de la enzima: una es la forma A "activa" totalmente competente, y la otra es la forma D catalíticamente silenciosa, latente, "inactiva". Después de la exposición de la enzima inactiva a temperaturas elevadas pero fisiológicas (>30 °C) en ausencia de sustrato, la enzima se convierte en la forma D. Esta forma es catalíticamente incompetente, pero puede activarse mediante la reacción lenta (k~4 min−1) de oxidación de NADH con la subsiguiente reducción de ubiquinona. Después de una o varias rotaciones, la enzima se vuelve activa y puede catalizar la reacción fisiológica NADH:ubiquinona a una velocidad mucho mayor (k~104 min−1). En presencia de cationes divalentes (Mg2+, Ca2+), oa pH alcalino la activación tarda mucho más.
La alta energía de activación (270 kJ/mol) del proceso de desactivación indica la ocurrencia de importantes cambios conformacionales en la organización del complejo I. Sin embargo, hasta ahora, la única diferencia conformacional observada entre estas dos formas es el número de residuos de cisteína expuestos en la superficie de la enzima. El tratamiento de la forma D del complejo I con los reactivos de sulfhidrilo N-etilmaleimida o DTNB bloquea de forma irreversible los residuos críticos de cisteína, anulando la capacidad de la enzima para responder a la activación, inactivándola así de forma irreversible. La forma A del complejo I es insensible a los reactivos de sulfhidrilo.
Se encontró que estos cambios conformacionales pueden tener un significado fisiológico muy importante. La forma inactiva, pero no la activa, del complejo I era susceptible de inhibición por los nitrosotioles y el peroxinitrito. Es probable que la transición de la forma activa a la inactiva del complejo I tenga lugar durante condiciones patológicas cuando el recambio de la enzima está limitado a temperaturas fisiológicas, como durante la hipoxia, la isquemia o cuando aumenta la proporción tisular de óxido nítrico:oxígeno (es decir, hipoxia metabólica).
Producción de superóxido
Investigaciones recientes sugieren que el complejo I es una potente fuente de especies reactivas de oxígeno. El complejo I puede producir superóxido (así como peróxido de hidrógeno), a través de al menos dos vías diferentes. Durante la transferencia directa de electrones, solo se producen cantidades muy pequeñas de superóxido (probablemente menos del 0,1% del flujo total de electrones).
Durante la transferencia inversa de electrones, el complejo I podría ser el sitio más importante de producción de superóxido dentro de las mitocondrias, con alrededor del 3-4 % de los electrones desviados a la formación de superóxido. Transferencia inversa de electrones, el proceso mediante el cual los electrones del conjunto reducido de ubiquinol (suministrado por la succinato deshidrogenasa, la glicerol-3-fosfato deshidrogenasa, la flavoproteína de transferencia de electrones o la dihidroorotato deshidrogenasa en las mitocondrias de los mamíferos) pasan a través del complejo I para reducir el NAD+< /sup> a NADH, impulsada por el potencial eléctrico de la membrana mitocondrial interna. Aunque no se sabe con precisión en qué condiciones patológicas ocurriría la transferencia de electrones inversos in vivo, los experimentos in vitro indican que este proceso puede ser una fuente muy potente de superóxido cuando las concentraciones de succinato son altas y las concentraciones de oxalacetato o malato son bajas. Esto puede ocurrir durante la isquemia tisular, cuando se bloquea el suministro de oxígeno.
El superóxido es una especie de oxígeno reactivo que contribuye al estrés oxidativo celular y está relacionado con las enfermedades neuromusculares y el envejecimiento. La NADH deshidrogenasa produce superóxido al transferir un electrón de FMNH2 (o flavina semireducida) al oxígeno (O2). El residuo de flavina radical es inestable y transfiere el electrón restante a los centros de hierro-azufre. Es la proporción de NADH a NAD+ lo que determina la tasa de formación de superóxido.
Patología
Las mutaciones en las subunidades del complejo I pueden causar enfermedades mitocondriales, incluido el síndrome de Leigh. Las mutaciones puntuales en varias subunidades del complejo I derivadas del ADN mitocondrial (ADNmt) también pueden provocar la neuropatía óptica hereditaria de Leber. Existe alguna evidencia de que los defectos del complejo I pueden desempeñar un papel en la etiología de la enfermedad de Parkinson, quizás debido a las especies reactivas del oxígeno (el complejo I puede, como el complejo III, filtrar electrones al oxígeno, formando superóxido altamente tóxico).
Aunque la etiología exacta de la enfermedad de Parkinson no está clara, es probable que la disfunción mitocondrial, junto con la inhibición del proteasoma y las toxinas ambientales, desempeñen un papel importante. De hecho, se ha demostrado que la inhibición del complejo I provoca la producción de peróxidos y una disminución de la actividad del proteasoma, lo que puede conducir a la enfermedad de Parkinson. Además, Esteves et al. (2010) encontraron que las líneas celulares con la enfermedad de Parkinson muestran una mayor fuga de protones en el complejo I, lo que provoca una disminución de la capacidad respiratoria máxima.
La lesión por isquemia/reperfusión cerebral está mediada por el deterioro del complejo I. Recientemente se descubrió que la privación de oxígeno conduce a condiciones en las que el complejo mitocondrial I pierde su cofactor natural, el mononucleótido de flavina (FMN) y se vuelve inactivo. Cuando hay oxígeno presente, la enzima cataliza una reacción fisiológica de oxidación de NADH por la ubiquinona, suministrando electrones aguas abajo de la cadena respiratoria (complejos III y IV). La isquemia conduce a un aumento dramático del nivel de succinato. En presencia de succinato, las mitocondrias catalizan la transferencia de electrones inversos, de modo que la fracción de electrones del succinato se dirige corriente arriba al FMN del complejo I. La transferencia de electrones inversos da como resultado una reducción del complejo I FMN, una mayor generación de ROS, seguida de una pérdida del reducido cofactor (FMNH2) y deterioro de la producción de energía de las mitocondrias. La pérdida de FMN por lesión del complejo I y I/R puede aliviarse mediante la administración del precursor de FMN, riboflavina.
Estudios recientes han examinado otras funciones de la actividad del complejo I en el cerebro. Andreazza et al. (2010) encontraron que el nivel de actividad del complejo I estaba significativamente disminuido en pacientes con trastorno bipolar, pero no en pacientes con depresión o esquizofrenia. Descubrieron que los pacientes con trastorno bipolar mostraban una mayor oxidación y nitración de proteínas en la corteza prefrontal. Estos resultados sugieren que los estudios futuros deberían apuntar al complejo I para posibles estudios terapéuticos para el trastorno bipolar. Del mismo modo, Moran et al. (2010) encontraron que los pacientes con deficiencia grave del complejo I mostraban tasas de consumo de oxígeno disminuidas y tasas de crecimiento más lentas. Sin embargo, encontraron que las mutaciones en diferentes genes en el complejo I conducen a diferentes fenotipos, lo que explica las variaciones de las manifestaciones fisiopatológicas de la deficiencia del complejo I.
La exposición a pesticidas también puede inhibir el complejo I y causar síntomas de enfermedades. Por ejemplo, se ha demostrado que la exposición crónica a niveles bajos de diclorvos, un organofosfato utilizado como pesticida, causa disfunción hepática. Esto ocurre porque el diclorvos altera los niveles de actividad de los complejos I y II, lo que conduce a una disminución de las actividades de transferencia de electrones mitocondriales y una disminución de la síntesis de ATP.
En cloroplastos
Un complejo NADH deshidrogenasa que utiliza ubiquinona y bombea protones, homólogo al complejo I, se encuentra en los genomas del cloroplasto de la mayoría de las plantas terrestres bajo el nombre ndh. Este complejo se hereda de la simbiosis original de las cianobacterias, pero se ha perdido en la mayoría de las algas eucariotas, algunas gimnospermas (Pinus y gnetofitas) y algunos linajes muy jóvenes de angiospermas. El propósito de este complejo es originalmente críptico ya que el cloroplasto no participa en la respiración, pero ahora se sabe que ndh sirve para mantener la fotosíntesis en situaciones de estrés. Esto lo hace al menos parcialmente prescindible en condiciones favorables. Es evidente que los linajes de angiospermas sin ndh no duran mucho desde su juventud, pero se desconoce cómo sobreviven las gimnospermas en tierra sin ndh durante tanto tiempo.
Genes
La siguiente es una lista de genes humanos que codifican componentes del complejo I:
- NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa
- NDUFA1 – NADH dehydrogenase (ubiquinona) 1 subcomplex alfa, 1, 7.5kDa
- NDUFA2 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 2, 8kDa
- NDUFA3 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 3, 9kDa
- NDUFA4 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 4, 9kDa - recientemente descrito como parte de complejo IV
- NDUFA4L – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 4 similares
- NDUFA4L2 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 4-como 2
- NDUFA5 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 5, 13kDa
- NDUFA6 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 6, 14kDa
- NDUFA7 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 7, 14.5kDa
- NDUFA8 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 8, 19kDa
- NDUFA9 – NADH dehydrogenase (ubiquinona) 1 subcomplex alfa, 9, 39kDa
- NDUFA10 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 10, 42kDa
- NDUFA11 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 11, 14.7kDa
- NDUFA12 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, 12
- NDUFA13 – NADH dehydrogenase (ubiquinona) 1 subcomplex alfa, 13
- NDUFAB1 – NADH dehydrogenase (ubiquinona) 1, alfa/beta subcomplex, 1, 8kDa
- NDUFAF1 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, factor de montaje 1
- NDUFAF2 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, factor de montaje 2
- NDUFAF3 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, factor de montaje 3
- NDUFAF4 – NADH dehidrogenasa (ubiquinona) 1 subcomplex alfa, factor de montaje 4
- NADH dehidrogenasa (ubiquinona) 1 beta subcomplex
- NDUFB1 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 1, 7kDa
- NDUFB2 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 2, 8kDa
- NDUFB3 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 3, 12kDa
- NDUFB4 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 4, 15k Da
- NDUFB5 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 5, 16kDa
- NDUFB6 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 6, 17kDa
- NDUFB7 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 7, 18kDa
- NDUFB8 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 8, 19kDa
- NDUFB9 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 9, 22kDa
- NDUFB10 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 10, 22kDa
- NDUFB11 – NADH dehydrogenase (ubiquinona) 1 beta subcomplex, 11, 17.3kDa
- NADH dehydrogenase (ubiquinona) 1, subcomplex unknown
- NDUFC1 – NADH dehydrogenase (ubiquinone) 1, subcomplex unknown, 1, 6kDa
- NDUFC2 – NADH dehydrogenase (ubiquinona) 1, subcomplex unknown, 2, 14.5kDa
- NADH dehydrogenase (ubiquinona) Fe-S protein
- NDUFS1 – NADH dehydrogenase (ubiquinona) Fe-S protein 1, 75kDa (NADH-coenzyme Q reductase)
- NDUFS2 – NADH dehydrogenase (ubiquinona) Fe-S protein 2, 49kDa (NADH-coenzyme Q reductase)
- NDUFS3 – NADH dehydrogenase (ubiquinona) Fe-S protein 3, 30kDa (NADH-coenzyme Q reductase)
- NDUFS4 – NADH dehydrogenase (ubiquinona) Fe-S protein 4, 18kDa (NADH-coenzyme Q reductase)
- NDUFS5 – NADH dehydrogenase (ubiquinona) Fe-S protein 5, 15kDa (NADH-coenzyme Q reductase)
- NDUFS6 – NADH dehydrogenase (ubiquinona) Fe-S protein 6, 13kDa (NADH-coenzyme Q reductase)
- NDUFS7 – NADH dehydrogenase (ubiquinona) Fe-S protein 7, 20kDa (NADH-coenzyme Q reductase)
- NDUFS8 – NADH dehydrogenase (ubiquinona) Fe-S protein 8, 23kDa (NADH-coenzyme Q reductase)
- NADH dehidrogenasa (ubiquinona) flavoproteína 1
- NDUFV1 – NADH dehydrogenase (ubiquinona) flavoproteína 1, 51kDa
- NDUFV2 – NADH dehydrogenase (ubiquinona) flavoproteína 2, 24kDa
- NDUFV3 – NADH dehydrogenase (ubiquinona) flavoproteína 3, 10kDa
- mitocondrially encoded NADH dehydrogenase subunit
- MT-ND1 - subunidad de deshidrogenasa codificada mitocondrialmente 1
- MT-ND2 - subunidad de deshidrogenasa codificada mitocondrialmente 2
- MT-ND3 - subunidad de deshidrogenasa codificada mitocondrialmente 3
- MT-ND4 - mitocondrially encoded NADH dehydrogenase subunit 4
- MT-ND4L - mitocondrially encoded NADH dehydrogenase subunit 4L
- MT-ND5 - subunidad de deshidrogenasa codificada mitocondrialmente 5
- MT-ND6 - subunidad de deshidrogenasa codificada mitocondrialmente 6
Contenido relacionado
Aleta
DIU de cobre
Invertebrado