Clasificación de virus
La clasificación de virus es el proceso de nombrar los virus y colocarlos en un sistema taxonómico similar a los sistemas de clasificación utilizados para los organismos celulares.
Los virus se clasifican por características fenotípicas, como la morfología, el tipo de ácido nucleico, el modo de replicación, los organismos huéspedes y el tipo de enfermedad que causan. La clasificación taxonómica formal de los virus es responsabilidad del sistema del Comité Internacional de Taxonomía de Virus (ICTV), aunque el sistema de clasificación de Baltimore se puede utilizar para colocar los virus en uno de siete grupos según su forma de síntesis de ARNm. ICTV establece convenciones de nomenclatura específicas y pautas de clasificación adicionales.
Se ha propuesto un catálogo de todos los virus conocidos en el mundo y, en 2013, se estaban realizando algunos esfuerzos preliminares.
Definiciones
Definición de especie
Las especies forman la base de cualquier sistema de clasificación biológica. Antes de 1982, se pensaba que los virus no podían adaptarse al concepto reproductivo de especie de Ernst Mayr, por lo que no eran susceptibles de tal tratamiento. En 1982, el ICTV comenzó a definir una especie como "un grupo de cepas" con cualidades identificativas únicas. En 1991, se adoptó el principio más específico de que una especie de virus es una clase politética de virus que constituye un linaje replicante y ocupa un nicho ecológico particular.
En julio de 2013, la definición de especie de ICTV cambió para indicar: "Una especie es un grupo monofilético de virus cuyas propiedades se pueden distinguir de las de otras especies por múltiples criterios." Estos criterios incluyen la estructura de la cápside, la existencia de una envoltura, el programa de expresión génica para sus proteínas, el rango de huéspedes, la patogenicidad y, lo que es más importante, la similitud de la secuencia genética y la relación filogenética.
Los criterios reales utilizados varían según el taxón y, en ocasiones, pueden ser incoherentes (umbrales de similitud arbitrarios) o no estar relacionados con el linaje (geografía). El asunto, para muchos, aún no está resuelto.
Definición de virus
La definición formal y actualmente aceptada de un 'virus' fue aceptado por el Comité Ejecutivo de ICTV en noviembre de 2020 y ratificado en marzo de 2021, y es el siguiente:
Viruses sensu stricto son definidos operacionalmente por el ICTV como un tipo de MGE (elemento genético móvil) que codifica al menos una proteína que es un componente importante del virión que encaje el ácido nucleico del MGE respectivo y por lo tanto el gen que codifica la proteína de virión mayor en sí o MGEs que son claramente demostrables para ser miembros de una línea de ascendencia evolutiva de tales principales entidades de codificación de proteína virión. Cualquier grupo monofilético de MGEs que se origine de un ancestro de proteínas de codificación virión debe clasificarse como un grupo de virus.
Clasificación de las TIC
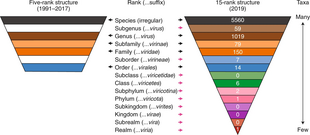
El Comité Internacional de Taxonomía de Virus comenzó a diseñar e implementar reglas para nombrar y clasificar virus a principios de la década de 1970, un esfuerzo que continúa hasta el presente. El ICTV es el único organismo encargado por la Unión Internacional de Sociedades Microbiológicas con la tarea de desarrollar, refinar y mantener una taxonomía universal de virus. El sistema comparte muchas características con el sistema de clasificación de los organismos celulares, como la estructura del taxón. Sin embargo, existen algunas diferencias, como el uso universal de la cursiva para todos los nombres taxonómicos, a diferencia del Código Internacional de Nomenclatura para algas, hongos y plantas y el Código Internacional de Nomenclatura Zoológica.
La clasificación viral comienza a nivel de reino y continúa de la siguiente manera, con los sufijos taxonómicos entre paréntesis:
- Reino-viria)
- Subrealm-vira)
- Reino (Reino Unido)-virae)
- Subkingdom (subkingdom)-viritos)
- Phylum-viricota)
- Subphylum (Subphylum)-viricotina)
- Clase (-viricetes)
- Subclase-viricetidae)
- Orden (Orden)-virales)
- Suborder (Suborder)-virineae)
- Familia (-viridae)
- Subfamilia-virinae)
- Genus-virus)
- Subgenus (Subgenus)-virus)
- Especies
- Subgenus (Subgenus)-virus)
- Genus-virus)
- Subfamilia-virinae)
- Familia (-viridae)
- Suborder (Suborder)-virineae)
- Orden (Orden)-virales)
- Subclase-viricetidae)
- Clase (-viricetes)
- Subphylum (Subphylum)-viricotina)
- Phylum-viricota)
- Subkingdom (subkingdom)-viritos)
- Reino (Reino Unido)-virae)
- Subrealm-vira)
A diferencia del sistema de nomenclatura binomial adoptado en las especies celulares, actualmente no existe una forma estandarizada para los nombres de las especies de virus. En la actualidad, el ICTV exige que el nombre de una especie debe contener la menor cantidad de palabras posible sin dejar de ser distinto, y no debe contener solo la palabra virus y el nombre del huésped. Los nombres de las especies a menudo toman la forma de virus de [enfermedad], particularmente para plantas y animales superiores. En 2019, ICTV publicó una propuesta para adoptar un sistema más formalizado de nomenclatura binomial para nombres de especies de virus, que se someterá a votación en 2020. Sin embargo, algunos virólogos se opusieron más tarde al posible cambio en el sistema de nombres, argumentando que el debate se produjo cuando muchos en el campo estaba preocupado debido a la pandemia de COVID-19.
A partir de 2021, se utilizan todos los niveles de taxones excepto el subreino, el subreino y la subclase. Se reconocen seis reinos, una clase incertae sedis, 19 familias incertae sedis y dos géneros incertae sedis:
Reinos: Duplodnaviria, Monodnaviria, Adnaviria, Ribozyviria, Riboviria, y Varidnaviria
Incertae sedis familias y clases:
- Alphasatellitidae
- Ampullaviridae
- Anelloviridae
- Avsunviroidae
- Bicaudaviridae
- Clavaviridae
- Finnlakeviridae
- Fuselloviridae
- Globuloviridae
- Guttaviridae
- Halspiviridae
- Naldaviricetes
- Ovaliviridae
- Plasmaviridae
- Polydnaviriformidae
- Portogloboviridae
- Pospiviroidae
- Spiraviridae
- Thaspiviridae
- Tolecusatellitidae
Incertae sedis géneros: Dinodnavirus, Rhizidiovirus
Clasificación de virus basada en estructuras
Se ha sugerido que la similitud en el ensamblaje y la estructura del virión observada para ciertos grupos virales que infectan huéspedes de diferentes dominios de la vida (p. ej., tectivirus bacterianos y adenovirus eucariotas o caudovirales procariotas y herpesvirus eucariotas) refleja una relación evolutiva entre estos virus. Por lo tanto, se ha sugerido que la relación estructural entre virus se utilice como base para definir taxones de nivel superior (linajes virales basados en la estructura) que podrían complementar el esquema de clasificación de ICTV de 2010.
El ICTV ha agregado gradualmente muchos taxones de nivel superior utilizando relaciones en pliegues de proteínas. Los cuatro reinos definidos en el lanzamiento de 2019 se definen por la presencia de una proteína de una determinada familia estructural.
Clasificación de Baltimore
La clasificación de Baltimore (definida por primera vez en 1971) es un sistema de clasificación que coloca a los virus en uno de siete grupos según una combinación de su ácido nucleico (ADN o ARN), cadena (monocatenaria o bicatenaria), sentido, y método de replicación. Nombrados en honor a David Baltimore, un biólogo ganador del Premio Nobel, estos grupos se designan con números romanos. Otras clasificaciones están determinadas por la enfermedad causada por el virus o su morfología, ninguna de las cuales es satisfactoria debido a que diferentes virus causan la misma enfermedad o se ven muy similares. Además, las estructuras virales suelen ser difíciles de determinar bajo el microscopio. Clasificar los virus según su genoma significa que todos los de una categoría dada se comportarán de manera similar, lo que ofrece alguna indicación de cómo proceder con futuras investigaciones. Los virus se pueden colocar en uno de los siete grupos siguientes:
- I: virus dsDNA (por ejemplo, Adenovirus, Herpesvirus, Poxvirus)
- II: ssDNA virus (+ hilo o "sentido") ADN (por ejemplo, Parvoviruses)
- III: virus dsRNA (por ejemplo, reovirus)
- IV:(+)ssRNA virus (+ hilo o sentido) RNA (por ejemplo, Coronavirus, Picornavirus, Togaviruses)
- V: (−)ssRNA virus (- hilo o antisentido) RNA (por ejemplo, ortomexovirus, rabdovirus)
- VI: SsRNA-RT virus (+ hilo o sentido) ARN con ADN intermedio en ciclo de vida (por ejemplo, retrovirus)
- VII: dsDNA-RT virus ADN con ARN intermedio en ciclo de vida (por ejemplo, Hepadnavirus)
Virus de ADN
Los virus con un genoma de ADN, a excepción de los virus de transcripción inversa de ADN, son miembros de tres de los cuatro reinos virales reconocidos: Duplodnaviria, Monodnaviria y Varidnaviria. Pero el orden incertae sedis Ligamenvirales y muchas otras familias y géneros incertae sedis también se utilizan para clasificar los virus de ADN. Los dominios Duplodnaviria y Varidnaviria consisten en virus de ADN de doble cadena; otros virus de ADN de doble cadena son incertae sedis. El dominio Monodnaviria consta de virus de ADN monocatenario que generalmente codifican una endonucleasa HUH; otros virus de ADN monocatenario son incertae sedis.
- Grupo I: los virus poseen ADN doble. Los virus que causan la varicela y los herpes se encuentran aquí.
- Grupo II: los virus poseen ADN de una sola tirada.
| Familia de virus | Ejemplos (nombres comunes) | Virion desnudo/desarrollado | Capsid simetría | Tipo de ácido nucleico | Grupo |
|---|---|---|---|---|---|
| 1. Adenoviridae | Virus de la hepatitis canina, Algunos tipos de frío común | Desnudo | Icosahedral | ds | I |
| 2. Papovaviridae | virus JC, VPH | Desnudo | Icosahedral | ds circular | I |
| 3. Parvoviridae | Parvovirus humano B19, canino parvovirus | Desnudo | Icosahedral | ss | II |
| 4. Herpesviridae | Herpes simplex virus, varicella-zoster virus, citomegalovirus, Epstein-Barr virus | Enveloped | Icosahedral | ds | I |
| 5. Poxviridae | Virus de la viruela, varicela, virus de mixoma, monopox, virus de la vacuna | Recubrimientos complejos | Complejo | ds | I |
| 6. Anelloviridae | Torque teno virus | Desnudo | Icosahedral | ss circular | II |
| 7. Pleolipoviridae | HHPV1, HRPV1 | Enveloped | ss/ds linear/circular | I/II |
Virus ARN
Todos los virus que tienen un genoma de ARN, y que codifican una ARN polimerasa dependiente de ARN (RdRp), son miembros del reino Orthornavirae, dentro del reino Riboviria.
- Grupo III: virus poseen genomas de ARN dobles, por ejemplo rotavirus.
- Grupo IV: los virus poseen genomas RNA de sentido positivo. Muchos virus bien conocidos se encuentran en este grupo, incluyendo los picornavirus (que es una familia de virus que incluye virus conocidos como el virus Hepatitis A, enterovirus, rinovirus, poliovirus y virus del pie y la boca), el virus del SARS, el virus de la hepatitis C, el virus de la fiebre amarilla y el virus de rubéola.
- Grupo V: los virus poseen genomas de ARN sin sentido negativo. Los virus del Ébola y Marburg son miembros bien conocidos de este grupo, junto con el virus de la gripe, el sarampión, las paperas y la rabia.
| Virus Family | Ejemplos (nombres comunes) | Capsid desnudo/desarrollado | Capsid Simmetría | Tipo de ácido nucleico | Grupo |
|---|---|---|---|---|---|
| 1. Reoviridae | Reovirus, rotavirus | Desnudo | Icosahedral | ds | III |
| 2. Picornaviridae | Enterovirus, rinovirus, hepatovirus, cardiovirus, aphthovirus, poliovirus, parechovirus, erbovirus, kobuvirus, teschovirus, coxsackie | Desnudo | Icosahedral | ss | IV |
| 3. Caliciviridae | Virus de Norwalk | Desnudo | Icosahedral | ss | IV |
| 4. Togaviridae | Encefalitis equino oriental | Enveloped | Icosahedral | ss | IV |
| 5. Arenaviridae | Virus de la coreomeningitis linfocítica, fiebre de Lassa | Enveloped | Complejo | ss(−) | V |
| 6. Flaviviridae | Virus de dengue, hepatitis virus C, virus de fiebre amarilla, virus Zika | Enveloped | Icosahedral | ss | IV |
| 7. Ortonomíaxoviridae | Influenzavirus A, influenzavirus B, influenzavirus C, isavirus, thogotovirus | Enveloped | Helical | ss(−) | V |
| 8. Paramyxoviridae | Virus de sarampión, virus de paperas, virus sincitial respiratorio, virus de Rinderpest, virus de distemper canino | Enveloped | Helical | ss(−) | V |
| 9. Bunyaviridae | California encephalitis virus, Sin nombre virus | Enveloped | Helical | ss(−) | V |
| 10. Rhabdoviridae | Virus de la rabia, estematitis vesicular | Enveloped | Helical | ss(−) | V |
| 11. Filoviridae | Ebola virus, Marburg virus | Enveloped | Helical | ss(−) | V |
| 12. Coronaviridae | coronavirus humano 229E, coronavirus humano NL63, coronavirus humano OC43, coronavirus humano HKU1, coronavirus relacionado con el síndrome respiratorio del Oriente Medio, coronavirus del síndrome respiratorio agudo grave y coronavirus del síndrome respiratorio agudo grave 2 | Enveloped | Helical | ss | IV |
| 13. Astroviridae | Astrovirus | Desnudo | Icosahedral | ss | IV |
| 14. Bornaviridae | Virus de la enfermedad de Borna | Enveloped | Helical | ss(−) | V |
| 15. Arteriviridae | Arterivirus, virus de la arteritis equina | Enveloped | Icosahedral | ss | IV |
| 16. Hepeviridae | Hepatitis E virus | Desnudo | Icosahedral | ss | IV |
Virus de transcripción inversa
Todos los virus que codifican una transcriptasa inversa (también conocida como RT o ADN polimerasa dependiente de ARN) son miembros de la clase Revtraviricetes, dentro del filo Arterviricota, reino Pararnavirae, y dominio Riboviria. La clase Blubervirales contiene la familia única Hepadnaviridae de virus de ADN RT (transcripción inversa); todos los demás virus RT son miembros de la clase Ortervirales.
- Grupo VI: los virus poseen virus de ARN unitarios que se replican a través de un intermediario de ADN. Los retrovirus se incluyen en este grupo, del cual el VIH es miembro.
- Grupo VII: los virus poseen genomas de ADN dobles y replican usando transcriptasa inversa. El virus de la hepatitis B se puede encontrar en este grupo.
| Virus Family | Ejemplos (nombres comunes) | Capsid desnudo/desarrollado | Capsid Simmetría | Tipo de ácido nucleico | Grupo |
|---|---|---|---|---|---|
| 1. Retroviridae | VIH | Enveloped | dimer RNA | VI | |
| 2. Caulimoviridae | Caulimovirus, Cacao swollen-shoot virus (CSSV) | Desnudo | VII | ||
| 3. Hepadnaviridae | Hepatitis B virus | Enveloped | Icosahedral | circular, parcialmente ds | VII |
Sistemas históricos
Clasificación de Holmes
Holmes (1948) utilizó una taxonomía linneana con nomenclatura binomial para clasificar los virus en 3 grupos bajo un orden, Virales. Se colocan de la siguiente manera:
- Grupo I: Phaginae (ataques bacterias)
- Grupo II: Fitofaginae (plantas de ataque)
- Grupo III: Zoophaginae (ataques animales)
El sistema no fue aceptado por otros debido a su descuido de las similitudes morfológicas.
Agentes subvirales
Los siguientes agentes infecciosos son más pequeños que los virus y solo tienen algunas de sus propiedades. Desde 2015, el ICTV permite clasificarlos de forma similar a los virus.
Viroides y agentes dependientes de virus
Viroides
- Familia Avsunviroidae
- Genus Avsunviroid; especie tipo: Avocado solblotch viroid
- Genus Pelamoviroid; especie tipo: Peach latente mosaico viroide
- Genus Elaviroid; especie tipo: Viroide de latente de berenjena
- Familia Pospiviroidae
- Genus Pospiviroid; especie tipo: Viroide de tubero husillo de patata
- Genus Hostuviroid; especie tipo: Hop stunt viroid
- Genus Cocadviroid; especie tipo: Coconut cadang-cadang viroid
- Genus Apscaviroid; especie tipo: Manzana cicatrizante piel viroide
- Genus Coleviroid; especie tipo: Coleus blumei viroid 1
Satélites
Los satélites dependen de la coinfección de una célula huésped con un virus auxiliar para la multiplicación productiva. Sus ácidos nucleicos tienen secuencias de nucleótidos sustancialmente distintas de su virus auxiliar o de su huésped. Cuando un agente subviral satélite codifica la proteína de cubierta en la que está encapsulado, se denomina virus satélite.
Los ácidos nucleicos tipo satélite se asemejan a los ácidos nucleicos satélite, ya que se replican con la ayuda de virus auxiliares. Sin embargo, difieren en que pueden codificar funciones que pueden contribuir al éxito de sus virus auxiliares; aunque a veces se los considera elementos genómicos de sus virus auxiliares, no siempre se encuentran dentro de sus virus auxiliares.
- Virus de satélites
- Virus de los satélites de ARN monoplazados
- (familia sin nombre)
- Aumaivirus – Satélite de mosaico de línea blanca de maíz
- Papanivirus – Panicum mosaic satellite virus
- Virtovirus – Satélite de mosaico de tabaco
- Albetovirus – Tobacco necrosis satellite virus
- Familia Sarthroviridae
- Macronovirus – Macrobrachium satellite virus 1 (extra virus pequeño)
- (nombre del género) – Nilaparvata lugens commensal X virus
- (nombre del género) – Satélite de parálisis crónica
- (familia sin nombre)
- Virus de ADN de doble tirada
- Familia Lavidaviridae – Virofages
- Satélite de ADN de una sola tirada
- Genus Dependoparvovirus – Grupo de virus asociado con Adeno
- Virus de los satélites de ARN monoplazados
- Ácidos nucleicos satelitales
- ADN de satélites de una sola tirada
- Familia Alphasatellitidae (encoding a replication initiator protein)
- Familia Tolecusatellitidae (encoding a pathogenicity determinante βC1)
- ARN satelital doble
- ARN satelital monoplaza
- Subgrupo 1: Grandes ARN satelitales
- Subgrupo 2: pequeños ARN satelitales lineales
- Subgrupo 3: ARNs satelitales circulares (virusoides)
- Genus Deltavirus
- ARNs asociados con Polerovirus
- ARN tipo satélite
- ADN como satélite
- ADN de satélites de una sola tirada
Partículas de interferencia defectuosas
Las partículas de interferencia defectuosas son virus defectuosos que han perdido su capacidad de replicación excepto en presencia de un virus auxiliar, que normalmente es el virus original. También pueden interferir con el virus auxiliar.
- Partículas interferentes defectuosas (RNA)
- Partículas interferentes defectuosas (ADN)
Contenido relacionado
Ciencia del suelo
Río Tigre
Cernícalo vulgar