Catálisis enzimática
La catálisis enzimática es el aumento de la velocidad de un proceso por una "enzima", una molécula biológica. La mayoría de las enzimas son proteínas y la mayoría de estos procesos son reacciones químicas. Dentro de la enzima, la catálisis generalmente ocurre en un sitio localizado, llamado sitio activo.
La mayoría de las enzimas están formadas predominantemente por proteínas, ya sea una sola cadena proteica o muchas cadenas de este tipo en un complejo de múltiples subunidades. Las enzimas a menudo también incorporan componentes no proteicos, como iones metálicos o moléculas orgánicas especializadas conocidas como cofactores (p. ej., trifosfato de adenosina). Muchos cofactores son vitaminas, y su papel como vitaminas está directamente relacionado con su uso en la catálisis de procesos biológicos dentro del metabolismo. La catálisis de reacciones bioquímicas en la célula es vital, ya que muchas reacciones metabólicamente esenciales, pero no todas, tienen tasas muy bajas cuando no están catalizadas. Un factor impulsor de la evolución de las proteínas es la optimización de dichas actividades catalíticas, aunque solo las enzimas más cruciales operan cerca de los límites de eficiencia catalítica, y muchas enzimas están lejos de ser óptimas. Los factores importantes en la catálisis enzimática incluyen la catálisis general de ácidos y bases, la dirección orbital, la restricción entrópica, los efectos de orientación (es decir, la catálisis de cerradura y llave), así como los efectos de movimiento que involucran la dinámica de las proteínas.
Los mecanismos de catálisis enzimática varían, pero todos son similares en principio a otros tipos de catálisis química en el sentido de que el factor crucial es una reducción de la(s) barrera(s) energética(s) que separan los reactivos (o sustratos) de los productos. La reducción de la energía de activación (Ea) aumenta la fracción de moléculas reactivas que pueden superar esta barrera y formar el producto. Un principio importante es que, dado que solo reducen las barreras energéticas entre productos y reactivos, las enzimas siempre catalizan reacciones en ambas direcciones y no pueden impulsar una reacción hacia adelante ni afectar la posición de equilibrio, solo la velocidad con la que se alcanza. Al igual que con otros catalizadores, la enzima no se consume ni se modifica por la reacción (como ocurre con un sustrato), sino que se recicla de manera que una sola enzima realiza muchas rondas de catálisis.
Las enzimas suelen ser muy específicas y actúan solo sobre determinados sustratos. Algunas enzimas son absolutamente específicas, es decir, actúan solo sobre un sustrato, mientras que otras muestran especificidad de grupo y pueden actuar sobre grupos químicos similares pero no idénticos, como el enlace peptídico en diferentes moléculas. Muchas enzimas tienen especificidad estereoquímica y actúan sobre un estereoisómero pero no sobre otro.
Induced fit
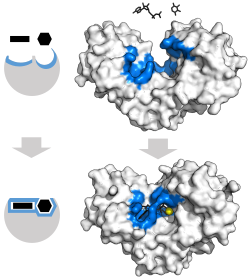
El modelo clásico para la interacción enzima-sustrato es el modelo de ajuste inducido. Este modelo propone que la interacción inicial entre enzima y sustrato es relativamente débil, pero que estas interacciones débiles inducen rápidamente cambios conformacionales en la enzima que fortalecen la unión.
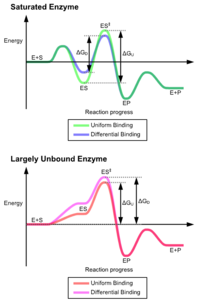
Las ventajas del mecanismo de ajuste inducido surgen debido al efecto estabilizador de la fuerte unión de la enzima. Existen dos mecanismos diferentes de unión del sustrato: la unión uniforme, que tiene una fuerte unión del sustrato, y la unión diferencial, que tiene una fuerte unión del estado de transición. El efecto estabilizador de la unión uniforme aumenta tanto la afinidad de unión del sustrato como del estado de transición, mientras que la unión diferencial aumenta solo la afinidad de unión del estado de transición. Ambos son utilizados por las enzimas y han sido elegidos evolutivamente para minimizar la energía de activación de la reacción. Las enzimas que están saturadas, es decir, que tienen una alta afinidad de unión del sustrato, requieren una unión diferencial para reducir la energía de activación, mientras que las enzimas pequeñas que no se unen al sustrato pueden utilizar una unión diferencial o uniforme.
Estos efectos han llevado a que la mayoría de las proteínas utilicen el mecanismo de unión diferencial para reducir la energía de activación, por lo que la mayoría de los sustratos tienen una alta afinidad por la enzima mientras se encuentran en el estado de transición. La unión diferencial se lleva a cabo mediante el mecanismo de ajuste inducido: el sustrato primero se une débilmente, luego la enzima cambia de conformación aumentando la afinidad por el estado de transición y estabilizándolo, reduciendo así la energía de activación para alcanzarlo.
Es importante aclarar, sin embargo, que el concepto de ajuste inducido no puede utilizarse para racionalizar la catálisis. Es decir, la catálisis química se define como la reducción de Ea‡ (cuando el sistema ya está en el ES‡) en relación con Ea‡ en la reacción no catalizada en agua (sin la enzima). El ajuste inducido sólo sugiere que la barrera es menor en la forma cerrada de la enzima, pero no nos dice cuál es la razón de la reducción de la barrera.
El ajuste inducido puede ser beneficioso para la fidelidad del reconocimiento molecular en presencia de competencia y ruido a través del mecanismo de corrección conformacional.
Mecanismos de una ruta de reacción alternativa
Estos cambios conformacionales también acercan los residuos catalíticos del sitio activo a los enlaces químicos del sustrato que se modificarán en la reacción. Una vez que se produce la unión, uno o más mecanismos de catálisis reducen la energía del estado de transición de la reacción, proporcionando una vía química alternativa para la reacción. Hay seis mecanismos posibles de catálisis "por encima de la barrera", así como un mecanismo "a través de la barrera":
Proximidad y orientación
Las interacciones enzima-sustrato alinean los grupos químicos reactivos y los mantienen juntos en una geometría óptima, lo que aumenta la velocidad de la reacción. Esto reduce la entropía de los reactivos y, por lo tanto, hace que las reacciones de adición o transferencia sean menos desfavorables, ya que una reducción en la entropía general cuando dos reactivos se convierten en un solo producto. Sin embargo, este es un efecto general y se ve en reacciones de no adición o transferencia donde ocurre debido a un aumento en la "concentración efectiva" de los reactivos. Esto se entiende al considerar cómo los aumentos en la concentración conducen a aumentos en la velocidad de reacción: esencialmente, cuando los reactivos están más concentrados, chocan más a menudo y, por lo tanto, reaccionan más a menudo. En la catálisis enzimática, la unión de los reactivos a la enzima restringe el espacio conformacional de los reactivos, manteniéndolos en la "orientación adecuada" y cerca uno del otro, de modo que chocan con más frecuencia y con la geometría correcta, para facilitar la reacción deseada. La "concentración efectiva" es la concentración que debería tener el reactivo, libre en solución, para experimentar la misma frecuencia de colisión. A menudo, estas concentraciones efectivas teóricas no son físicas y son imposibles de lograr en la realidad, lo que es un testimonio del gran poder catalítico de muchas enzimas, con aumentos masivos de velocidad en comparación con el estado no catalizado.
Sin embargo, la situación podría ser más compleja, ya que los estudios computacionales modernos han establecido que los ejemplos tradicionales de efectos de proximidad no pueden relacionarse directamente con los efectos entrópicos de las enzimas. Además, se ha descubierto que la propuesta entrópica original sobrestima en gran medida la contribución de la entropía de orientación a la catálisis.
Proton donors or acceptors
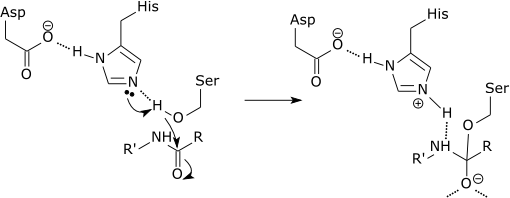
Los donantes y aceptores de protones, es decir, los ácidos y las bases, pueden donar y aceptar protones para estabilizar las cargas en desarrollo en el estado de transición. Esto está relacionado con el principio general de la catálisis, el de reducir las barreras energéticas, ya que en general los estados de transición son estados de alta energía y al estabilizarlos se reduce esta alta energía, bajando la barrera. Una característica clave de la catálisis enzimática en comparación con muchas catálisis no biológicas es que tanto la catálisis ácida como la básica se pueden combinar en la misma reacción. En muchos sistemas abióticos, los ácidos (grandes [H+]) o las bases (grandes sumideros de H+ o especies con pares de electrones) pueden aumentar la velocidad de la reacción; pero, por supuesto, el entorno solo puede tener un pH general (medida de acidez o basicidad [alcalinidad]). Sin embargo, dado que las enzimas son moléculas grandes, pueden colocar tanto grupos ácidos como grupos básicos en su sitio activo para interactuar con sus sustratos y emplear ambos modos independientemente del pH en masa.
A menudo se emplea la catálisis ácida o básica general para activar grupos nucleófilos y/o electrófilos, o para estabilizar grupos salientes. Muchos aminoácidos con grupos ácidos o básicos se emplean en este sitio activo, como el ácido glutámico y aspártico, la histidina, la cistina, la tirosina, la lisina y la arginina, así como la serina y la treonina. Además, a menudo se emplea la cadena principal peptídica, con grupos N carbonilo y amida. La cistina y la histidina están muy comúnmente involucradas, ya que ambas tienen un pKa cercano al pH neutro y, por lo tanto, pueden aceptar y donar protones.
Muchos mecanismos de reacción que implican catálisis ácido-base suponen un pKa sustancialmente alterado. Esta alteración del pKa es posible gracias al entorno local del residuo.
| Condiciones | Ácidos | Bases |
|---|---|---|
| Entorno hidrofóbico | Incremento pKa | Disminuir pKa |
| Residuos adyacentes de carga similar | Incremento pKa | Disminuir pKa |
| Puente de sal (y hidrógeno) unión) formación | Disminuir pKa | Incremento pKa |
El pKa también puede verse afectado significativamente por el entorno circundante, hasta el punto de que los residuos que son básicos en solución pueden actuar como donadores de protones, y viceversa.
La modificación de los pKa es una parte pura del mecanismo electrostático. El efecto catalítico del ejemplo anterior está asociado principalmente con la reducción del pKa del oxianión y el aumento del pKa de la histidina, mientras que la transferencia de protones de la serina a la histidina no se cataliza de manera significativa, ya que no es la barrera que determina la velocidad. Nótese que en el ejemplo mostrado, el ácido conjugado de la histidina actúa como un catalizador ácido general para la pérdida posterior de la amina de un intermediario tetraédrico. Sin embargo, la evidencia que apoya este mecanismo propuesto (Figura 4 en Ref. 13) ha sido controvertida.
Catálisis electrostática
La estabilización de los estados de transición cargados también puede realizarse mediante residuos en el sitio activo que forman enlaces iónicos (o interacciones de carga iónica parcial) con el intermediario. Estos enlaces pueden provenir de cadenas laterales ácidas o básicas que se encuentran en aminoácidos como la lisina, la arginina, el ácido aspártico o el ácido glutámico, o pueden provenir de cofactores metálicos como el zinc. Los iones metálicos son particularmente eficaces y pueden reducir el pKa del agua lo suficiente como para convertirla en un nucleófilo eficaz.
Estudios sistemáticos de simulación por ordenador han demostrado que los efectos electrostáticos son los que más contribuyen a la catálisis, ya que pueden aumentar la velocidad de reacción hasta en un factor de 107. En particular, se ha descubierto que la enzima proporciona un entorno más polar que el agua y que los estados de transición iónicos se estabilizan mediante dipolos fijos. Esto es muy diferente de la estabilización del estado de transición en el agua, donde las moléculas de agua deben pagar con "energía de reorganización" para estabilizar los estados iónicos y cargados. Por tanto, la catálisis está asociada al hecho de que los grupos polares de la enzima están preorganizados.
Se ha demostrado que la magnitud del campo electrostático ejercido por el sitio activo de una enzima está altamente correlacionada con el aumento de la tasa catalítica de la enzima.
La unión del sustrato generalmente excluye el agua del sitio activo, lo que reduce la constante dieléctrica local a la de un solvente orgánico. Esto fortalece las interacciones electrostáticas entre los sustratos cargados/polares y los sitios activos. Además, los estudios han demostrado que las distribuciones de carga alrededor de los sitios activos están organizadas de manera que estabilizan los estados de transición de las reacciones catalizadas. En varias enzimas, estas distribuciones de carga aparentemente sirven para guiar a los sustratos polares hacia sus sitios de unión de modo que las velocidades de estas reacciones enzimáticas sean mayores que sus límites aparentes controlados por difusión.
| Por ejemplo: |
| Mecanismo catalítico de Carboxypeptidase |
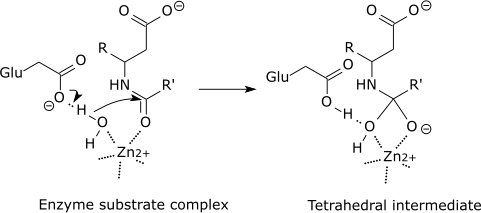 |
| El intermedio tetraedral está estabilizado por un vínculo iónico parcial entre el Zn2+ ion y la carga negativa en el oxígeno. |
Catálisis covalente
La catálisis covalente implica que el sustrato forme un enlace covalente transitorio con residuos en el sitio activo de la enzima o con un cofactor. Esto añade un intermediario covalente adicional a la reacción y ayuda a reducir la energía de los estados de transición posteriores de la reacción. El enlace covalente debe romperse en una etapa posterior de la reacción para regenerar la enzima. Este mecanismo es utilizado por la tríada catalítica de enzimas como las proteasas como la quimotripsina y la tripsina, donde se forma un intermediario acil-enzima. Un mecanismo alternativo es la formación de la base de Schiff utilizando la amina libre de un residuo de lisina, como se observa en la enzima aldolasa durante la glucólisis.
Algunas enzimas utilizan cofactores que no son aminoácidos, como el fosfato de piridoxal (PLP) o el pirofosfato de tiamina (TPP), para formar intermediarios covalentes con moléculas reactivas. Estos intermediarios covalentes funcionan para reducir la energía de los estados de transición posteriores, de manera similar a cómo los intermediarios covalentes formados con residuos de aminoácidos del sitio activo permiten la estabilización, pero las capacidades de los cofactores permiten a las enzimas llevar a cabo reacciones que los residuos de aminoácidos por sí solos no podrían. Las enzimas que utilizan estos cofactores incluyen la enzima aspartato transaminasa dependiente de PLP y la enzima piruvato deshidrogenasa dependiente de TPP.
En lugar de reducir la energía de activación de una vía de reacción, la catálisis covalente proporciona una vía alternativa para la reacción (a través del intermediario covalente) y, por lo tanto, es distinta de la catálisis verdadera. Por ejemplo, la energía del enlace covalente a la molécula de serina en la quimotripsina debe compararse con el enlace covalente bien conocido al nucleófilo en la reacción en solución no catalizada. Una verdadera propuesta de catálisis covalente (donde la barrera es menor que la barrera correspondiente en solución) requeriría, por ejemplo, un enlace covalente parcial al estado de transición por parte de un grupo enzimático (por ejemplo, un enlace de hidrógeno muy fuerte), y tales efectos no contribuyen significativamente a la catálisis.
Catálisis de iones de metal
Un ion metálico en el sitio activo participa en la catálisis coordinando la estabilización y el apantallamiento de la carga. Debido a la carga positiva de un metal, solo las cargas negativas pueden estabilizarse a través de iones metálicos. Sin embargo, los iones metálicos son ventajosos en la catálisis biológica porque no se ven afectados por los cambios de pH. Los iones metálicos también pueden actuar para ionizar el agua al actuar como un ácido de Lewis. Los iones metálicos también pueden ser agentes de oxidación y reducción.
Tensión ósea
Este es el principal efecto de la unión por ajuste inducido, en el que la afinidad de la enzima por el estado de transición es mayor que por el propio sustrato. Esto induce reordenamientos estructurales que tensan los enlaces del sustrato hacia una posición más cercana a la conformación del estado de transición, lo que reduce la diferencia de energía entre el sustrato y el estado de transición y ayuda a catalizar la reacción.
Sin embargo, el efecto de la tensión es, de hecho, un efecto de desestabilización del estado fundamental, en lugar de un efecto de estabilización del estado de transición. Además, las enzimas son muy flexibles y no pueden aplicar un gran efecto de tensión.
Además de la tensión de enlace en el sustrato, también se puede inducir una tensión de enlace dentro de la propia enzima para activar residuos en el sitio activo.
Tunelaje cuántico
Estos mecanismos tradicionales de "superación de la barrera" han sido cuestionados en algunos casos por modelos y observaciones de mecanismos de "a través de la barrera" (efecto túnel cuántico). Algunas enzimas operan con cinéticas que son más rápidas que las que se predecirían con los modelos ΔG‡ clásicos. En los modelos de "a través de la barrera", un protón o un electrón pueden atravesar barreras de activación mediante un efecto túnel. Se ha observado un efecto túnel cuántico para protones en la oxidación de triptamina por la amina aromática deshidrogenasa.
La tunelización cuántica no parece proporcionar una ventaja catalítica importante, ya que las contribuciones de la tunelización son similares en las reacciones catalizadas y no catalizadas en solución. Sin embargo, la contribución de la tunelización (que normalmente mejora las constantes de velocidad en un factor de ~1000 en comparación con la velocidad de reacción para la ruta clásica "sobre la barrera") es probablemente crucial para la viabilidad de los organismos biológicos. Esto enfatiza la importancia general de las reacciones de tunelización en biología.
En 1971-1972 se formuló el primer modelo mecánico cuántico de catálisis enzimática.
Enzima activa
La energía de enlace del complejo enzima-sustrato no puede considerarse como una energía externa necesaria para la activación del sustrato. La enzima con alto contenido energético puede transferir primero un grupo energético específico X1 desde el sitio catalítico de la enzima al lugar final del primer reactivo enlazado, luego otro grupo X2 desde el segundo reactivo enlazado (o desde el segundo grupo del reactivo único) debe transferirse al sitio activo para finalizar la conversión del sustrato en producto y la regeneración de la enzima.
Podemos presentar toda la reacción enzimática como dos reacciones de acoplamiento:
| S1 + EX1 → S1EX1 → P1 + EP2 | ()1) |
| S2 + EP2 → S2EP2 → P2 + EX2 | ()2) |
De la reacción (1) se desprende que el grupo X1 de la enzima activa aparece en el producto debido a la posibilidad de que la reacción de intercambio se produzca dentro de la enzima para evitar tanto la inhibición electrostática como la repulsión de átomos. Por lo tanto, representamos la enzima activa como un reactivo potente de la reacción enzimática. La reacción (2) muestra una conversión incompleta del sustrato porque su grupo X2 permanece dentro de la enzima. Esta idea se había propuesto anteriormente basándose en las conversiones enzimáticas extremadamente altas hipotéticas (enzima catalíticamente perfecta).
El punto crucial para la verificación del presente enfoque es que el catalizador debe ser un complejo de la enzima con el grupo de transferencia de la reacción. Este aspecto químico está respaldado por los mecanismos bien estudiados de las diversas reacciones enzimáticas. Considérese la reacción de hidrólisis del enlace peptídico catalizada por una proteína pura, la α-quimotripsina (una enzima que actúa sin un cofactor), que es un miembro bien estudiado de la familia de las serina proteasas, ver.
Presentamos los resultados experimentales de esta reacción en dos pasos químicos:
| S1 + EH → P1 + EP2 | ()3) |
| EP2 + H−O−H → EH + P2 | ()4) |
donde S1 es un polipéptido, P1 y P2 son productos. El primer paso químico (3) incluye la formación de un intermediario covalente acilo-enzima. El segundo paso (4) es el paso de desacilación. Es importante notar que el grupo H+, inicialmente encontrado en la enzima, pero no en el agua, aparece en el producto antes del paso de hidrólisis, por lo tanto puede ser considerado como un grupo adicional de la reacción enzimática.
Así, la reacción (3) muestra que la enzima actúa como un potente reactante de la reacción. Según el concepto propuesto, el transporte de H desde la enzima promueve la conversión del primer reactante, la ruptura del primer enlace químico inicial (entre los grupos P1 y P2). El paso de hidrólisis conduce a una ruptura del segundo enlace químico y a la regeneración de la enzima.
El mecanismo químico propuesto no depende de la concentración de los sustratos o productos en el medio. Sin embargo, un cambio en su concentración provoca principalmente cambios de energía libre en los pasos inicial y final de las reacciones (1) y (2) debido a los cambios en el contenido de energía libre de cada molécula, ya sea S o P, en solución acuosa. Este enfoque está de acuerdo con el siguiente mecanismo de contracción muscular. El paso final de la hidrólisis de ATP en el músculo esquelético es la liberación del producto causada por la asociación de las cabezas de miosina con la actina. El cierre de la hendidura de unión de la actina durante la reacción de asociación está estructuralmente acoplado con la apertura del bolsillo de unión de nucleótidos en el sitio activo de la miosina.
Cabe destacar que los pasos finales de la hidrólisis del ATP incluyen la liberación rápida de fosfato y la liberación lenta de ADP. La liberación de un anión fosfato a partir del anión ADP unido a una solución acuosa puede considerarse una reacción exergónica porque el anión fosfato tiene una masa molecular baja.
Por lo tanto, llegamos a la conclusión de que la liberación primaria del fosfato inorgánico H2PO4− conduce a la transformación de una parte significativa de la energía libre de la hidrólisis del ATP en energía cinética del fosfato solvatado, lo que produce un flujo activo. Esta suposición de una transducción mecanoquímica local concuerda con el mecanismo de contracción muscular de Tirosh, donde la fuerza muscular deriva de una acción integrada de flujo activo creado por la hidrólisis del ATP.
Ejemplos de mecanismos catalíticos
En realidad, la mayoría de los mecanismos enzimáticos implican una combinación de varios tipos diferentes de catálisis.
Triose phosphate isomerase
La triosa fosfato isomerasa (EC 5.3.1.1) cataliza la interconversión reversible de los dos isómeros de triosa fosfato, dihidroxiacetona fosfato y D-gliceraldehído 3-fosfato.
Trypsin
La tripsina (EC 3.4.21.4) es una serina proteasa que escinde los sustratos proteicos después de los residuos de lisina o arginina utilizando una tríada catalítica para realizar la catálisis covalente y un agujero de oxianión para estabilizar la acumulación de carga en los estados de transición.
Aldolase
La aldolasa (EC 4.1.2.13) cataliza la descomposición de la fructosa 1,6-bisfosfato (F-1,6-BP) en gliceraldehído 3-fosfato y dihidroxiacetona fosfato (DHAP).
Difusividad enzimática
La aparición de estudios de moléculas individuales en la década de 2010 condujo a la observación de que el movimiento de enzimas no ligadas aumenta con el aumento de la concentración de sustrato y el aumento de la entalpía de reacción. Observaciones posteriores sugieren que este aumento de la difusividad está impulsado por el desplazamiento transitorio del centro de masa de la enzima, lo que da como resultado un "efecto de retroceso que impulsa a la enzima".
Semejanza de reacción
La similitud entre reacciones enzimáticas (EC) se puede calcular utilizando cambios de enlaces, centros de reacción o métricas de subestructura (EC-BLAST Archivado el 30 de mayo de 2019 en Wayback Machine).
Véase también
- Triada catalítica
- Enzyme assay
- Inhibidor enzima
- Enzyme kinetics
- Enzyme promiscuity
- Dinámica de proteínas
- Pseudoenzimas, cuya ubicuidad a pesar de su inactividad catalítica sugiere implicaciones omicas
- Tunelaje cuántico
- Mapa de Proteolisis
- Cristalografía resuelta
Referencias
- ^ Kamerlin SC, Warshel A (mayo de 2010). "En el amanecer del siglo XXI: ¿Es dinámica el eslabón perdido para entender la catalisis de enzimas?". Proteínas. 78 (6): 1339–1375. doi:10.1002/prot.22654. PMC 2841229. PMID 20099310.
- ^ Laidler KJ (1978). Química Física con Aplicaciones Biológicas. Benjamin/Cummings. p. 427. ISBN 978-0-8053-5680-9.
- ^ Koshland DE (febrero de 1958). "Aplicación de una teoría de la especificación enzimática a la síntesis de proteínas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 44 (2): 98–104. Bibcode:1958PNAS...44...98K doi:10.1073/pnas.44.2.98. PMC 335371. PMID 16590179.

- ^ Anslyn EV, Dougherty DA (2006). Química Orgánica Física Moderna. Libros de Ciencias de la Universidad. ISBN 978-1-891389-31-3.
- ^ Savir Y, Tlusty T (mayo de 2007). Scalas E (ed.). "La corrección de pruebas conformacionales: el impacto de los cambios conformacionales en la especificidad del reconocimiento molecular". PLOS ONE. 2 (5): e468. Código:2007PLoSO...2..468S. doi:10.1371/journal.pone.0000468. PMC 1868595. PMID 17520027.

- ^ Stanton RV, Peräkylä M, Bakowies D, Kollman PA (1998). "Combined ab initio and Free Energy Calculations To Study Reactions in Enzymes and Solution: Amide Hydrolysis in Trypsin and Aqueous Solution". J. Am. Chem. Soc. 120 (14): 3448–3457. doi:10.1021/ja972723x.
- ^ Kuhn B, Kollman PA (2000). "QM-FE y Dinámica Molecular Cálculos sobre Catechol O-Methyltransferase: Energía Libre de Activación en la Enzima y en Solución Acuosa y Regioselectividad de la Reacción Caatalyzed Enzyme". J. Am. Chem. Soc. 122 (11): 2586–2596. doi:10.1021/ja992218v.
- ^ Bruice TC, Lightstone FC (1999). "Ground State and Transition State Contributions to the Rates of Intramolecular and Enzymatic Reactions". Acc. Chem. Res. 32 (2): 127–136. doi:10.1021/ar960131y.
- ^ Página MI, Jencks WP (agosto 1971). "Contribuciones metrópicas para acelerar las velocidades en las reacciones enzimáticas e intramoleculares y el efecto chelato". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 68 (8): 1678-1683. Bibcode:1971PNAS...68.1678P. doi:10.1073/pnas.68.8.1678. PMC 389269. PMID 5288752.
- ^ Warshel A, Parson WW (noviembre de 2001). "Dinamics of biochemical and biophysical reactions: insight from computer simulations". Reseñas trimestrales de la biofísica. 34 (4): 563–679. doi:10.1017/s0033583501003730. PMID 11852595. S2CID 28961992.
- ^ a b c d Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH (agosto de 2006). "Base electrostática para la catalisis de enzimas". Reseñas químicas. 106 (8): 3210–3235. doi:10.1021/cr0503106. PMID 16895325.
- ^ Warshel A, Naray-Szabo G, Sussman F, Hwang JK (mayo de 1989). "¿Cómo funcionan realmente las proteasas serinas?". Bioquímica. 28 (9): 3629–3637. doi:10.1021/bi00435a001. PMID 2665806.
- ^ Fersht AR, Requena Y (diciembre de 1971). "Mecanismo de la hidrólisis de chymotrypsin-catalyzed de amides. dependencia de k c y K m. Detección cinética de un intermediario". Journal of the American Chemical Society. 93 (25): 7079–7087. doi:10.1021/ja00754a066. PMID 5133099.
- ^ Zeeberg B, Caswell M, Caplow M (abril de 1973). "Concertando un cambio reportado en el paso de determinación de tarifas en la catalisis de quimotrypsin". Journal of the American Chemical Society. 95 (8): 2734–2735. doi:10.1021/ja00789a081. PMID 4694533.
- ^ Voet D, Voet JG (2011). Bioquímica. John Wiley & Sons. OCLC 808679090.
- ^ Marcus RA (1965). "En la Teoría de Reacciones Electron-Transfer. VI. Tratamiento unificado para las reacciones homogéneas y electrode" (PDF). J. Chem. Phys. 43 (2): 679–701. Bibcode:1965JChPh..43..679M. doi:10.1063/1.1696792.
- ^ Warshel A (noviembre de 1978). "Energética de catalisis de enzimas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 75 (11): 5250–5254. Bibcode:1978PNAS...75.5250W doi:10.1073/pnas.75.11.5250. PMC 392938. PMID 281676.
- ^ Fried SD, Bagchi S, Boxer SG (diciembre de 2014). "Extreme electric fields catalysis in the active site of ketosteroid isomerase". Ciencia. 346 (6216). Nueva York, N.Y.: 1510-4. Bibcode:2014Sci...346.1510F doi:10.1126/ciencia.1259802. PMC 4668018. PMID 255245.
- ^ Toney, M. D. "Especificidad de reacción en enzimas piridoxales". Archivos de bioquímica y biofísica (2005) 433: 279-287
- ^ "Micronutrient Information Center, Oregon State University". Archivado desde el original el 21 de marzo de 2015. Retrieved 30 de septiembre 2009.
- ^ Voet D, Voet JG (2004). Bioquímica. John Wiley ' Sons Inc. pp. 986–989. ISBN 978-0-471-25090-6.
- ^ Voet D, Voet JG (2004). Bioquímica. John Wiley ' Sons Inc. pp. 604–606. ISBN 978-0-471-25090-6.
- ^ Piccirilli JA, Vyle JS, Caruthers MH, Cech TR (enero de 1993). "Catálisis de iones metálicos en la reacción de ribozyme Tetrahymena". Naturaleza. 361 (6407): 85–88. código:1993Natur.361...85P. doi:10.1038/361085a0. PMID 8421499. S2CID 4326584.
- ^ Bender ML (1 de enero de 1962). "Catálisis Iónica Metal de las Reacciones Orgánicas Nucleofílicas en la Solución". Reacciones de ligandos coordinados. Avances en Química. Vol. 37. American Chemical Society. pp. 19–36. doi:10.1021/ba-1963-0037.ch002. ISBN 978-0-8412-0038-8.
- ^ Fife TH, Przystas TJ (1 de febrero de 1985). "Calálisis de iones metálicos líquidos en la hidrolisis de ésteres de ácido picolínico. El ión metálico promovió las reacciones de iones hidroxidos y catalizantes de agua". Journal of the American Chemical Society. 107 (4): 1041-1047. doi:10.1021/ja00290a048. ISSN 0002-7863.
- ^ Stadtman ER (1o de enero de 1990). "Oxidación metalizada de proteínas: mecanismo bioquímico y consecuencias biológicas". Biología Radical Libre & Medicina. 9 (4): 315–325. doi:10.1016/0891-5849(90)90006-5. PMID 2283087.
- ^ Jencks WP (1987) [1969]. Catalisis en química y enzimología. McGraw-Hill series en química avanzada (reprint ed.). Nueva York: Dover Publications. ISBN 978-0-486-65460-7.
- ^ Warshel A, Levit M (mayo de 1976). "Estudios teóricos de reacciones enzimáticas: estabilización dieléctrica, electrostática y esterica del ión de carbonio en la reacción de la lysozyme". Journal of Molecular Biology. 103 (2): 227–249. doi:10.1016/0022-2836(76)90311-9. PMID 985660.

- ^ Voet D, Voet JG, Pratt CW (2013). Fundamentos de la Bioquímica: Vida a nivel molecular (Cuarta edición). Wiley. ISBN 978-0-470-54784-7.
- ^ García-Viloca M, Gao J, Karplus M, Truhlar DG (enero de 2004). "Cómo funcionan las enzimas: análisis por teoría moderna de tarifas y simulaciones de ordenador". Ciencia. 303 186-195. Bibcode:2004Sci...303..186G. doi:10.1126/science.1088172. PMID 14716003. S2CID 17498715.
- ^ a b Olsson MH, Siegbahn PE, Warshel A (marzo de 2004). "Simulaciones del gran efecto isótopo cinético y la dependencia de temperatura de la transferencia de átomos de hidrógeno en lipoxigenasa". Journal of the American Chemical Society. 126 (9): 2820–2828. doi:10.1021/ja037233l. PMID 14995199.
- ^ a b Masgrau L, Roujeinikova A, Johannissen LO, Hothi P, Basran J, Ranaghan KE, et al. (abril de 2006). "Descripción atómica de una reacción enzimática dominada por el túnel protón". Ciencia. 312 (5771): 237–241. Bibcode:2006...312..237M. doi:10.1126/science.1126002. PMID 16614214. S2CID 27201250.
- ^ Hwang JK, Warshel A (1996). "Cuán importantes son los movimientos nucleares mecánicos cuánticos en la catalisis de enzimas". J. Am. Chem. Soc. 118 (47): 11745–11751. doi:10.1021/ja962007f.
- ^ Ball P (septiembre 2004). "Enzimas: por casualidad, o por diseño?". Naturaleza. 431 (7007): 396–397. Código:2004Natur.431..396B. doi:10.1038/431396a. PMID 15385982. S2CID 228263.
- ^ Olsson MH, Parson WW, Warshel A (mayo de 2006). "Contribuciones Dinámicas a la catalisis de enzimas: pruebas críticas de una hipótesis popular". Reseñas químicas. 106 (5): 1737-1756. doi:10.1021/cr040427e. PMID 16683752.
- ^ Vol'kenshtein MV, Dogonadze RR, Madumarov AK, Urushadze ZD, Kharkats YI (1972). "La teoría de la catalisis de enzimas". Biología molecular. 6 3). Moscú: 347–353. PMID 4645409.
- ^ Volkenshtein MV, Dogonadze RR, Madumarov AK, Urushadze ZD, Kharkats Yu I (1973). "Interacciones electrónicas y conformacionales en la catalisis de Enzyme.". Konformatsionnie Izmenenia Biopolimerov v Rastvorakh. Moscú: Nauka Publishing House. pp. 153–157.
- ^ Foigel AG (junio de 2011). "¿Es la enzima un poderoso reaccionante de la reacción bioquímica?". Bioquímica molecular y celular. 352 (1–2): 87–89. doi:10.1007/s11010-011-0742-4. PMID 21318350. S2CID 11133081.
- ^ Fogel AG (agosto de 1982). "Cooperatividad de reacciones enzimáticas y aspectos moleculares de la transducción energética". Bioquímica molecular y celular. 47 (1): 59–64. doi:10.1007/bf00241567. PMID 7132966. S2CID 21790380.
- ^ Hengge AC, Stein RL (enero de 2004). "Role of protein conformational mobility in enzima catalysis: acylation of alpha-chymotrypsin by specific peptide substrates". Bioquímica. 43 (3): 742-747. doi:10.1021/bi030222k. PMID 14730979.
- ^ Lymn RW, Taylor EW (diciembre de 1971). "Mecanismo de la hidrolisis adenosina triphosphate por la actomyosin". Bioquímica. 10 (25): 4617–4624. doi:10.1021/bi00801a004. PMID 4258719.
- ^ Holmes KC, Angert I, Kull FJ, Jahn W, Schröder RR (septiembre de 2003). "La criomicroscopia electrónica muestra cuán fuerte unión de la miosina para actuar libera nucleótido". Naturaleza. 425 (6956): 423-427. Código:2003Natur.425..423H. doi:10.1038/nature02005. PMID 14508495. S2CID 2686184.
- ^ Siemankowski RF, Wiseman MO, White HD (febrero de 1985). "La disociación ADP del subfragmento de la acciónmyosina 1 es suficientemente lenta para limitar la velocidad de acortamiento descargada en el músculo vertebrado". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 82 (3): 658-662. Bibcode:1985PNAS...82..658S. doi:10.1073/pnas.82.3.658. PMC 397104. PMID 3871943.
- ^ White HD, Belknap B, Webb MR (septiembre de 1997). "Kinetics of nucleoside triphosphate cleavage and phosphate release steps by associated conejo esquelético actomyosin, medido utilizando una nueva sonda fluorescente para fosfato". Bioquímica. 36 (39): 11828–11836. doi:10.1021/bi970540h. PMID 9305974.
- ^ Tirosh R, Low WZ, Oplatka A (marzo de 1990). "Modo de traducción de filamentos de actina en presencia de meromyosina pesada y MgATP medido por Doppler ampliando la dispersión de luz láser". Biochimica et Biophysica Acta (BBA) - Estructura de proteínas y enzimología molecular. 1037 (3): 274–280. doi:10.1016/0167-4838(90)90025-b. PMID 2178685.
- ^ Tirosh R (2006). "Protones balísticos y soluciones de agua inducidas por microondas (solitons) en transformaciones bioenergéticas". Int. J. Mol.. 7 (9): 320-345. doi:10.3390/i7090320.
- ^ Muddana HS, Sengupta S, Mallouk TE, Sen A, Butler PJ (febrero de 2010). "La catalisis substrato mejora la difusión de una sola enzima". Journal of the American Chemical Society. 132 (7): 2110–2111. doi:10.1021/ja908773a. PMC 2832858. PMID 20108965.

- ^ Riedel C, Gabizon R, Wilson CA, Hamadani K, Tsekouras K, Marqusee S, et al. (enero de 2015). "El calor liberado durante la rotación catalítica aumenta la difusión de una enzima". Naturaleza. 517 (7533): 227–230. Código:2015Natur.517..227R. doi:10.1038/nature14043. PMC 4363105. PMID 25487146.

- ^ Rahman SA, Cuesta SM, Furnham N, Holliday GL, Thornton JM (febrero de 2014). "EC-BLAST: una herramienta para buscar y comparar automáticamente las reacciones enzimáticas". Métodos naturales. 11 (2): 171–174. doi:10.1038/nmeth.2803. PMC 4122987. PMID 24412978.
Más lectura
- Fersht A (1998). Estructura y mecanismo en la ciencia de proteínas: una guía para la catalisis de enzimas y plegamiento de proteínas. W.H. Freeman. ISBN 978-0-7167-3268-6.
- Sutcliffe M, Munro A (agosto de 2006). "Catálisis quántica en enzimas -más allá de la teoría estatal de transición". Transacciones filosóficas B. 361 (1472): 1291–1455. doi:10.1098/rstb.2006.1879. PMC 1647302.