Biología estructural
biología estructural es un campo con muchos siglos de antigüedad que, tal como lo define el Journal of Structural Biology, se ocupa del análisis estructural de la materia viva (formada, compuesta y/o mantenida y refinado por células vivas) en cada nivel de organización. Los primeros biólogos estructurales a lo largo del siglo XIX y principios del XX solo pudieron estudiar estructuras hasta el límite de la agudeza visual a simple vista y a través de lupas y microscopios ópticos.
En el siglo XX, se desarrollaron una variedad de técnicas experimentales para examinar las estructuras 3D de las moléculas biológicas. Las técnicas más destacadas son la cristalografía de rayos X, la resonancia magnética nuclear y la microscopía electrónica. A través del descubrimiento de los rayos X y sus aplicaciones a los cristales de proteínas, la biología estructural se revolucionó, ya que ahora los científicos podían obtener las estructuras tridimensionales de las moléculas biológicas en detalle atómico. Asimismo, la espectroscopia de RMN permitió obtener información sobre la estructura y dinámica de las proteínas. Finalmente, en el siglo XXI, la microscopía electrónica también experimentó una revolución drástica con el desarrollo de fuentes de electrones más coherentes, corrección de aberraciones para microscopios electrónicos y software de reconstrucción que permitió la implementación exitosa de microscopía crioelectrónica de alta resolución, lo que permitió el estudio de proteínas individuales y complejos moleculares en tres dimensiones con una resolución de angstrom.
Con el desarrollo de estas tres técnicas, el campo de la biología estructural se expandió y también se convirtió en una rama de la biología molecular, la bioquímica y la biofísica que se ocupa de la estructura molecular de las macromoléculas biológicas (especialmente las proteínas, compuestas por aminoácidos, ARN o ADN, formadas por formado por nucleótidos, y membranas, formadas por lípidos), cómo adquieren las estructuras que tienen, y cómo las alteraciones en sus estructuras afectan a su función. Este tema es de gran interés para los biólogos porque las macromoléculas realizan la mayoría de las funciones de las células, y solo al enrollarse en formas tridimensionales específicas pueden realizar estas funciones. Esta arquitectura, la "estructura terciaria" de moléculas, depende de manera complicada de la composición básica de cada molécula, o 'estructura primaria'. A resoluciones más bajas, herramientas como la tomografía FIB-SEM han permitido una mayor comprensión de las células y sus orgánulos en 3 dimensiones, y cómo cada nivel jerárquico de varias matrices extracelulares contribuye a la función (por ejemplo, en el hueso). En los últimos años también se ha hecho posible predecir modelos moleculares físicos de alta precisión para complementar el estudio experimental de estructuras biológicas. Las técnicas computacionales, como las simulaciones de dinámica molecular, se pueden utilizar junto con estrategias de determinación de estructura empírica para ampliar y estudiar la estructura, la conformación y la función de las proteínas.
Historia
En 1912, Max Von Laue dirigió rayos X al sulfato de cobre cristalizado generando un patrón de difracción. Estos experimentos llevaron al desarrollo de la cristalografía de rayos X y su uso en la exploración de estructuras biológicas. En 1951, Rosalind Franklin y Maurice Wilkins usaron patrones de difracción de rayos X para capturar la primera imagen del ácido desoxirribonucleico (ADN). Francis Crick y James Watson modelaron la estructura de doble hélice del ADN utilizando esta misma técnica en 1953 y recibieron el Premio Nobel de Medicina junto con Wilkins en 1962.
Los cristales de pepsina fueron las primeras proteínas que se cristalizaron para su uso en la difracción de rayos X, por Theodore Svedberg, quien recibió el Premio Nobel de Química en 1962. La primera estructura de proteína terciaria, la de la mioglobina, fue publicada en 1958 por John Kendrew. Durante este tiempo, el modelado de estructuras de proteínas se realizó utilizando modelos de madera de balsa o alambre. Con la invención del software de modelado como CCP4 a fines de la década de 1970, el modelado ahora se realiza con la ayuda de una computadora. Los desarrollos recientes en el campo han incluido la generación de láseres de electrones libres de rayos X, lo que permite el análisis de la dinámica y el movimiento de las moléculas biológicas, y el uso de la biología estructural para ayudar a la biología sintética.
A finales de la década de 1930 y principios de la de 1940, la combinación del trabajo realizado por Isidor Rabi, Felix Bloch y Edward Mills Purcell condujo al desarrollo de la resonancia magnética nuclear (RMN). Actualmente, la RMN de estado sólido se usa ampliamente en el campo de la biología estructural para determinar la estructura y la naturaleza dinámica de las proteínas (proteína RMN).
En 1990, Richard Henderson produjo la primera imagen tridimensional de alta resolución de la bacteriorrodopsina mediante microscopía electrónica criogénica (crio-EM). Desde entonces, la crio-EM se ha convertido en una técnica cada vez más popular para determinar estructuras tridimensionales de alta resolución de imágenes biológicas.
Más recientemente, se han desarrollado métodos computacionales para modelar y estudiar estructuras biológicas. Por ejemplo, la dinámica molecular (MD) se usa comúnmente para analizar los movimientos dinámicos de las moléculas biológicas. En 1975, se publicó en Nature la primera simulación de un proceso de plegamiento biológico usando MD. Recientemente, la predicción de la estructura de la proteína se mejoró significativamente mediante un nuevo método de aprendizaje automático llamado AlphaFold. Algunos afirman que los enfoques computacionales están comenzando a liderar el campo de la investigación en biología estructural.
Técnicas
Las biomoléculas son demasiado pequeñas para verlas en detalle, incluso con los microscopios ópticos más avanzados. Los métodos que utilizan los biólogos estructurales para determinar sus estructuras generalmente implican mediciones en un gran número de moléculas idénticas al mismo tiempo. Estos métodos incluyen:
- Espectrometría masiva
- Cristalografía macromolecular
- Difracción de neutrones
- Proteolisis
- Espectroscopia de resonancia magnética nuclear de proteínas (NMR)
- Resonancia paramagnética electrones (EPR)
- Microscopia de electrones criogénicos (cryoEM)
- Cristalografía de electrones y difracción de electrones Microcrystal
- Multiángulo de la luz dispersa
- Difusión de ángulo pequeño
- Espectroscopia láser ultrarrápida
- Anisotropic terahertz microspectroscopia
- Espectroscopia infrarroja bidimensional
- Interferometría de doble polarización y dicroismo circular
La mayoría de las veces, los investigadores los usan para estudiar los "estados nativos" de macromoléculas. Pero también se utilizan variaciones de estos métodos para observar cómo las moléculas nacientes o desnaturalizadas asumen o reasumen sus estados nativos. Ver plegamiento de proteínas.
Un tercer enfoque que adoptan los biólogos estructurales para comprender la estructura es la bioinformática para buscar patrones entre las diversas secuencias que dan lugar a formas particulares. Los investigadores a menudo pueden deducir aspectos de la estructura de las proteínas integrales de la membrana en función de la topología de la membrana predicha por el análisis de hidrofobicidad. Ver predicción de la estructura de la proteína.
Aplicaciones
Los biólogos estructurales han hecho contribuciones significativas para comprender los componentes y mecanismos moleculares que subyacen a las enfermedades humanas. Por ejemplo, se han utilizado crio-EM y ssNMR para estudiar la agregación de fibrillas de amiloide, que están asociadas con la enfermedad de Alzheimer, la enfermedad de Parkinson y la diabetes tipo II. Además de las proteínas amiloides, los científicos han utilizado crio-EM para producir modelos de alta resolución de filamentos de tau en el cerebro de pacientes con Alzheimer, lo que puede ayudar a desarrollar mejores tratamientos en el futuro. Las herramientas de biología estructural también se pueden utilizar para explicar las interacciones entre patógenos y huéspedes. Por ejemplo, las herramientas de biología estructural han permitido a los virólogos comprender cómo la envoltura del VIH permite que el virus evada las respuestas inmunitarias humanas.
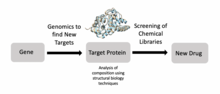
La biología estructural también es un componente importante del descubrimiento de fármacos. Los científicos pueden identificar objetivos utilizando la genómica, estudiar esos objetivos utilizando la biología estructural y desarrollar fármacos que sean adecuados para esos objetivos. Específicamente, la RMN de ligando, la espectrometría de masas y la cristalografía de rayos X son técnicas comúnmente utilizadas en el proceso de descubrimiento de fármacos. Por ejemplo, los investigadores han utilizado la biología estructural para comprender mejor a Met, que es un importante objetivo farmacológico para el cáncer. Se han realizado investigaciones similares para objetivos de VIH para tratar a personas con SIDA. Los investigadores también están desarrollando nuevos antimicrobianos para las infecciones por micobacterias mediante el descubrimiento de fármacos basados en la estructura.
Contenido relacionado
Collie de la frontera
Tarbosaurio
Pan (género)