Biología Computacional
Biología computacional se refiere al uso de análisis de datos, modelos matemáticos y simulaciones computacionales para comprender los sistemas y las relaciones biológicas. Una intersección de informática, biología y big data, el campo también tiene fundamentos en matemáticas aplicadas, química y genética. Se diferencia de la computación biológica, un subcampo de la ingeniería informática que utiliza la bioingeniería para construir computadoras.
Historia
La bioinformática, el análisis de procesos informáticos en sistemas biológicos, comenzó a principios de la década de 1970. En ese momento, la investigación en inteligencia artificial utilizaba modelos de red del cerebro humano para generar nuevos algoritmos. Este uso de datos biológicos empujó a los investigadores biológicos a usar computadoras para evaluar y comparar grandes conjuntos de datos en su propio campo.
Para 1982, los investigadores compartían información a través de tarjetas perforadas. La cantidad de datos creció exponencialmente a fines de la década de 1980, lo que requirió nuevos métodos computacionales para interpretar rápidamente la información relevante.
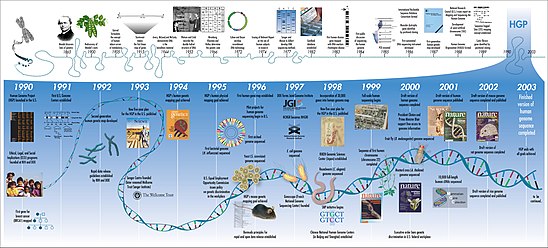
Quizás el ejemplo más conocido de biología computacional, el Proyecto Genoma Humano, comenzó oficialmente en 1990. Para 2003, el proyecto había mapeado alrededor del 85 % del genoma humano, satisfaciendo sus objetivos iniciales. Sin embargo, el trabajo continuó y, para 2021, el nivel "genoma completo" se alcanzó con solo el 0,3% de las bases restantes cubiertas por problemas potenciales. El cromosoma Y faltante se agregó en enero de 2022.
Desde finales de la década de 1990, la biología computacional se ha convertido en una parte importante de la biología, lo que lleva a numerosos subcampos. En la actualidad, la Sociedad Internacional de Biología Computacional reconoce 21 "Comunidades de interés especial" diferentes, cada una de las cuales representa una porción del campo más grande. Además de ayudar a secuenciar el genoma humano, la biología computacional ha ayudado a crear modelos precisos del cerebro humano, mapear la estructura 3D de los genomas y modelar sistemas biológicos.
Aplicaciones
Anatomía
La anatomía computacional es el estudio de la forma anatómica y la forma en la anatomía visible o burda 50− − 100μ μ {displaystyle 50-100mu} escala de morfología. Implica el desarrollo de métodos matemáticos computacionales y analíticos de datos para modelar y simular estructuras biológicas. Se centra en las estructuras anatómicas que se están viendo, en lugar de los dispositivos de imagen médica. Debido a la disponibilidad de mediciones 3D densas a través de tecnologías como la resonancia magnética, la anatomía computacional ha surgido como un subcampo de imágenes médicas y bioingeniería para extraer sistemas de coordenadas anatómicas en la escala de morfemo en 3D.
La formulación original de la anatomía computacional es como un modelo generativo de forma y forma a partir de ejemplares actuados a través de transformaciones. El grupo diffeomorfismo se utiliza para estudiar diferentes sistemas de coordenadas a través de transformaciones de coordenadas generadas a través de las velocidades lagrangianas y eulerias del flujo de una configuración anatómica en R3{displaystyle {Mathbb {R} } {3} a otro. Se relaciona con estadísticas de forma y morfometría, con la distinción de que los diffeomorfismos se utilizan para mapear sistemas de coordenadas, cuyo estudio se conoce como diffeomorfometría.
Datos y modelado
La biología matemática es el uso de modelos matemáticos de organismos vivos para examinar los sistemas que gobiernan la estructura, el desarrollo y el comportamiento en los sistemas biológicos. Esto implica un enfoque más teórico de los problemas, en lugar de su contraparte más empírica de la biología experimental. La biología matemática se basa en las matemáticas discretas, la topología (también útil para el modelado computacional), las estadísticas bayesianas, el álgebra lineal y el álgebra booleana.
Estos enfoques matemáticos han permitido la creación de bases de datos y otros métodos para almacenar, recuperar y analizar datos biológicos, un campo conocido como bioinformática. Por lo general, este proceso involucra la genética y el análisis de genes.
La recopilación y el análisis de grandes conjuntos de datos han dejado espacio para campos de investigación en crecimiento, como la extracción de datos y el biomodelado computacional, que se refiere a la creación de modelos informáticos y simulaciones visuales de sistemas biológicos. Esto permite a los investigadores predecir cómo reaccionarán dichos sistemas en diferentes entornos, lo que es útil para determinar si un sistema puede "mantener su estado y funciones frente a perturbaciones externas e internas". Si bien las técnicas actuales se enfocan en pequeños sistemas biológicos, los investigadores están trabajando en enfoques que permitirán analizar y modelar redes más grandes. La mayoría de los investigadores creen que esto será esencial en el desarrollo de enfoques médicos modernos para crear nuevos medicamentos y terapias génicas. Un enfoque de modelado útil es usar redes de Petri a través de herramientas como esyN.
De manera similar, hasta décadas recientes, la ecología teórica se ha ocupado en gran medida de modelos analíticos que se separaron de los modelos estadísticos utilizados por los ecólogos empíricos. Sin embargo, los métodos computacionales han ayudado a desarrollar la teoría ecológica a través de la simulación de sistemas ecológicos, además de aumentar la aplicación de métodos de estadísticas computacionales en análisis ecológicos.
Biología de Sistemas
La biología de sistemas consiste en computar las interacciones entre varios sistemas biológicos que van desde el nivel celular hasta poblaciones enteras con el objetivo de descubrir propiedades emergentes. Este proceso suele implicar la creación de redes de señalización celular y vías metabólicas. La biología de sistemas a menudo utiliza técnicas computacionales de modelado biológico y teoría de grafos para estudiar estas complejas interacciones a nivel celular.
Biología evolutiva
La biología computacional ha ayudado a la biología evolutiva al:
- Utilizando datos de ADN para reconstruir el árbol de la vida con la filogenética computacional
- Fijar modelos de genética poblacional (ya sea tiempo de avance o tiempo atrasado) a datos de ADN para hacer inferencias sobre historia demográfica o selectiva
- Elaboración de modelos genéticos demográficos de sistemas evolutivos desde los primeros principios para predecir lo que es probable que evolucione
Genómica
La genómica computacional es el estudio de los genomas de las células y los organismos. El Proyecto Genoma Humano es un ejemplo de genómica computacional. Este proyecto busca secuenciar todo el genoma humano en un conjunto de datos. Una vez implementado por completo, esto podría permitir a los médicos analizar el genoma de un paciente individual. Esto abre la posibilidad de una medicina personalizada, prescribiendo tratamientos basados en los patrones genéticos preexistentes de un individuo. Los investigadores buscan secuenciar los genomas de animales, plantas, bacterias y todos los demás tipos de vida.
Una de las principales formas en que se comparan los genomas es por homología de secuencia. La homología es el estudio de estructuras biológicas y secuencias de nucleótidos en diferentes organismos que provienen de un ancestro común. La investigación sugiere que entre el 80 y el 90 % de los genes en los genomas procarióticos recién secuenciados se pueden identificar de esta manera.
La alineación de secuencias es otro proceso para comparar y detectar similitudes entre secuencias biológicas o genes. La alineación de secuencias es útil en varias aplicaciones bioinformáticas, como calcular la subsecuencia común más larga de dos genes o comparar variantes de ciertas enfermedades.
Un proyecto intacto en genómica computacional es el análisis de regiones intergénicas, que comprenden aproximadamente el 97 % del genoma humano. Los investigadores están trabajando para comprender las funciones de las regiones no codificantes del genoma humano a través del desarrollo de métodos computacionales y estadísticos ya través de grandes proyectos de consorcios como ENCODE y Roadmap Epigenomics Project.
La comprensión de cómo los genes individuales contribuyen a la biología de un organismo a nivel molecular, celular y de organismo se conoce como ontología génica. La misión del Gene Ontology Consortium es desarrollar un modelo computacional completo y actualizado de sistemas biológicos, desde el nivel molecular hasta vías más grandes, sistemas celulares y a nivel de organismo. El recurso Gene Ontology proporciona una representación computacional del conocimiento científico actual sobre las funciones de los genes (o, más correctamente, las proteínas y las moléculas de ARN no codificantes producidas por los genes) de muchos organismos diferentes, desde humanos hasta bacterias.
La genómica 3D es una subsección de la biología computacional que se centra en la organización y la interacción de los genes dentro de una célula eucariota. Un método utilizado para recopilar datos genómicos en 3D es a través del mapeo de la arquitectura del genoma (GAM). GAM mide las distancias en 3D de la cromatina y el ADN en el genoma mediante la combinación de criosección, el proceso de cortar una tira del núcleo para examinar el ADN, con microdisección láser. Un perfil nuclear es simplemente esta tira o rebanada que se toma del núcleo. Cada perfil nuclear contiene ventanas genómicas, que son ciertas secuencias de nucleótidos, la unidad base del ADN. GAM captura una red genómica de contactos de cromatina complejos y potenciadores múltiples en toda una célula.
Neurociencia
La neurociencia computacional es el estudio de la función cerebral en términos de las propiedades de procesamiento de información del sistema nervioso. Un subconjunto de la neurociencia, busca modelar el cerebro para examinar aspectos específicos del sistema neurológico. Los modelos del cerebro incluyen:
- Realistic Brain Modelos: Estos modelos buscan representar todos los aspectos del cerebro, incluyendo tanto detalle a nivel celular como sea posible. Los modelos realistas proporcionan la mayor información sobre el cerebro, pero también tienen el mayor margen de error. Más variables en un modelo cerebral crean la posibilidad de que ocurra más error. Estos modelos no representan partes de la estructura celular que los científicos no conocen. Los modelos de cerebro realistas son los más pesados computacionalmente y los más caros para implementar.
- Simplifying Brain Models: Estos modelos buscan limitar el alcance de un modelo para evaluar una propiedad física específica del sistema neurológico. Esto permite resolver los problemas computacionales intensivos y reduce la cantidad de error potencial de un modelo cerebral realista.
Es el trabajo de los neurocientíficos computacionales mejorar los algoritmos y las estructuras de datos que se utilizan actualmente para aumentar la velocidad de dichos cálculos.
La neuropsiquiatría computacional es un campo emergente que utiliza modelos matemáticos y asistidos por computadora de los mecanismos cerebrales involucrados en los trastornos mentales. Varias iniciativas han demostrado que el modelado computacional es una contribución importante para comprender los circuitos neuronales que podrían generar funciones y disfunciones mentales.
Farmacología
La farmacología computacional es "el estudio de los efectos de los datos genómicos para encontrar vínculos entre genotipos y enfermedades específicos y luego examinar los datos de fármacos". La industria farmacéutica requiere un cambio en los métodos para analizar los datos de medicamentos. Los farmacólogos pudieron usar Microsoft Excel para comparar datos químicos y genómicos relacionados con la eficacia de los medicamentos. Sin embargo, la industria ha llegado a lo que se conoce como la barricada de Excel. Esto surge del número limitado de celdas accesibles en una hoja de cálculo. Este desarrollo condujo a la necesidad de la farmacología computacional. Los científicos e investigadores desarrollan métodos computacionales para analizar estos conjuntos de datos masivos. Esto permite una comparación eficiente entre los puntos de datos notables y permite desarrollar fármacos más precisos.
Los analistas proyectan que si los principales medicamentos fallan debido a las patentes, será necesaria la biología computacional para reemplazar los medicamentos actuales en el mercado. Se alienta a los estudiantes de doctorado en biología computacional a seguir carreras en la industria en lugar de tomar puestos posdoctorales. Este es un resultado directo de que las principales compañías farmacéuticas necesitan analistas más calificados de los grandes conjuntos de datos necesarios para producir nuevos medicamentos.
Del mismo modo, la oncología computacional tiene como objetivo determinar las futuras mutaciones en el cáncer a través de enfoques algorítmicos. La investigación en este campo ha llevado al uso de mediciones de alto rendimiento que millones de puntos de datos utilizan robótica y otros dispositivos de detección. Estos datos se recopilan a partir de ADN, ARN y otras estructuras biológicas. Las áreas de enfoque incluyen la determinación de las características de los tumores, el análisis de moléculas que son deterministas en la causa del cáncer y la comprensión de cómo el genoma humano se relaciona con la causa de los tumores y el cáncer.
Técnicas
Los biólogos informáticos utilizan una amplia variedad de software y algoritmos para llevar a cabo sus investigaciones.
Aprendizaje no supervisado
El aprendizaje no supervisado es un tipo de algoritmo que encuentra patrones en datos no etiquetados. Un ejemplo es el agrupamiento de k-medias, que tiene como objetivo dividir n puntos de datos en k grupos, en los que cada punto de datos pertenece al grupo con la media más cercana. Otra versión es el algoritmo k-medoids, que, al seleccionar un centro de conglomerado o un centroide de conglomerado, elegirá uno de sus puntos de datos en el conjunto, y no solo un promedio del conglomerado.
El algoritmo sigue estos pasos:
- Selección aleatoria k distintos puntos de datos. Estos son los grupos iniciales.
- Medir la distancia entre cada punto y cada uno de los clusters 'k'. (Esta es la distancia de los puntos desde cada punto k).
- Asignar cada punto al clúster más cercano.
- Encuentra el centro de cada cluster (medoid).
- Repita hasta que los racimos ya no cambien.
- Evaluar la calidad de la agrupación agregando la variación dentro de cada grupo.
- Repita los procesos con diferentes valores de k.
- Elija el mejor valor para 'k' al encontrar el "codo" en la parcela de que valor k tiene la varianza más baja.
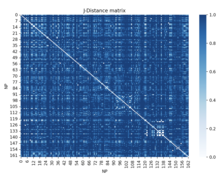
Un ejemplo de esto en biología se usa en el mapeo 3D de un genoma. La información de la región HIST1 del cromosoma 13 de un ratón se obtiene de Gene Expression Omnibus. Esta información contiene datos sobre qué perfiles nucleares aparecen en ciertas regiones genómicas. Con esta información, la distancia de Jaccard se puede utilizar para encontrar una distancia normalizada entre todos los loci.
Análisis de gráficos
El análisis de gráficos, o análisis de redes, es el estudio de gráficos que representan conexiones entre diferentes objetos. Los gráficos pueden representar todo tipo de redes en biología, como redes de interacción proteína-proteína, redes reguladoras, redes metabólicas y bioquímicas y mucho más. Hay muchas maneras de analizar estas redes. Uno de los cuales está mirando la centralidad en los gráficos. Encontrar centralidad en gráficos asigna clasificaciones de nodos a su popularidad o centralidad en el gráfico. Esto puede ser útil para encontrar qué nodos son los más importantes. Esto puede ser muy útil en biología de muchas maneras. Por ejemplo, si tuviéramos datos sobre la actividad de los genes en un período de tiempo determinado, podemos usar la centralidad de grado para ver qué genes están más activos en toda la red, o qué genes interactúan más con otros en toda la red. Esto puede ayudarnos a comprender qué roles juegan ciertos genes en la red.
Hay muchas formas de calcular la centralidad en gráficos, todas las cuales pueden brindar diferentes tipos de información sobre la centralidad. Encontrar centralidades en biología se puede aplicar en muchas circunstancias diferentes, algunas de las cuales son la regulación de genes, la interacción de proteínas y las redes metabólicas.
Aprendizaje supervisado
El aprendizaje supervisado es un tipo de algoritmo que aprende de datos etiquetados y aprende a asignar etiquetas a datos futuros que no están etiquetados. En biología, el aprendizaje supervisado puede ser útil cuando tenemos datos que sabemos cómo categorizar y nos gustaría categorizar más datos en esas categorías.
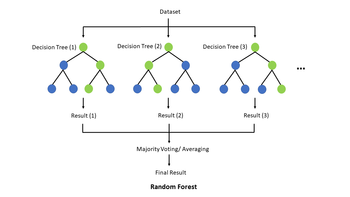
Un algoritmo de aprendizaje supervisado común es el bosque aleatorio, que utiliza numerosos árboles de decisión para entrenar un modelo para clasificar un conjunto de datos. Formando la base del bosque aleatorio, un árbol de decisión es una estructura que tiene como objetivo clasificar, o etiquetar, algún conjunto de datos utilizando ciertas características conocidas de esos datos. Un ejemplo biológico práctico de esto sería tomar los datos genéticos de un individuo y predecir si ese individuo está predispuesto o no a desarrollar una determinada enfermedad o cáncer. En cada nodo interno, el algoritmo verifica el conjunto de datos en busca de exactamente una característica, un gen específico en el ejemplo anterior, y luego se bifurca hacia la izquierda o hacia la derecha según el resultado. Luego, en cada nodo hoja, el árbol de decisión asigna una etiqueta de clase al conjunto de datos. Entonces, en la práctica, el algoritmo recorre un camino específico de raíz a hoja basado en el conjunto de datos de entrada a través del árbol de decisión, lo que da como resultado la clasificación de ese conjunto de datos. Por lo general, los árboles de decisión tienen variables de destino que toman valores discretos, como sí/no, en cuyo caso se denomina árbol de clasificación, pero si la variable de destino es continua, se denomina árbol de regresión. Para construir un árbol de decisión, primero debe entrenarse utilizando un conjunto de entrenamiento para identificar qué características son los mejores predictores de la variable objetivo.
Software de código abierto
El software de código abierto proporciona una plataforma para la biología computacional donde todos pueden acceder y beneficiarse del software desarrollado en la investigación. PLOS cita cuatro razones principales para el uso de software de código abierto:
- Reproducibilidad: Esto permite a los investigadores utilizar los métodos exactos utilizados para calcular las relaciones entre datos biológicos.
- Desarrollo más rápido: los desarrolladores e investigadores no tienen que reinventar el código existente para tareas menores. En cambio, pueden utilizar programas preexistentes para ahorrar tiempo en el desarrollo y ejecución de proyectos más grandes.
- Mayor calidad: Tener entrada de múltiples investigadores que estudian el mismo tema proporciona una capa de seguridad de que los errores no estarán en el código.
- Disponibilidad a largo plazo: Los programas de código abierto no están vinculados a ninguna empresa o patente. Esto les permite publicarse en varias páginas web y asegurar que estén disponibles en el futuro.
Investigación
Hay varias conferencias importantes que se ocupan de la biología computacional. Algunos ejemplos notables son Sistemas Inteligentes para Biología Molecular, Conferencia Europea sobre Biología Computacional e Investigación en Biología Molecular Computacional.
También hay numerosas revistas dedicadas a la biología computacional. Algunos ejemplos notables incluyen Journal of Computational Biology y PLOS Computational Biology, una revista de acceso abierto revisada por pares que tiene muchos proyectos de investigación notables en el campo de la biología computacional. Proporcionan revisiones de software, tutoriales para software de código abierto y muestran información sobre las próximas conferencias de biología computacional.
Campos relacionados
La biología computacional, la bioinformática y la biología matemática son enfoques interdisciplinarios de las ciencias de la vida que se basan en disciplinas cuantitativas como las matemáticas y las ciencias de la información. El NIH describe la biología computacional/matemática como el uso de enfoques computacionales/matemáticos para abordar cuestiones teóricas y experimentales en biología y, por el contrario, la bioinformática como la aplicación de la ciencia de la información para comprender datos complejos de las ciencias de la vida.
Específicamente, el NIH define
Biología computacional: Desarrollo y aplicación de métodos analíticos y teóricos de datos, modelos matemáticos y técnicas de simulación computacional para el estudio de sistemas biológicos, conductuales y sociales.
Bioinformática: Investigación, desarrollo o aplicación de herramientas y enfoques computacionales para ampliar el uso de datos biológicos, médicos, conductuales o de salud, incluyendo aquellos para adquirir, almacenar, organizar, archivar, analizar o visualizar dichos datos.
Si bien cada campo es distinto, puede haber una superposición significativa en su interfaz, tanto que, para muchos, la bioinformática y la biología computacional son términos que se usan indistintamente.
Los términos biología computacional y computación evolutiva tienen un nombre similar, pero no deben confundirse. A diferencia de la biología computacional, la computación evolutiva no se ocupa de modelar y analizar datos biológicos. En su lugar, crea algoritmos basados en las ideas de evolución entre especies. A veces denominados algoritmos genéticos, la investigación de este campo se puede aplicar a la biología computacional. Si bien la computación evolutiva no es inherentemente una parte de la biología computacional, la biología evolutiva computacional es un subcampo de la misma.
Contenido relacionado
Gen egoísta
Efecto del segundo sistema
Señalización de canal separado