ARN
Algunas moléculas de ARN desempeñan un papel activo dentro de las células al catalizar reacciones biológicas, controlar la expresión génica o detectar y comunicar respuestas a señales celulares. Uno de estos procesos activos es la síntesis de proteínas, una función universal en la que las moléculas de ARN dirigen la síntesis de proteínas en los ribosomas. Este proceso utiliza moléculas de ARN de transferencia (ARNt) para entregar aminoácidos al ribosoma, donde el ARN ribosómico (ARNr) luego une los aminoácidos para formar proteínas codificadas.
Comparación con ADN
Al igual que el ADN, la mayoría de los ARN biológicamente activos, incluidos ARNm, ARNt, ARNr, ARNsn y otros ARN no codificantes, contienen secuencias autocomplementarias que permiten que partes del ARN se plieguen y se emparejen consigo mismo para formar hélices dobles. El análisis de estos ARN ha revelado que están muy estructurados. A diferencia del ADN, sus estructuras no consisten en largas hélices dobles, sino en colecciones de hélices cortas empaquetadas en estructuras similares a las proteínas.
De esta manera, los ARN pueden lograr una catálisis química (como las enzimas). Por ejemplo, la determinación de la estructura del ribosoma, un complejo ARN-proteína que cataliza la formación de enlaces peptídicos, reveló que su sitio activo está compuesto enteramente por ARN.
Estructura
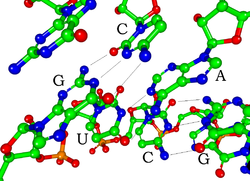
Cada nucleótido en el ARN contiene un azúcar ribosa, con carbonos numerados 1' hasta 5'. Se adjunta una base al 1' posición, en general, adenina (A), citosina (C), guanina (G), o uracilo (U). La adenina y la guanina son purinas, la citosina y el uracilo son pirimidinas. Un grupo fosfato está unido al 3' posición de una ribosa y el 5' posición del siguiente. Los grupos fosfato tienen una carga negativa cada uno, lo que hace que el ARN sea una molécula cargada (polianión). Las bases forman enlaces de hidrógeno entre citosina y guanina, entre adenina y uracilo y entre guanina y uracilo. Sin embargo, son posibles otras interacciones, como un grupo de bases de adenina que se unen entre sí en un bulto, o el tetraloop GNRA que tiene un par de bases guanina-adenina.
Un componente estructural importante del ARN que lo distingue del ADN es la presencia de un grupo hidroxilo en el extremo 2' posición del azúcar ribosa. La presencia de este grupo funcional hace que la hélice adopte principalmente la geometría de forma A, aunque en contextos de dinucleótidos monocatenarios, el ARN rara vez puede adoptar también la forma B que se observa más comúnmente en el ADN. La geometría en forma de A da como resultado un surco mayor estrecho y muy profundo y un surco menor poco profundo y ancho. Una segunda consecuencia de la presencia del grupo 2'-hidroxilo es que en regiones conformacionalmente flexibles de una molécula de ARN (es decir, no involucradas en la formación de una doble hélice), puede atacar químicamente el enlace fosfodiéster adyacente para escindir el columna vertebral.
El ARN se transcribe con solo cuatro bases (adenina, citosina, guanina y uracilo), pero estas bases y los azúcares adjuntos pueden modificarse de muchas maneras a medida que maduran los ARN. La pseudouridina (Ψ), en la que el enlace entre el uracilo y la ribosa cambia de un enlace C-N a un enlace C-C, y la ribotimidina (T) se encuentran en varios lugares (los más notables son el bucle TΨC del ARNt).). Otra base modificada notable es la hipoxantina, una base de adenina desaminada cuyo nucleósido se denomina inosina (I). La inosina juega un papel clave en la hipótesis del tambaleo del código genético.
Hay más de 100 nucleósidos modificados naturales. La mayor diversidad estructural de modificaciones se puede encontrar en el ARNt, mientras que la pseudouridina y los nucleósidos con 2'-O-metilribosa a menudo presentes en el ARNr son los más comunes. Las funciones específicas de muchas de estas modificaciones en el ARN no se conocen por completo. Sin embargo, cabe destacar que, en el ARN ribosómico, muchas de las modificaciones postranscripcionales ocurren en regiones altamente funcionales, como el centro de la peptidil transferasa y la interfaz de la subunidad, lo que implica que son importantes para el funcionamiento normal.
La forma funcional de las moléculas de ARN monocatenario, al igual que las proteínas, requiere con frecuencia una estructura terciaria específica. El andamiaje para esta estructura lo proporcionan elementos estructurales secundarios que son enlaces de hidrógeno dentro de la molécula. Esto conduce a varios "dominios" reconocibles. de estructura secundaria como bucles de horquilla, protuberancias y bucles internos. Para crear, es decir, diseñar, un ARN para cualquier estructura secundaria dada, dos o tres bases no serían suficientes, pero cuatro bases son suficientes. Esta es probablemente la razón por la cual la naturaleza ha "elegido" un alfabeto de cuatro bases: menos de cuatro no permite crear todas las estructuras, mientras que más de cuatro bases no son necesarias. Dado que el ARN está cargado, se necesitan iones metálicos como Mg2+ para estabilizar muchas estructuras secundarias y terciarias.
El enantiómero natural del ARN es el D-ARN compuesto de D-ribonucleótidos. Todos los centros de quiralidad están ubicados en la D-ribosa. Mediante el uso de L-ribosa o más bien L-ribonucleótidos, se puede sintetizar L-RNA. L-RNA es mucho más estable frente a la degradación por RNase.
Al igual que otros biopolímeros estructurados, como las proteínas, se puede definir la topología de una molécula de ARN plegada. Esto a menudo se hace en función de la disposición de los contactos dentro de la cadena dentro de un ARN plegado, denominado topología de circuito.
Síntesis
La síntesis de ARN suele estar catalizada por una enzima, la ARN polimerasa, que utiliza el ADN como plantilla, un proceso conocido como transcripción. El inicio de la transcripción comienza con la unión de la enzima a una secuencia promotora en el ADN (generalmente se encuentra "aguas arriba" de un gen). La doble hélice del ADN se desenrolla por la actividad helicasa de la enzima. Luego, la enzima avanza a lo largo de la hebra molde en la dirección 3' a 5', sintetizando una molécula de ARN complementaria con elongación en la dirección 5' a 3'. La secuencia de ADN también dicta dónde ocurrirá la terminación de la síntesis de ARN.
Los ARN de transcripción primaria a menudo son modificados por enzimas después de la transcripción. Por ejemplo, una cola de poli(A) y una de 5' cap se agregan al pre-mRNA eucariótico y los intrones son eliminados por el spliceosoma.
También hay varias polimerasas de ARN dependientes de ARN que utilizan el ARN como molde para la síntesis de una nueva cadena de ARN. Por ejemplo, varios virus de ARN (como el poliovirus) utilizan este tipo de enzima para replicar su material genético. Además, la polimerasa de ARN dependiente de ARN es parte de la ruta de interferencia de ARN en muchos organismos.
Tipos de ARN
Resumen
El ARN mensajero (ARNm) es el ARN que transporta información desde el ADN hasta el ribosoma, los sitios de síntesis de proteínas (traducción) en la célula. El ARNm es una copia del ADN. La secuencia de codificación del ARNm determina la secuencia de aminoácidos en la proteína que se produce. Sin embargo, muchos ARN no codifican proteínas (alrededor del 97% de la producción transcripcional no codifica proteínas en eucariotas).
Estos denominados ARN no codificantes ("ncRNA") pueden ser codificados por sus propios genes (genes de ARN), pero también pueden derivar de intrones de ARNm. Los ejemplos más destacados de ARN no codificantes son el ARN de transferencia (ARNt) y el ARN ribosómico (ARNr), los cuales están involucrados en el proceso de traducción. También hay ARN no codificantes involucrados en la regulación de genes, el procesamiento de ARN y otras funciones. Ciertos ARN pueden catalizar reacciones químicas como cortar y ligar otras moléculas de ARN y la catálisis de la formación de enlaces peptídicos en el ribosoma; estos se conocen como ribozimas.
En longitud
Según la longitud de la cadena de ARN, el ARN incluye ARN pequeño y ARN largo. Por lo general, los ARN pequeños tienen una longitud inferior a 200 nt y los ARN largos tienen una longitud superior a 200 nt. Los ARN largos, también llamados ARN grandes, incluyen principalmente ARN no codificante largo (lncRNA) y ARNm. Los ARN pequeños incluyen principalmente ARN ribosómico 5.8S (ARNr), ARNr 5S, ARN de transferencia (ARNt), microARN (miARN), ARN de interferencia pequeño (ARNip), ARN nucleolar pequeño (ARNsno), ARN que interactúa con Piwi (ARNpi), ARNt- ARN pequeño derivado (tsRNA) y ARN derivado de rDNA pequeño (srRNA). Hay ciertas excepciones como en el caso del rRNA 5S de los miembros del género Halococcus (Archaea), que tienen una inserción, aumentando así su tamaño.
En traducción
El ARN mensajero (ARNm) lleva información sobre una secuencia de proteínas a los ribosomas, las fábricas de síntesis de proteínas en la célula. Está codificado de manera que cada tres nucleótidos (un codón) corresponde a un aminoácido. En las células eucariotas, una vez que el ARNm precursor (pre-ARNm) se ha transcrito a partir del ADN, se procesa para convertirlo en ARNm maduro. Esto elimina sus intrones: secciones no codificantes del pre-ARNm. Luego, el ARNm se exporta desde el núcleo al citoplasma, donde se une a los ribosomas y se traduce a su forma de proteína correspondiente con la ayuda del ARNt. En las células procarióticas, que no tienen compartimentos de núcleo y citoplasma, el ARNm puede unirse a los ribosomas mientras se transcribe a partir del ADN. Después de una cierta cantidad de tiempo, el mensaje se degrada en los nucleótidos que lo componen con la ayuda de las ribonucleasas.
El ARN de transferencia (ARNt) es una pequeña cadena de ARN de aproximadamente 80 nucleótidos que transfiere un aminoácido específico a una cadena polipeptídica en crecimiento en el sitio ribosomal de síntesis de proteínas durante la traducción. Tiene sitios para la unión de aminoácidos y una región de anticodón para el reconocimiento de codones que se une a una secuencia específica en la cadena de ARN mensajero a través de enlaces de hidrógeno.
El ARN ribosomal (ARNr) es el componente catalítico de los ribosomas. El rRNA es el componente del ribosoma que alberga la traducción. Los ribosomas eucarióticos contienen cuatro moléculas de ARNr diferentes: ARNr 18S, 5.8S, 28S y 5S. Tres de las moléculas de ARNr se sintetizan en el nucléolo y una se sintetiza en otro lugar. En el citoplasma, el ARN ribosomal y la proteína se combinan para formar una nucleoproteína llamada ribosoma. El ribosoma se une al ARNm y lleva a cabo la síntesis de proteínas. Varios ribosomas pueden unirse a un solo ARNm en cualquier momento. Casi todo el ARN que se encuentra en una célula eucariota típica es ARNr.
El ARN mensajero de transferencia (tmRNA) se encuentra en muchas bacterias y plástidos. Marca proteínas codificadas por ARNm que carecen de codones de parada para la degradación y evita que el ribosoma se detenga.
ARN regulador
Los primeros reguladores conocidos de la expresión génica fueron proteínas conocidas como represores y activadores, reguladores con sitios de unión cortos específicos dentro de las regiones potenciadoras cerca de los genes que se van a regular. Estudios posteriores han demostrado que los ARN también regulan los genes. Hay varios tipos de procesos dependientes de ARN en eucariotas que regulan la expresión de genes en varios puntos, como ARNi que reprime genes postranscripcionalmente, ARN largos no codificantes que cierran bloques de cromatina epigenéticamente y ARN potenciadores que inducen una mayor expresión génica. También se ha demostrado que las bacterias y las arqueas utilizan sistemas de ARN reguladores, como los ARN pequeños bacterianos y CRISPR. Fire y Mello recibieron el Premio Nobel de Fisiología o Medicina de 2006 por descubrir los microARN (miARN), moléculas cortas específicas de ARN que pueden formar pares de bases con los ARNm.
Interferencia de ARN por miRNAs
Los niveles de expresión postranscripcional de muchos genes pueden controlarse mediante la interferencia de ARN, en la que los miARN, moléculas cortas específicas de ARN, se emparejan con regiones de ARNm y las dirigen para su degradación. Este proceso basado en antisentido implica pasos que primero procesan el ARN para que pueda emparejarse con una región de sus ARNm objetivo. Una vez que ocurre el apareamiento de bases, otras proteínas dirigen el ARNm para que sea destruido por las nucleasas.
ARN largos no codificantes
Los siguientes en vincularse con la regulación fueron Xist y otros ARN largos no codificantes asociados con la inactivación del cromosoma X. Jeannie T. Lee y otros demostraron que sus roles, al principio misteriosos, eran el silenciamiento de bloques de cromatina mediante el reclutamiento del complejo Polycomb para que el ARN mensajero no pudiera transcribirse a partir de ellos. Se han encontrado lncRNA adicionales, actualmente definidos como ARN de más de 200 pares de bases que no parecen tener potencial de codificación, asociados con la regulación de la pluripotencia de las células madre y la división celular.
ARN potenciadores
El tercer grupo principal de ARN reguladores se denomina ARN potenciadores. No está claro en la actualidad si son una categoría única de ARN de varias longitudes o si constituyen un subconjunto distinto de lncRNA. En cualquier caso, se transcriben a partir de potenciadores, que son sitios reguladores conocidos en el ADN cerca de los genes que regulan. Regulan al alza la transcripción de los genes bajo el control del potenciador desde el que se transcriben.
ARN regulador en procariotas
Al principio, se pensó que el ARN regulador era un fenómeno eucariótico, una parte de la explicación de por qué se observó mucha más transcripción en organismos superiores de lo que se había predicho. Pero tan pronto como los investigadores comenzaron a buscar posibles reguladores de ARN en bacterias, también aparecieron allí, denominados ARN pequeño (ARNs). Actualmente, la naturaleza ubicua de los sistemas de regulación de genes por ARN se ha discutido como apoyo a la teoría del mundo del ARN. Los ARN pequeños bacterianos generalmente actúan a través del emparejamiento antisentido con el ARNm para regular a la baja su traducción, ya sea afectando la estabilidad o afectando la capacidad de unión a cis. También se han descubierto ribointerruptores. Son secuencias de ARN reguladoras que actúan en cis y actúan alostéricamente. Cambian de forma cuando se unen a metabolitos para que ganen o pierdan la capacidad de unirse a la cromatina para regular la expresión de genes.
Las arqueas también tienen sistemas de ARN regulador. El sistema CRISPR, utilizado recientemente para editar ADN in situ, actúa a través de ARN reguladores en arqueas y bacterias para brindar protección contra los invasores de virus.
En procesamiento de ARN
Muchos ARN participan en la modificación de otros ARN. Los intrones se separan del pre-ARNm mediante empalmesomas, que contienen varios ARN nucleares pequeños (ARNsn), o los intrones pueden ser ribozimas que se empalman por sí mismos. El ARN también se puede alterar modificando sus nucleótidos a nucleótidos distintos de A, C, G y U. En eucariotas, las modificaciones de los nucleótidos de ARN están dirigidas en general por pequeños ARN nucleolares (snoRNA; 60–300 nt), que se encuentran en el nucléolo y los cuerpos cajal. Los snoRNA se asocian con enzimas y las guían a un punto en un ARN mediante el emparejamiento de bases con ese ARN. Estas enzimas luego realizan la modificación de nucleótidos. Los ARNr y los ARNt se modifican ampliamente, pero los ARNsn y los ARNm también pueden ser el objetivo de la modificación de bases. El ARN también se puede metilar.
Genomas de ARN
Al igual que el ADN, el ARN puede transportar información genética. Los virus de ARN tienen genomas compuestos de ARN que codifica varias proteínas. Algunas de esas proteínas replican el genoma viral, mientras que otras proteínas protegen el genoma a medida que la partícula del virus se traslada a una nueva célula huésped. Los viroides son otro grupo de patógenos, pero consisten solo en ARN, no codifican ninguna proteína y son replicados por la polimerasa de una célula vegetal huésped.
En transcripción inversa
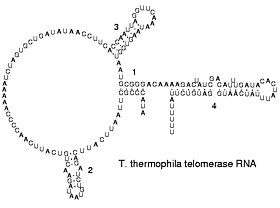
Los virus de transcripción inversa replican sus genomas mediante la transcripción inversa de copias de ADN de su ARN; estas copias de ADN luego se transcriben a nuevo ARN. Los retrotransposones también se propagan copiando el ADN y el ARN entre sí, y la telomerasa contiene un ARN que se usa como plantilla para construir los extremos de los cromosomas eucariotas.
ARN de doble cadena
El ARN de doble cadena (dsRNA) es ARN con dos cadenas complementarias, similar al ADN que se encuentra en todas las células, pero con la sustitución de la timina por uracilo y la adición de un átomo de oxígeno. dsRNA forma el material genético de algunos virus (virus de ARN de doble cadena). El ARN de doble cadena, como el ARN viral o el siARN, puede desencadenar la interferencia del ARN en los eucariotas, así como la respuesta del interferón en los vertebrados. En eucariotas, el ARN de doble cadena (dsRNA) juega un papel en la activación del sistema inmunológico innato contra las infecciones virales.
ARN circular
A fines de la década de 1970, se demostró que existe una forma circular de ARN de una sola hebra covalentemente cerrada expresada en todo el reino animal y vegetal (ver circRNA). Se cree que los circRNA surgen a través de un "empalme posterior" reacción donde el spliceosoma se une a un 3' aceptador a un 5' sitio de empalme donante. Hasta el momento, se desconoce en gran medida la función de los circRNA, aunque en algunos ejemplos se ha demostrado una actividad esponjada de microRNA.
Descubrimientos clave en biología del ARN
La investigación sobre el ARN ha dado lugar a muchos descubrimientos biológicos importantes y a numerosos premios Nobel. Los ácidos nucleicos fueron descubiertos en 1868 por Friedrich Miescher, quien llamó al material 'nucleína' ya que se encontró en el núcleo. Más tarde se descubrió que las células procariotas, que no tienen núcleo, también contienen ácidos nucleicos. El papel del ARN en la síntesis de proteínas ya se sospechaba en 1939. Severo Ochoa ganó el Premio Nobel de Medicina de 1959 (compartido con Arthur Kornberg) después de descubrir una enzima que puede sintetizar ARN en el laboratorio. Sin embargo, más tarde se demostró que la enzima descubierta por Ochoa (polinucleótido fosforilasa) era responsable de la degradación del ARN, no de la síntesis del ARN. En 1956, Alex Rich y David Davies hibridaron dos hebras separadas de ARN para formar el primer cristal de ARN cuya estructura pudo determinarse mediante cristalografía de rayos X.
La secuencia de los 77 nucleótidos de un ARNt de levadura fue encontrada por Robert W. Holley en 1965, lo que le valió a Holley el Premio Nobel de Medicina de 1968 (compartido con Har Gobind Khorana y Marshall Nirenberg).
A principios de la década de 1970, se descubrieron los retrovirus y la transcriptasa inversa, lo que demostró por primera vez que las enzimas podían copiar el ARN en ADN (la ruta opuesta a la habitual para la transmisión de información genética). Por este trabajo, David Baltimore, Renato Dulbecco y Howard Temin recibieron el Premio Nobel en 1975. En 1976, Walter Fiers y su equipo determinaron la primera secuencia completa de nucleótidos de un genoma de virus ARN, la del bacteriófago MS2.
En 1977, se descubrieron intrones y empalmes de ARN tanto en virus de mamíferos como en genes celulares, lo que resultó en un Nobel de 1993 para Philip Sharp y Richard Roberts. Las moléculas de ARN catalítico (ribozimas) se descubrieron a principios de la década de 1980, lo que condujo a un premio Nobel de 1989 a Thomas Cech y Sidney Altman. En 1990, se descubrió en Petunia que los genes introducidos pueden silenciar genes similares propios de la planta, que ahora se sabe que son el resultado de la interferencia del ARN.
Al mismo tiempo, se descubrió que los ARN de 22 nt de largo, ahora llamados microARN, tenían un papel en el desarrollo de C. elegante. Los estudios sobre la interferencia del ARN obtuvieron un Premio Nobel para Andrew Fire y Craig Mello en 2006, y otro Nobel para los estudios sobre la transcripción del ARN para Roger Kornberg en el mismo año. El descubrimiento de los ARN reguladores de genes ha dado lugar a intentos de desarrollar fármacos hechos de ARN, como siRNA, para silenciar genes. Además de los premios Nobel otorgados por la investigación sobre el ARN en 2009, se otorgó por la elucidación de la estructura atómica del ribosoma a Venki Ramakrishnan, Thomas A. Steitz y Ada Yonath.
Relevancia para la química prebiótica y la abiogénesis
En 1968, Carl Woese planteó la hipótesis de que el ARN podría ser un catalizador y sugirió que las primeras formas de vida (moléculas autorreplicantes) podrían haber dependido del ARN tanto para transportar información genética como para catalizar reacciones bioquímicas: un mundo de ARN. En mayo de 2022, los científicos informaron que descubrieron formas de ARN espontáneamente en vidrio de lava basáltica prebiótica que se presume estuvo abundantemente disponible en la Tierra primitiva.
En marzo de 2015, se informó que en el laboratorio se formaron nucleótidos complejos de ADN y ARN, incluidos uracilo, citosina y timina, en condiciones del espacio exterior, utilizando químicos iniciadores, como la pirimidina, un compuesto orgánico que se encuentra comúnmente en los meteoritos. La pirimidina, como los hidrocarburos aromáticos policíclicos (HAP), es uno de los compuestos más ricos en carbono que se encuentran en el Universo y puede haberse formado en gigantes rojas o en nubes de gas y polvo interestelar. En julio de 2022, los astrónomos informaron del descubrimiento de cantidades masivas de moléculas prebióticas, incluidos posibles precursores de ARN, en el Centro Galáctico de la Vía Láctea.
Contenido relacionado
Nepeta
Autosoma
Aoto