Analógico de estado de transición
Los análogos del estado de transición (transition state analogs) son compuestos químicos con una estructura química que se asemeja al estado de transición de una molécula de sustrato en una reacción química catalizada por enzimas. Las enzimas interactúan con un sustrato mediante tensión o distorsiones, moviendo el sustrato hacia el estado de transición. Los análogos del estado de transición se pueden utilizar como inhibidores en reacciones catalizadas por enzimas al bloquear el sitio activo de la enzima. La teoría sugiere que los inhibidores de enzimas que se asemejan a la estructura del estado de transición se unirían más fuertemente a la enzima que el sustrato real. Algunos ejemplos de medicamentos que son inhibidores análogos del estado de transición incluyen medicamentos contra la gripe como el inhibidor de la neuraminidasa oseltamivir y los inhibidores de la proteasa del VIH saquinavir en el tratamiento del SIDA.
Transition state analogue

El estado de transición de una estructura se puede describir mejor en relación con la mecánica estadística, donde las energías de ruptura y formación de enlaces tienen la misma probabilidad de pasar del estado de transición hacia atrás a los reactivos o hacia adelante a los productos. En las reacciones catalizadas por enzimas, la energía de activación general de la reacción se reduce cuando una enzima estabiliza un intermediario de estado de transición de alta energía. Los análogos del estado de transición imitan este intermediario de alta energía, pero no experimentan una reacción química catalizada y, por lo tanto, pueden unirse mucho más fuertemente a una enzima que los análogos de sustrato o producto simples.
Designing transition state analogue
Para diseñar un análogo del estado de transición, el paso fundamental es la determinación de la estructura del estado de transición del sustrato en la enzima específica de interés con un método experimental, por ejemplo, el efecto isotópico cinético. Además, la estructura del estado de transición también se puede predecir con métodos computacionales como complemento al KIE. Explicaremos estos dos métodos brevemente.
Efecto de isótopo cinético
El efecto isotópico cinético (EIC) es una medida de la velocidad de reacción de los reactivos marcados con isótopos frente al sustrato natural más común. Los valores del efecto isotópico cinético son una relación del número de recambio e incluyen todos los pasos de la reacción. Los valores isotópicos cinéticos intrínsecos se derivan de la diferencia en el entorno vibracional de enlace de un átomo en los reactivos en estado fundamental con respecto al entorno del estado de transición del átomo. A través del efecto isotópico cinético se puede obtener mucha información sobre cómo se ve el estado de transición de una reacción catalizada por enzimas y guiar el desarrollo de análogos del estado de transición.
Simulación computacional
Los métodos computacionales se han considerado una herramienta útil para dilucidar el mecanismo de acción de las enzimas. La mecánica molecular por sí sola no puede predecir la transferencia de electrones, que es fundamental en las reacciones orgánicas, pero la simulación de dinámica molecular proporciona información suficiente considerando la flexibilidad de las proteínas durante la reacción catalítica. El método complementario sería la combinación de métodos de simulación de mecánica molecular y mecánica cuántica (QM/MM). Con este enfoque, solo los átomos responsables de la reacción enzimática en la región catalítica se tratarían con mecánica cuántica y el resto de los átomos se tratarían con mecánica molecular.
Ejemplos de diseño analógico estatal de transición
Después de determinar las estructuras de estado de transición utilizando KIE o simulaciones computacionales, el inhibidor puede diseñarse de acuerdo con las estructuras de estado de transición determinadas o los intermediarios. Los siguientes tres ejemplos ilustran cómo los inhibidores imitan la estructura de estado de transición al cambiar los grupos funcionales que corresponden a la geometría y la distribución electrostática de las estructuras de estado de transición.
Inhibidor de la metiltioadenosina nucleosidase
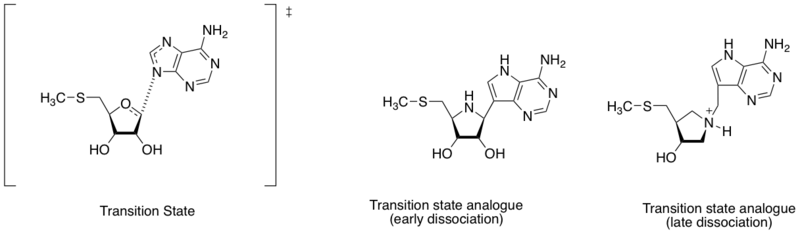
La metiltioadenosina nucleosidasa es una enzima que cataliza la reacción de desadenilación hidrolítica de la 5'-metiltioadenosina y la S-adenosilhomocisteína. También se considera un objetivo importante para el descubrimiento de fármacos antibacterianos porque es importante en el sistema metabólico de las bacterias y solo las bacterias la producen. Dada la diferente distancia entre el átomo de nitrógeno de la adenina y el carbono anomérico de la ribosa (ver en el diagrama de esta sección), la estructura del estado de transición se puede definir por la etapa de disociación temprana o tardía. Basándose en el hallazgo de diferentes estructuras de estado de transición, Schramm y colaboradores diseñaron dos análogos de estado de transición que imitaban el estado de transición disociativo temprano y tardío. El análogo de estado de transición temprano y tardío mostró una afinidad de unión (Kd) de 360 y 140 pM, respectivamente.
Inhibidor de la termosin

La termolisina es una enzima producida por Bacillus thermoproteolyticus que cataliza la hidrólisis de péptidos que contienen aminoácidos hidrofóbicos. Por lo tanto, también es un objetivo para los agentes antibacterianos. El mecanismo de reacción enzimática comienza a partir de la pequeña molécula de péptido y reemplaza la molécula de agua que se une al zinc hacia Glu143 de la termolisina. La molécula de agua es entonces activada tanto por el ion de zinc como por el residuo de Glu143 y ataca al carbono carbonílico para formar un estado de transición tetraédrico (ver figura). Holden y sus colaboradores luego imitaron ese estado de transición tetraédrico para diseñar una serie de análogos peptídicos de fosfonamidato. Entre los análogos sintetizados, R = L-Leu posee la actividad inhibidora más potente (Ki = 9,1 nM).
Inhibidor de la arginasa
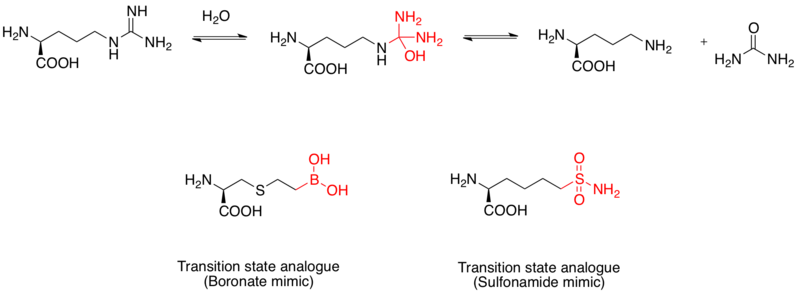
La arginasa es una metaloproteína binuclear de manganeso que cataliza la hidrólisis de la L-arginina a L-ornitina y urea. También se la considera un objetivo farmacológico para el tratamiento del asma. El mecanismo de hidrólisis de la L-arginina se lleva a cabo mediante un ataque nucleofílico al grupo guanidino por el agua, formando un intermediario tetraédrico. Los estudios han demostrado que una fracción de ácido borónico adopta una configuración tetraédrica y actúa como inhibidor. Además, el grupo funcional sulfonamida también puede imitar la estructura del estado de transición. La evidencia de que el ácido borónico imita a los inhibidores análogos del estado de transición de la arginasa I humana se dilucidó mediante estructuras cristalinas de rayos X.
Véase también
- Enzyme
- Analógico estructural, compuestos con estructura química similar
- Inhibidor enzima
- Substrate analog
- Inhibidor de suicidios
- Substrate
Referencias
- ^ Silverman RB (2004). La química orgánica del diseño de drogas y la acción de drogas. San Diego, CA: Elsevier Academic Press. ISBN 0-12-643732-7.
- ^ Davis JP, Cain GA, Pitts WJ, Magolda RL, Copeland RA (enero de 1996). "El metabolito inmunosupresivo de la leflunomida es un potente inhibidor de la deshidroorota humana". Bioquímica. 35 (4): 1270–1273. doi:10.1021/bi952168g. PMID 8573583.
- ^ a b Schramm VL (2011). "Estados de transición enzimática, análogos del estado de transición, dinámicas, termodinámicas y vidas". Examen anual de la bioquímica. 80 (1): 703–732. doi:10.1146/annurev-biochem-061809-100742. PMC 5502542. PMID 21675920.
- ^ Kollman P, Kuhn B, Peräkylä M (2002). "Estudios computacionales de reacciones amenazadas con la enzima: ¿Dónde estamos en mecanismos de procesamiento y en la comprensión de la naturaleza de la catalisis enzimática?". J. Phys. Chem. B. 106 (7): 1537–1542. doi:10.1021/jp012017p.
- ^ Hou G, Cui Q (enero de 2012). "El análisis QM/MM sugiere que la fosfatasa Alkaline (AP) y la pirofosfatasa nucleótido/fosfodiesterasa ajusta ligeramente el estado de transición para la hidrolisis de fósforo diester relativa a la solución: implicación para la promiscuidad catalítica en la superfamilia AP". Journal of the American Chemical Society. 134 (1): 229–246. doi:10.1021/ja205226d. PMC 3257412. PMID 22097879.
- ^ Saen-Oon S, Quaytman-Machleder S, Schramm VL, Schwartz SD (octubre de 2008). "Detalle atómico de la transformación química en el estado de transición de una reacción enzimática". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América. 105 (43): 16543–16548. Código:2008PNAS..10516543S. doi:10.1073/pnas.080808413105. PMC 2575456. PMID 18946041.
- ^ Singh V, Lee JE, Núñez S, Howell PL, Schramm VL (septiembre de 2005). "La estructura estatal de transición de 5'-metiltioadenosine/S-adenosylhomocysteine nucleosidase de Escherichia coli y su similitud con los análogos estatales de transición". Bioquímica. 44 (35): 11647–11659. doi:10.1021/bi050863a. PMID 16128565.
- ^ Gutiérrez JA, Luo M, Singh V, Li L, Brown RL, Norris GE, et al. (noviembre de 2007). "Inhibidores picomolar como sondas de estado de transición de núcleos nucleosidases de 5'-metiltioadenosina". ACS Chemical Biology. 2 (11): 725–734. doi:10.1021/cb700166z. PMID 18030989.
- ^ Endo S (1962). "Estudios sobre proteasa producidos por bacterias termofílicas". J. Ferment. Technol. 40: 346-353.
- ^ Holden HM, Tronrud DE, Monzingo AF, Weaver LH, Matthews BW (diciembre de 1987). "Los inhibidores de unión lenta y rápida de la termosin muestran diferentes modos de unión: análisis cristalinográfico de los análogos de transición-estatal ampliados de fosfonamidate". Bioquímica. 26 (26): 8542–8553. doi:10.1021/bi00400a008. PMID 3442675.
- ^ Maarsingh H, Zaagsma J, Meurs H (octubre de 2009). "Arginasa: una enzima clave en la fisiopatología del asma alérgico que abre nuevas perspectivas terapéuticas". British Journal of Pharmacology. 158 (3): 652–664. doi:10.1111/j.1476-5381.2009.00374.x. PMC 2765587. PMID 19703164.
- ^ Cama E, Shin H, Christianson DW (octubre de 2003). "Design of amino acid sulfonamides as transition-state analoguehibiors of arginase". Journal of the American Chemical Society. 125 (43): 13052–13057. doi:10.1021/ja036365b. PMID 14570477.
- ^ Shishova EY, Di Costanzo L, Emig FA, Ash DE, Christianson DW (enero de 2009). "Probing the specificity determinants of amino acid recognition by arginase". Bioquímica. 48 (1): 121–131. doi:10.1021/bi801911v. PMC 2665027. PMID 19093830.