Análisis del flujo metabólico
El análisis de flujo metabólico (MFA) es una técnica fluxómica experimental que se utiliza para examinar las tasas de producción y consumo de metabolitos en un sistema biológico. A nivel intracelular, permite la cuantificación de los flujos metabólicos, lo que permite dilucidar el metabolismo central de la célula. Varios métodos de MFA, incluidos el análisis de flujo metabólico isotópicamente estacionario, el análisis de flujo metabólico isotópicamente no estacionario y el análisis de flujo metabólico basado en la termodinámica, se pueden combinar con modelos estequiométricos de metabolismo y métodos de espectrometría de masas con resolución de masa isotópica para dilucidar la transferencia de fracciones que contienen trazadores isotópicos de un metabolito a otro y derivar información sobre la red metabólica. El análisis de flujo metabólico (MFA) tiene muchas aplicaciones, como determinar los límites de la capacidad de un sistema biológico para producir un bioquímico como el etanol, predecir la respuesta a la inactivación de genes y guiar la identificación de enzimas que constituyen cuellos de botella en las redes metabólicas para los esfuerzos de ingeniería metabólica.
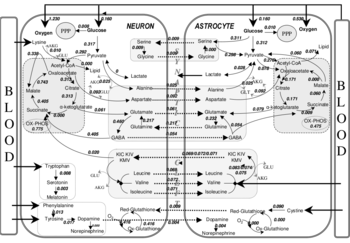
El análisis de flujo metabólico puede utilizar trazadores de isótopos marcados con 13C para experimentos de marcado isotópico. Las técnicas de resonancia magnética nuclear (RMN) y la espectrometría de masas pueden utilizarse para medir los patrones de marcado de metabolitos y proporcionar información para la determinación de los flujos de las vías metabólicas. Debido a que el análisis de flujo metabólico generalmente requiere un cálculo riguroso del flujo de redes metabólicas complejas, se han desarrollado herramientas de software disponibles públicamente para automatizar el análisis de flujo metabólico y reducir su carga computacional.
Método experimental
Aunque el uso de un equilibrio estequiométrico y de restricciones de los metabolitos que componen la red metabólica puede dilucidar los flujos, este enfoque tiene limitaciones, incluida la dificultad de estimular los flujos a través de vías paralelas, cíclicas y reversibles. Además, existe un conocimiento limitado sobre cómo los metabolitos se interconvierten en una red metabólica sin el uso de trazadores isotópicos. Por lo tanto, el uso de isótopos se ha convertido en la técnica dominante para la MFA.
Isotope etiquetando experimentos
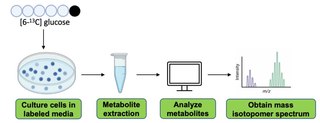
Metodologías
Isotopically stationary
Un método predominante para el análisis de flujo metabólico es el MFA isotópicamente estacionario. Esta técnica para la cuantificación de flujo es aplicable en estado estacionario metabólico e isotópico, dos condiciones que suponen que las concentraciones de metabolitos y las distribuciones de isotopómeros no cambian con el tiempo, respectivamente. El conocimiento de la matriz estequiométrica (S) que comprende el consumo y la producción de metabolitos dentro de las reacciones bioquímicas es necesario para equilibrar los flujos (v) en torno al modelo de red metabólica asumido. Suponiendo un estado estacionario metabólico, los flujos metabólicos pueden cuantificarse resolviendo la inversa de la siguiente ecuación de álgebra lineal simple:
Para reducir el espacio de solución posible para las distribuciones de flujo, el MFA isotópicamente estacionario requiere restricciones estequiométricas adicionales, como tasas de crecimiento, secreción y absorción de sustrato y tasas de acumulación de producto, así como límites superiores e inferiores para los flujos. Aunque el MFA isotópicamente estacionario permite una deducción precisa de los flujos metabólicos mediante modelos matemáticos, el análisis se limita a cultivos por lotes durante la fase exponencial. Además, después de la adición de un sustrato marcado, puede resultar difícil determinar el momento en el que se puede suponer con precisión el estado estacionario metabólico e isotópico.
Isotópicamente no estacionario
Cuando el etiquetado isotópico es transitorio y aún no se ha equilibrado, el método MFA isotópicamente no estacionario (INST-MFA) resulta ventajoso para deducir flujos, en particular para sistemas con dinámicas de etiquetado lentas. De manera similar al método MFA isotópicamente estacionario, este método requiere balances de masa e isotopómeros para caracterizar la estequiometría y las transiciones atómicas de la red metabólica. Sin embargo, a diferencia de los métodos MFA tradicionales, el método INST-MFA requiere la aplicación de ecuaciones diferenciales ordinarias para examinar cómo cambian con el tiempo los patrones de etiquetado isotópico de los metabolitos; dicho examen se puede lograr midiendo los patrones de etiquetado isotópico cambiantes en diferentes puntos de tiempo para ingresarlos en el método INST-MFA. Por lo tanto, el método INST-MFA es un método poderoso para dilucidar los flujos de sistemas con cuellos de botella en las vías y revelar fenotipos metabólicos de organismos autótrofos. Si bien las demandas computacionales intensivas de INST-MFA anteriormente obstaculizaban su uso generalizado, las herramientas de software desarrolladas recientemente han simplificado INST-MFA para reducir el tiempo computacional y la demanda.
Termodinámica basada en
El análisis de flujo metabólico basado en la termodinámica (TMFA) es un tipo especializado de análisis de flujo metabólico que utiliza restricciones termodinámicas lineales además de restricciones de balance de masa para generar flujos termodinámicamente factibles y perfiles de actividad de metabolitos. El TMFA solo tiene en cuenta las vías y los flujos que son factibles mediante el uso del cambio de energía libre de Gibbs de las reacciones y actividades de los metabolitos que forman parte del modelo. Al calcular las energías libres de Gibbs de las reacciones metabólicas y, en consecuencia, su favorabilidad termodinámica, el TMFA facilita la identificación de reacciones limitantes de cuellos de botella de las vías que pueden ser candidatos ideales para la regulación de las vías.
Software
Se necesitan algoritmos de simulación para modelar el sistema biológico y calcular los flujos de todas las vías en una red compleja. Existen varios programas informáticos para satisfacer la necesidad de herramientas eficientes y precisas para la cuantificación de flujos. En general, los pasos para aplicar software de modelado a la MFA incluyen la reconstrucción metabólica para compilar todas las reacciones enzimáticas y metabolitos deseados, proporcionar información experimental como el patrón de etiquetado del sustrato, definir restricciones como ecuaciones de crecimiento y minimizar el error entre los resultados experimentales y simulados para obtener flujos finales. Algunos ejemplos de software de MFA incluyen 13CFLUX2 y OpenFLUX, que evalúan experimentos de etiquetado de 13C para el cálculo de flujos en condiciones metabólicas e isotópicamente estacionarias. El creciente interés en el desarrollo de herramientas informáticas para el cálculo de INST-MFA también ha llevado al desarrollo de aplicaciones de software como INCA, que fue el primer software capaz de realizar INST-MFA y simular experimentos de etiquetado de isótopos transitorios.
Aplicaciones
Producción de biocombustibles
El análisis de flujo metabólico se ha utilizado para orientar los esfuerzos de ampliación de la fermentación de biocombustibles. Al medir directamente las tasas de reacción enzimática, el análisis de flujo metabólico puede capturar la dinámica del comportamiento de las células y los fenotipos metabólicos en biorreactores durante fermentaciones a gran escala. Por ejemplo, los modelos de análisis de flujo metabólico se utilizaron para optimizar la conversión de xilosa en etanol en levaduras fermentadoras de xilosa mediante el uso de distribuciones de flujo calculadas para determinar las capacidades teóricas máximas de la levadura seleccionada para la producción de etanol.
Ingeniería metabólica
La identificación de las enzimas que actúan como cuello de botella determina las reacciones que limitan la velocidad y que limitan la productividad de una vía biosintética. Además, el análisis de la modulación de la enzima puede ayudar a predecir fenotipos inesperados de cepas modificadas genéticamente al generar una comprensión fundamental de cómo se conectan los flujos en las células modificadas. Por ejemplo, al calcular las energías libres de Gibbs de las reacciones en el metabolismo de Escherichia coli, el análisis de la modulación de la enzima facilitó la identificación de una reacción termodinámica que actúa como cuello de botella en un modelo a escala del genoma de Escherichia coli.
Véase también
- Isotopic labeling
- Análisis del equilibrio de fluidos
- Fluxomics
- Modelado de red metabólico
- Metabolomics
- Ingeniería metabólica
Referencias
- ^ a b Wiechert W (julio de 2001). "análisis de flujo metabólico 13C". Ingeniería metabólica. 3 3): 195–206. doi:10.1006/mben.2001.0187. PMID 11461141.
- ^ Papoutsakis ET, Meyer CL (enero de 1985). "Ecuaciones y cálculos de rendimientos de productos y vías preferidas para fermentaciones butanedioles y mixtas de ácido". Biotecnología y Bioingeniería. 27 1): 50-66. doi:10.1002/bit.260270108. PMID 18553576. S2CID 41031084.
- ^ Burgard AP, Maranas CD (septiembre 2001). "Probing the performance limits of the Escherichia coli metabolic network subject to gene additions or deletions". Biotecnología y Bioingeniería. 74 5): 364–375. doi:10.1002/bit.1127. PMID 11427938.
- ^ Henry CS, Broadbelt LJ, Hatzimanikatis V (marzo de 2007). "Análisis de flujo metabólico basado en termodinámica". Biophysical Journal. 92 5): 1792 –1805. Bibcode:2007BpJ....92.1792H. doi:10.1529/biophysj.106.093138. PMC 1796839. PMID 17172310.
- ^ Antoniewicz MR (enero 2021). "Una guía para el análisis de flujo metabólico en ingeniería metabólica: Métodos, herramientas y aplicaciones". Ingeniería metabólica. Herramientas y Estrategias de Ingeniería Metabólica. 63: 2-12. doi:10.1016/j.ymben.2020.11.002. ISSN 1096-7176. S2CID 226276199.
- ^ Zamboni N, Fendt SM, Rühl M, Sauer U (2009-06-21). "(13)C-based metabolic flux analysis". Protocolos de la naturaleza. 4 (6): 878 –892. doi:10.1038/nprot.2009.58. PMID 19478804. S2CID 6731942.
- ^ Zamboni N (febrero de 2011). "13C análisis de flujo metabólico en sistemas complejos". Opinión actual en Biotecnología. 22 1): 103 –108. doi:10.1016/j.copbio.2010.08.009. PMID 20833526.
- ^ a b Wang, Y; Wondisford, FE; Song, C; Zhang, T; Su, X (2020-11-06). "Metabolic Flux Analysis—Linking Isotope Labeling and Metabolic Fluxes". Metabolitos. 10 11): 447. doi:10.3390/metabo10110447. ISSN 2218-1989. PMC 7694648. PMID 33172051.
- ^ Crown, SB; Antoniewicz, MR (marzo 2013). "Experimentos paralizantes y análisis de flujo metabólico: Metodologías pasadas, presentes y futuras". Ingeniería metabólica. 16: 21 –32. doi:10.1016/j.ymben.2012.11.010. PMC 6474253. PMID 23246523.
- ^ Heuillet M, Bellvert F, Cahoreau E, Letisse F, Millard P, Portais JC (Febrero 2018). "Metodología para la validación de Analisis Isotópica por Espectrometría Masiva en Experimentos de Etiqueta Estable-Isótopo". Química Analítica. 90 3): 1852 –1860. doi:10.1021/acs.analchem.7b03886. PMID 29260858.
- ^ a b Feng X, Zhuang WQ, Colletti P, Tang YJ (2012). "Metabolic Pathway Determination and Flux Analysis in Nonmodel Microorganisms Through 13C-Isotope Labeling". En Navid A (ed.). Microbial Systems Biology. Métodos en Biología Molecular. Vol. 881. Totowa, NJ: Humana Prensa. pp. 309 –330. doi:10.1007/978-1-61779-827-6_11. ISBN 978-1-61779-827-6. PMID 22639218.
- ^ Sellick CA, Hansen R, Stephens GM, Goodacre R, Dickson AJ (Julio 2011). "Metabolite extraction from suspension-cultured mammalian cells for global metabolite profiling". Protocolos de la naturaleza. 6 (8): 1241–1249. doi:10.1038/nprot.2011.366. PMID 21799492. S2CID 10329440.
- ^ a b c Dai Z, Locasale JW (septiembre de 2017). "El metabolismo independiente con análisis de flujo: De la teoría a la aplicación". Ingeniería metabólica. 43 (Pt B): 94 –102. doi:10.1016/j.ymben.2016.09.005. PMC 5362364. PMID 27667771.
- ^ Antoniewicz MR (marzo 2015). "Métodos y avances en análisis de flujo metabólico: una mini-revisión". Journal of Industrial Microbiology " Biotechnology. 42 3): 317–325. doi:10.1007/s10295-015-1585-x. PMID 25613286. S2CID 2233610.
- ^ Hollinshead W, He L, Tang YJ (2019). "13C-Fingerprinting and Metabolic Flux Analysis of Bacterial Metabolisms". En Santos CN, Ajikumar PK (eds.). Ingeniería metabólica microbiana. Métodos en Biología Molecular. Vol. 1927. Nueva York, NY: Springer New York. pp. 215–230. doi:10.1007/978-1-4939-9142-6_15. ISBN 978-1-4939-9141-9. OSTI 1529155. PMID 30788795. S2CID 73491481.
- ^ a b Cheah YE, Young JD (diciembre de 2018). "Análisis de flujo metabólico no estacionario (INST-MFA): poner la teoría en práctica". Opinión actual en Biotecnología. Biotecnología analítica. 54: 80–87. doi:10.1016/j.copbio.2018.02.013. OSTI 1611014. PMID 29522915. S2CID 3793073.
- ^ Jazmin LJ, O'Grady JP, Ma F, Allen DK, Morgan JA, Young JD (2014). "MFA Isotópicamente No Estacionaria (INST-MFA) del Metabolismo Autotrófico". En Dieuaide-Noubhani M, Alonso AP (eds.). Análisis de flujo metabólico. Métodos en biología molecular. Vol. 1090. Totowa, NJ: Humana Prensa. pp. 181 –210. doi:10.1007/978-1-62703-688-7_12. ISBN 978-1-62703-688-7. PMID 24222417.
- ^ a b Henry CS, Broadbelt LJ, Hatzimanikatis V (marzo de 2007). "Análisis de flujo metabólico basado en termodinámica". Biophysical Journal. 92 5): 1792 –1805. Bibcode:2007BpJ....92.1792H. doi:10.1529/biophysj.106.093138. PMC 1796839. PMID 17172310.
- ^ Dandekar T, Fieselmann A, Majeed S, Ahmed Z (enero de 2014). "Software applications towards quantitative metabolic flux analysis and modeling". Reuniones informativas en Bioinformática. 15 1): 91 –107. doi:10.1093/bib/bbs065. PMID 23142828.
- ^ Weitzel M, Nöh K, Dalman T, Niedenführ S, Stute B, Wiechert W (enero de 2013). "13CFLUX2 - suite de software de alto rendimiento para (13)C-metabólico análisis de flujo". Bioinformática. 29 1): 143 –145. doi:10.1093/bioinformática/bts646. PMC 3530911. PMID 23110970.
- ^ Quek LE, Wittmann C, Nielsen LK, Krömer JO (mayo de 2009). "OpenFLUX: software eficiente de modelado para el análisis de flujo metabólico basado en 13C". Factores de células microbianas. 8 1): 25. doi:10.1186/1475-2859-8-25. PMC 2689189. PMID 19409084.
- ^ Young JD (mayo de 2014). "INCA: una plataforma computacional para el análisis de flujo metabólico isotópico no estacionario". Bioinformática. 30 (9): 1333–1335. doi:10.1093/bioinformática/btu015. PMC 3998137. PMID 24413674.
- ^ a b Hollinshead W, He L, Tang YJ (2014). "Producción de Biofuel: una odisea de ingeniería metabólica a escala de fermentación". Fronteras en Microbiología. 5: 344. doi:10.3389/fmicb.2014.00344. PMC 4088188. PMID 25071754.
- ^ Bideaux C, Montheard J, Cameleyre X, Molina-Jouve C, Alfenore S (febrero 2016). "Metabolic flux analysis model for optimizing xylose conversion into ethanol by the natural C5-fermenting leeast Candida shehatae". Microbiología aplicada y biotecnología. 100 3): 1489–1499. doi:10.1007/s00253-015-7085-0. PMID 26536879. S2CID 18196579.
- ^ Antoniewicz MR (enero 2021). "Una guía para el análisis de flujo metabólico en ingeniería metabólica: Métodos, herramientas y aplicaciones". Ingeniería metabólica. Herramientas y Estrategias de Ingeniería Metabólica. 63: 2-12. doi:10.1016/j.ymben.2020.11.002. ISSN 1096-7176. S2CID 226276199.
